Seaborn:統計可視化利器
作為基于 Matplotlib 的高級繪圖庫,有一下功能:

一元特征數據
直方圖
import matplotlib.pyplot as plt
import pandas as pd
import seaborn as sns # import os
# # 如果文件夾不存在,創建文件夾
# if not os.path.isdir("Figures"):
# os.makedirs("Figures")# 導入鳶尾花數據
iris_sns = sns.load_dataset("iris") iris_sns# 繪制花萼長度樣本數據直方圖
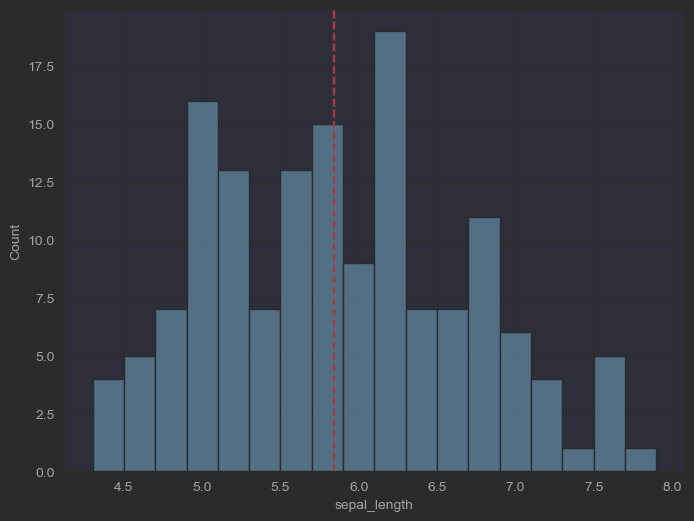
fig, ax = plt.subplots(figsize = (8, 6))sns.histplot(data=iris_sns, x="sepal_length", binwidth=0.2, ax = ax) # 縱軸三個選擇:頻率、概率、概率密度ax.axvline(x = iris_sns.sepal_length.mean(), color = 'r', ls = '--') # 增加均值位置豎直參考線# 參考https://seaborn.pydata.org/tutorial/distributions.html
效果:

代碼演示如下圖所示:
# 繪制花萼長度樣本數據直方圖, 考慮鳶尾花分類
fig, ax = plt.subplots(figsize = (8,6))
sns.histplot(data = iris_sns, x="sepal_length",
hue = 'species', binwidth=0.2, ax = ax,
element="step", stat = 'density')
# 縱軸為概率密度
如果要分組的話使用如下代碼:
fig, ax = plt.subplots(figsize=(8, 6))
sns.histplot(data=iris_sns, # 數據源(DataFrame)x="sepal_length", # 指定x軸為花萼長度hue='species', # 按鳶尾花種類分組著色binwidth=0.2, # 直方圖條柱寬度為0.2ax=ax, # 指定繪圖坐標軸element="step", # 直方圖樣式為階梯線stat='density' # 縱軸顯示密度而非計數
)
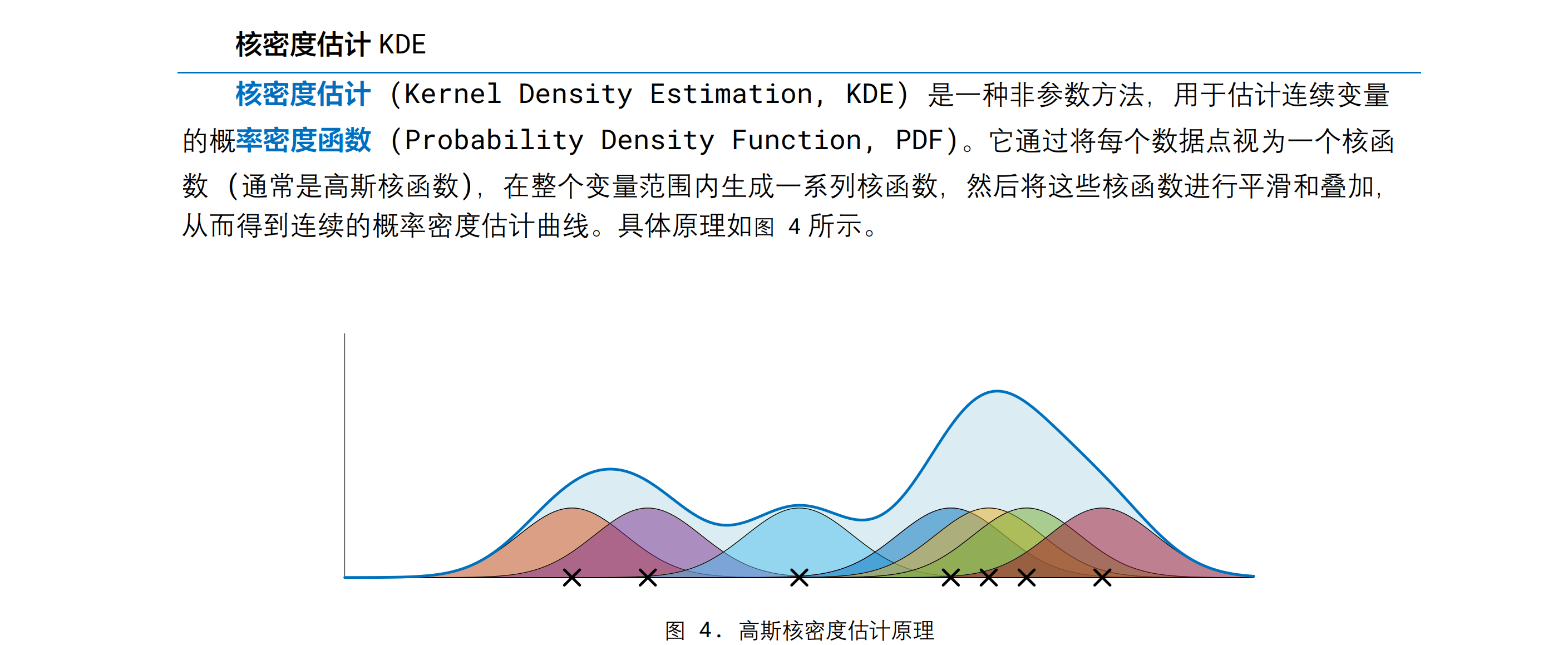
核密度估計KDE
將每個點變成一個高斯核函數(就是高斯分布的那個函數形式),然后再疊加
# 繪制花萼長度樣本數據,高斯核密度估計
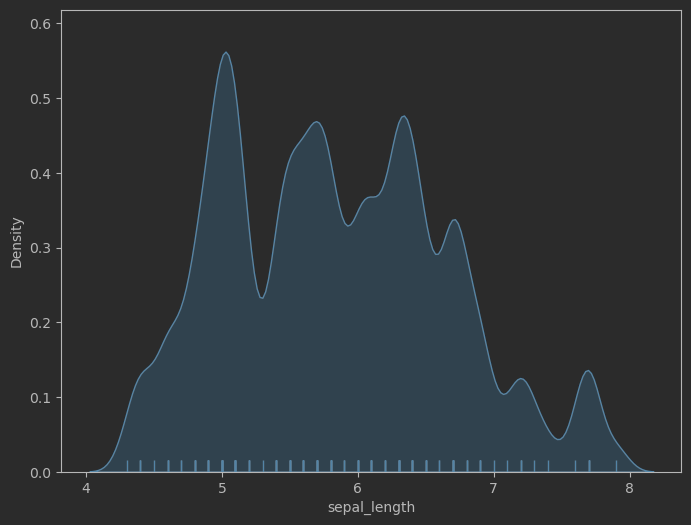
fig, ax = plt.subplots(figsize = (8,6))sns.kdeplot(data=iris_sns, x="sepal_length", # 生成核密度曲線bw_adjust=0.3, fill = True) # 調整曲率,填充區域
sns.rugplot(data=iris_sns, x="sepal_length") # 生產毛毯圖(小刺)
效果:
# 繪制花萼長度樣本數據,高斯核密度估計,考慮鳶尾花類別
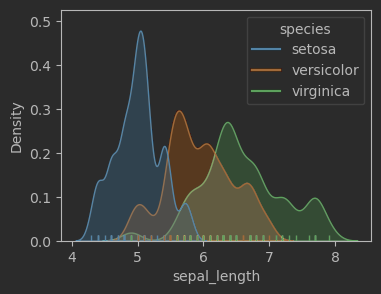
fig, ax = plt.subplots(figsize = (8,6))sns.kdeplot(data=iris_sns, x="sepal_length", hue = 'species', # 各自的分布bw_adjust=0.5, fill = True)
sns.rugplot(data=iris_sns, x="sepal_length", hue = 'species')# fig.savefig('Figures\一元,kdeplot + rugplot + hue.svg', format='svg')
# 繪制花萼長度樣本數據,高斯核密度估計,考慮鳶尾花類別,堆疊
fig, ax = plt.subplots(figsize = (8,6))sns.kdeplot(data=iris_sns, x="sepal_length", hue="species", multiple="stack", # 設置疊加屬性bw_adjust=0.5)效果:
# 繪制后驗概率 (成員值)fig, ax = plt.subplots(figsize = (8,6))
sns.kdeplot(data=iris_sns, x="sepal_length", hue="species", bw_adjust=0.5,multiple = 'fill') # 設置疊加效果
效果:
分散點圖/蜂群圖

較小的數據使用:seaborn.stripplot() 蜂群圖
較大的數據使用:seaborn.swarmplot() 分散點圖
# 繪制鳶尾花花萼長度分散點圖
fig, ax = plt.subplots(figsize = (8,6))
sns.stripplot(data=iris_sns, x="sepal_length", y="species", hue="petal_length", palette="RdYlBu_r", ax = ax)
效果:
# 繪制花萼長度樣本數據, 蜂群圖
fig, ax = plt.subplots(figsize = (8,4))
sns.swarmplot(data=iris_sns, x="sepal_length", ax = ax)
# 繪制花萼長度樣本數據, 蜂群圖, 考慮分類
fig, ax = plt.subplots(figsize = (8,4))
sns.swarmplot(data=iris_sns, x="sepal_length", y = 'species',
hue = 'species', ax = ax)
箱型圖
包含元素:

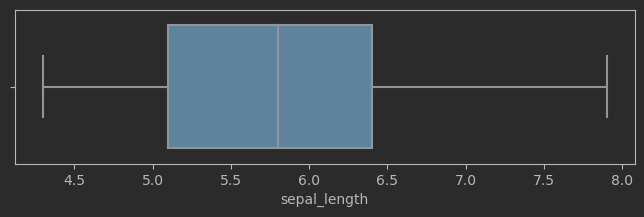
# 繪制鳶尾花花萼長度箱型圖
fig, ax = plt.subplots(figsize = (8,2))
sns.boxplot(data=iris_sns, x="sepal_length", ax = ax)
效果:
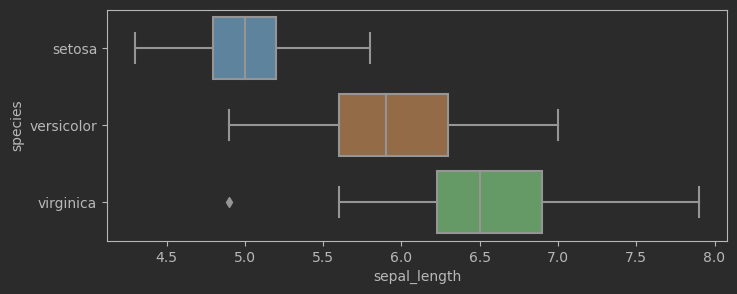
# 繪制鳶尾花花萼長度箱型圖,考慮鳶尾花分類
fig, ax = plt.subplots(figsize = (8,3))
sns.boxplot(data=iris_sns, x="sepal_length", y = 'species', ax = ax)
效果:
小提琴圖
可以看成用核密度曲線優化的箱線圖
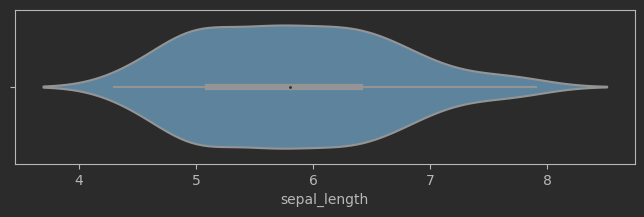
# 繪制花萼長度樣本數據,小提琴圖
fig, ax = plt.subplots(figsize = (8,2))
sns.violinplot(data=iris_sns, x="sepal_length", ax = ax)
效果:
# 繪制花萼長度樣本數據,小提琴圖,考慮分類
fig, ax = plt.subplots(figsize = (8,4))
sns.violinplot(data=iris_sns, x="sepal_length", y="species", ax = ax)
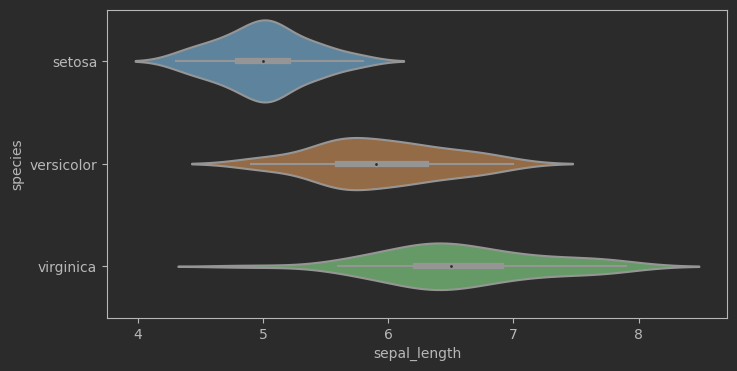
sns.violinplot(data=iris_sns, x="sepal_length", y="species", inner = 'stick')
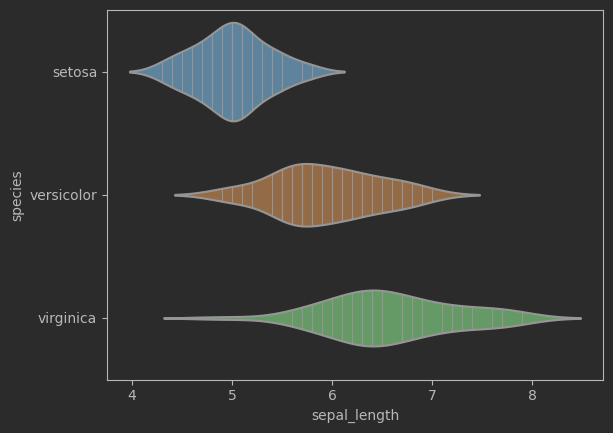
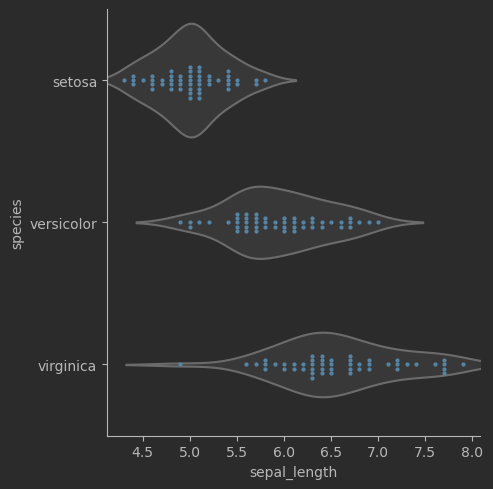
# 蜂群圖 + 小提琴圖,考慮鳶尾花分類sns.catplot(data=iris_sns, x="sepal_length", y="species", kind="violin", color=".9", inner=None)sns.swarmplot(data=iris_sns, x="sepal_length", y="species", size=3)
二元特征數據
散點圖
通過散點圖可以簡要查看兩個維度是否有何關系
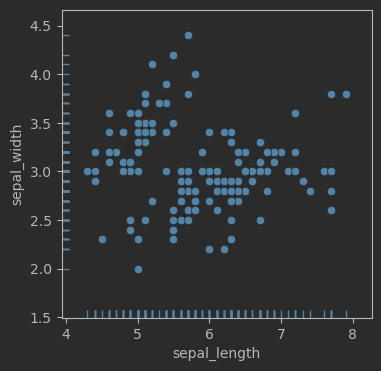
# 鳶尾花散點圖 + 毛毯圖
fig, ax = plt.subplots(figsize = (4,4))sns.scatterplot(data=iris_sns, x="sepal_length", y="sepal_width")
sns.rugplot(data=iris_sns, x="sepal_length", y="sepal_width")
效果:
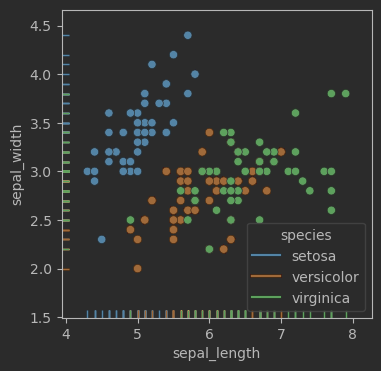
fig, ax = plt.subplots(figsize = (4,4))sns.scatterplot(data=iris_sns, x="sepal_length", y="sepal_width", hue = 'species')
sns.rugplot(data=iris_sns, x="sepal_length", y="sepal_width", hue = 'species')fig.savefig('Figures\二元,scatterplot + rugplot + hue.svg', format='svg')
效果:
二元直方熱圖
二維散點圖轉化為直方圖后效果并不清晰
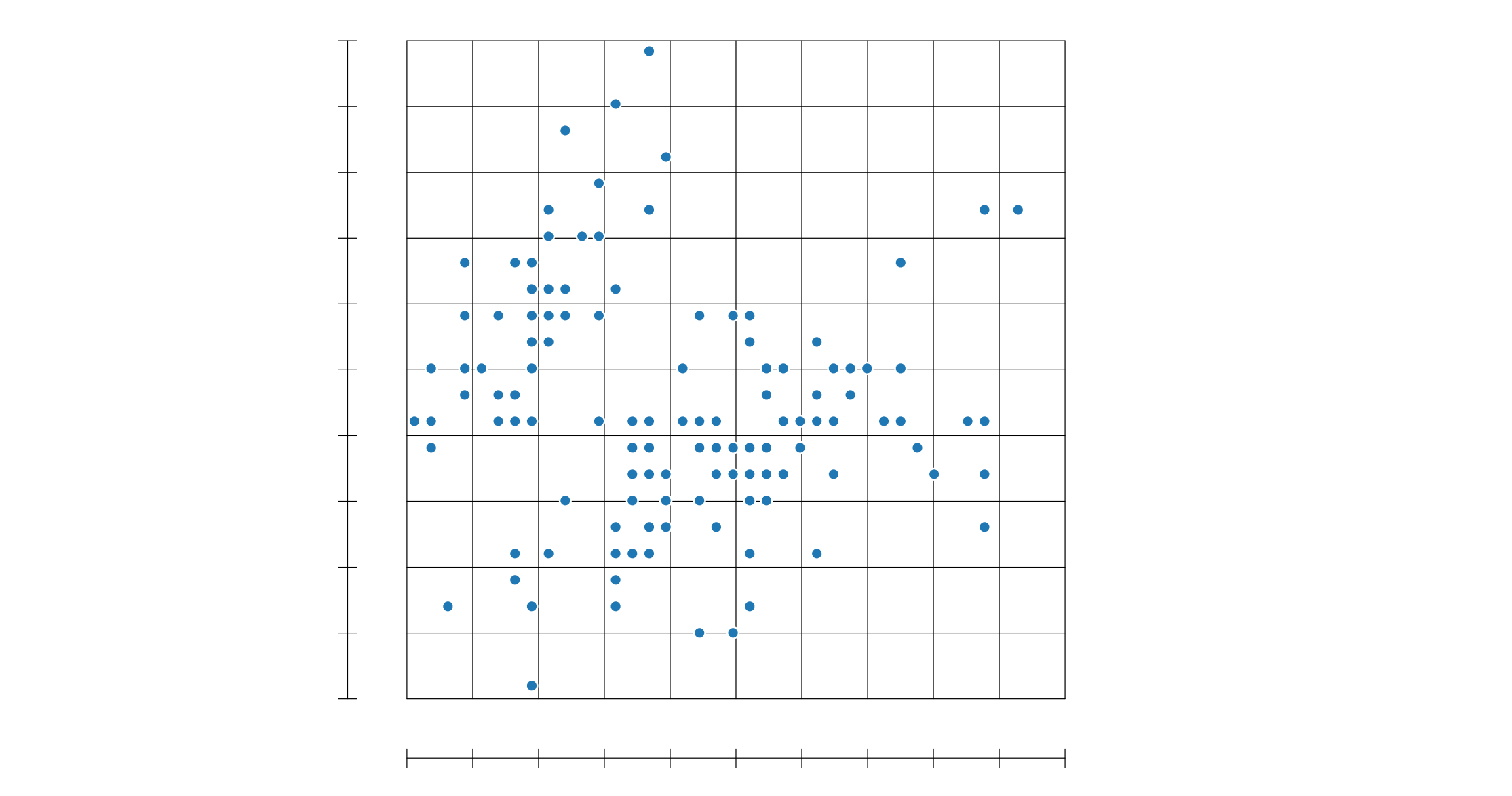

因此采用二維熱力圖:
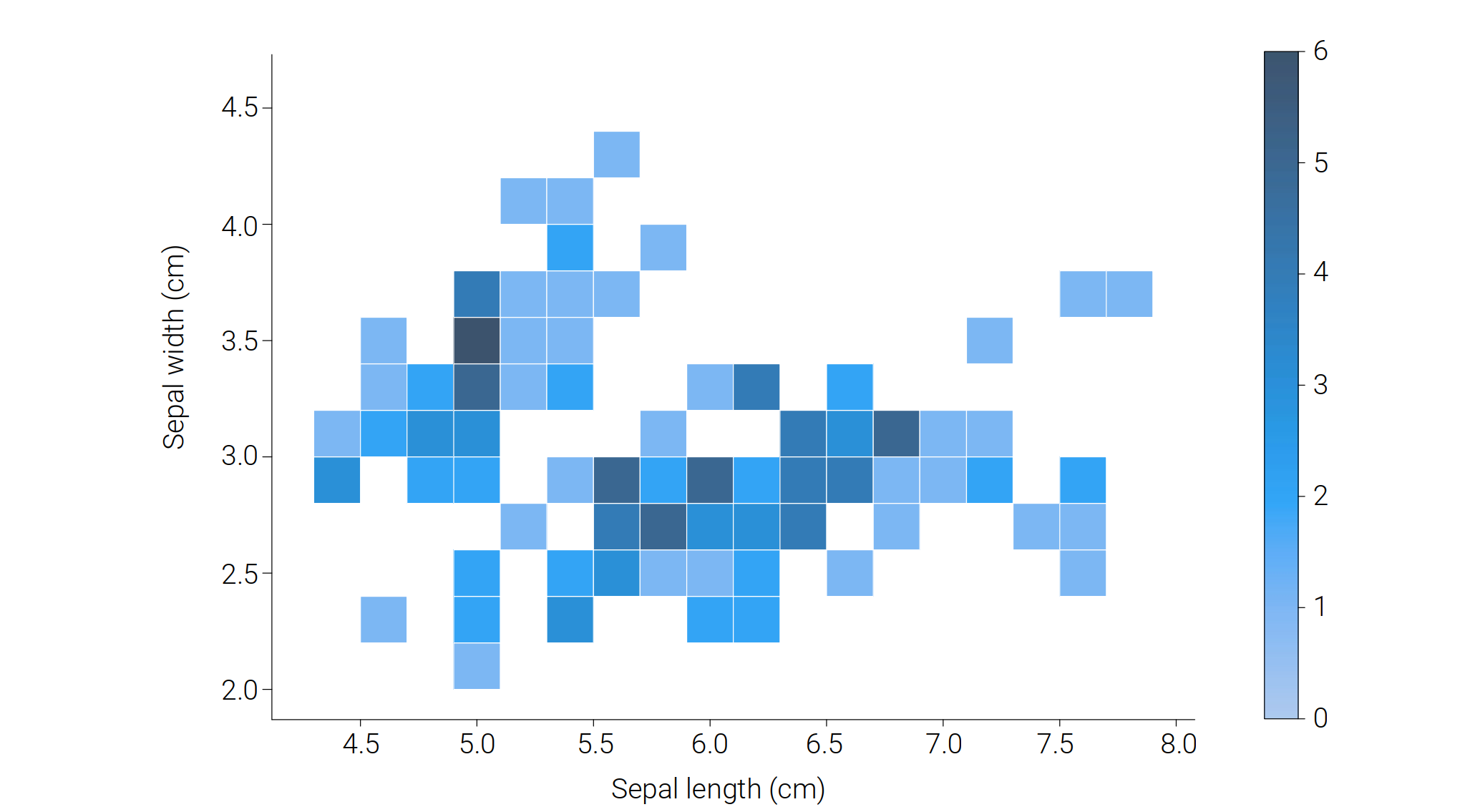
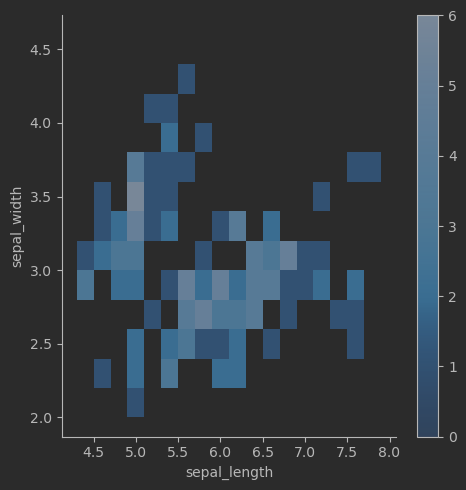
# 鳶尾花二元頻率直方熱圖sns.displot(data=iris_sns, x="sepal_length", y="sepal_width", binwidth=(0.2, 0.2), cbar=True)
聯合分布 KDE
使用高斯核函數可以估算聯合分布,這樣的聯合分布可以用等高線圖表示。
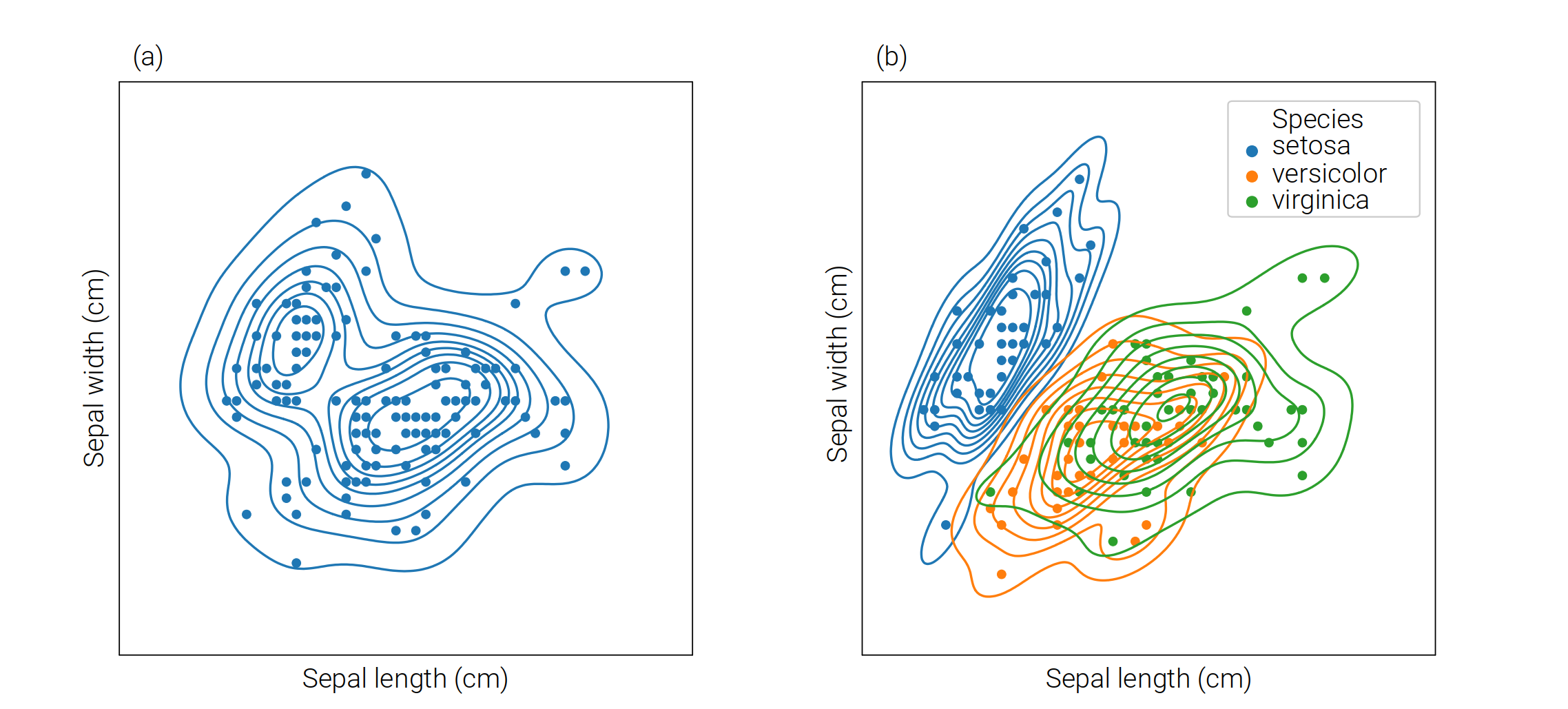
# 聯合分布概率密度等高線
sns.displot(data=iris_sns, x="sepal_length", y="sepal_width", kind="kde")
效果:
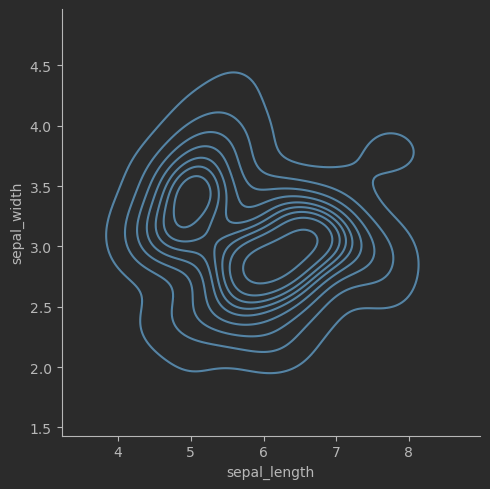
# 聯合分布概率密度等高線,考慮分布
sns.kdeplot(data=iris_sns, x="sepal_length", y="sepal_width", hue = 'species')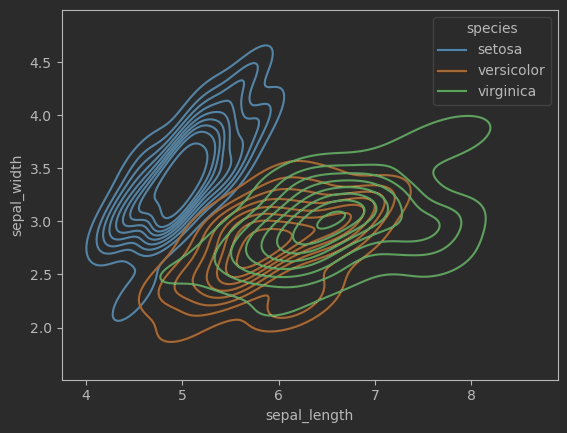
聯合分布+邊緣分布
看圖即可懂:
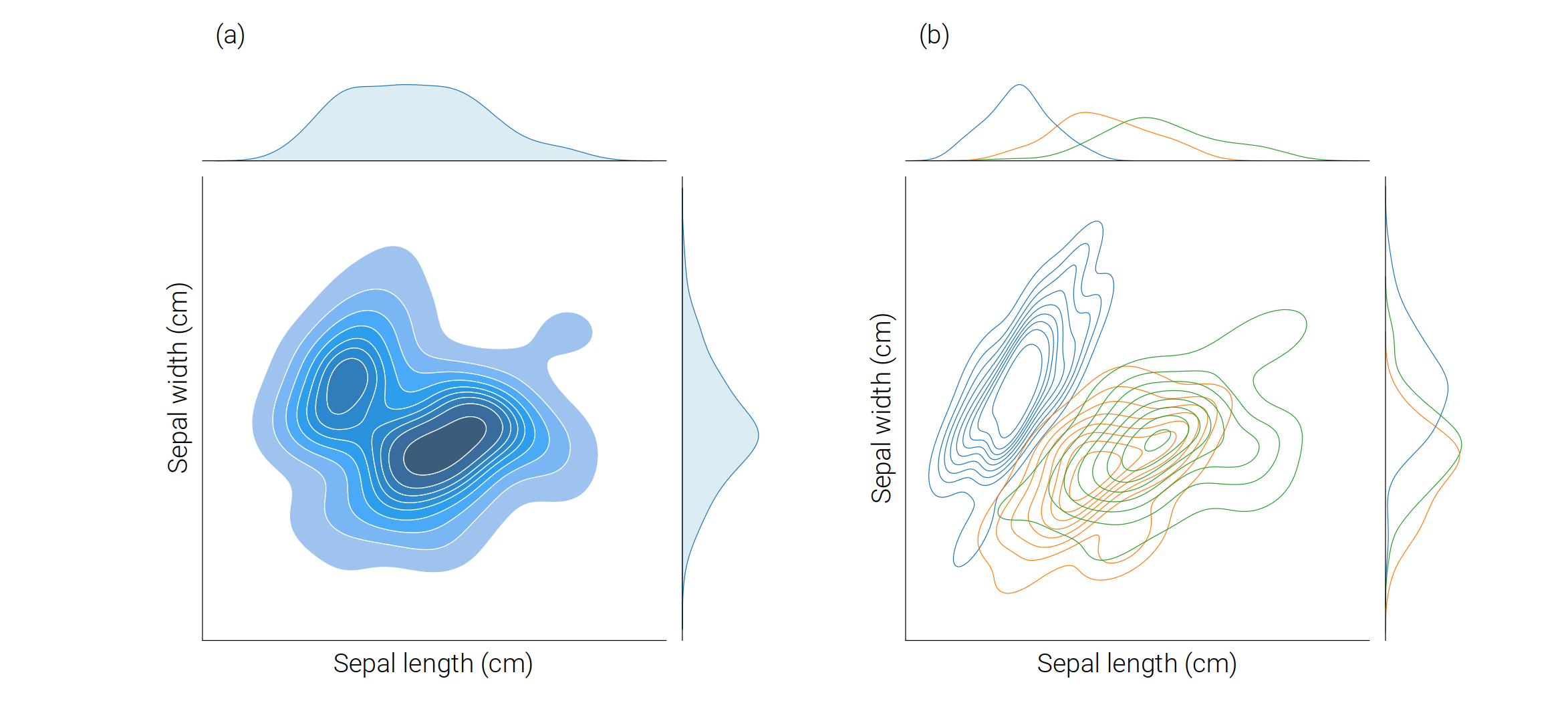
# 聯合分布、邊緣分布
sns.jointplot(data=iris_sns, x="sepal_length", y="sepal_width", kind = 'kde', fill = True)
這里僅放示范代碼,其他代碼查看附件。
線性回歸
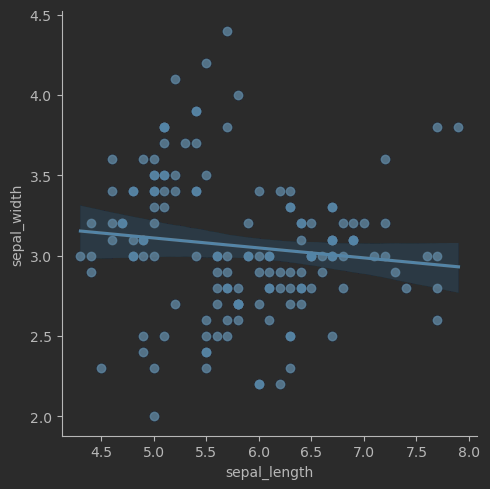
# 可視化線性回歸關系
sns.lmplot(data=iris_sns, x="sepal_length", y="sepal_width")
效果:
多元特征數據
可以用一元可視化方案展現多元特診
首先將寬格式轉化為長格式。
原來的寬格式:
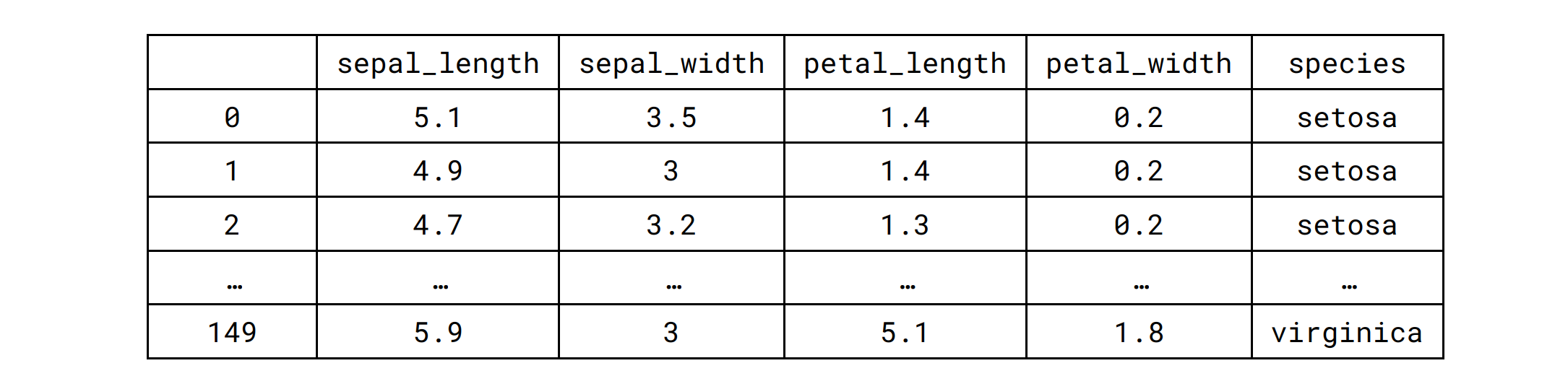
iris_melt = pd.melt(iris_sns, "species", var_name="measurement")
iris_melt
通過代碼結果可以查看長數據:
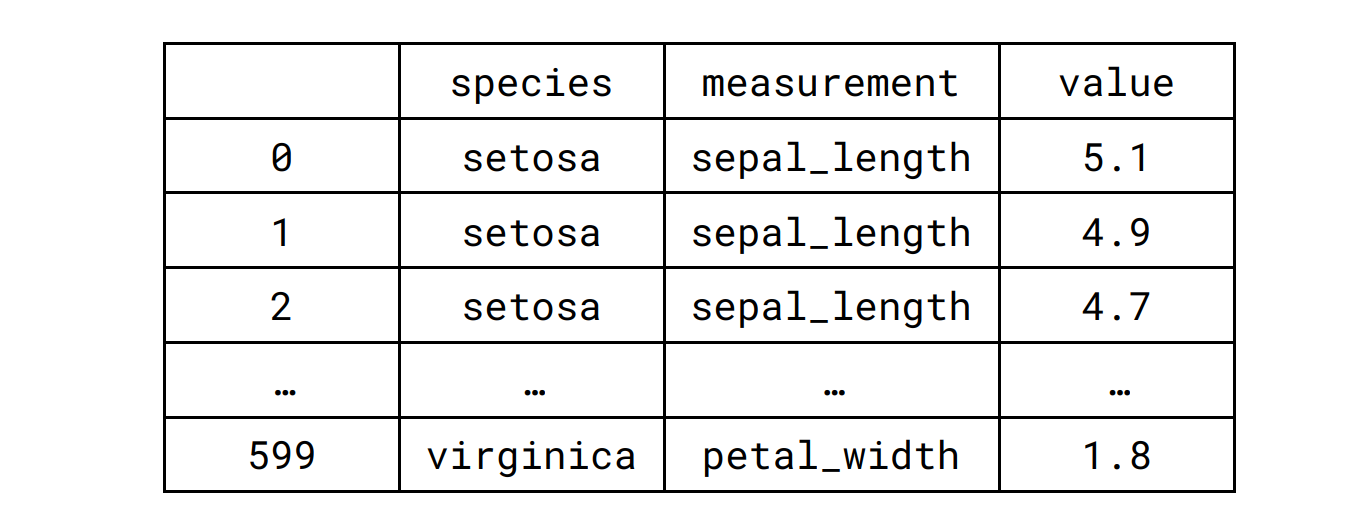
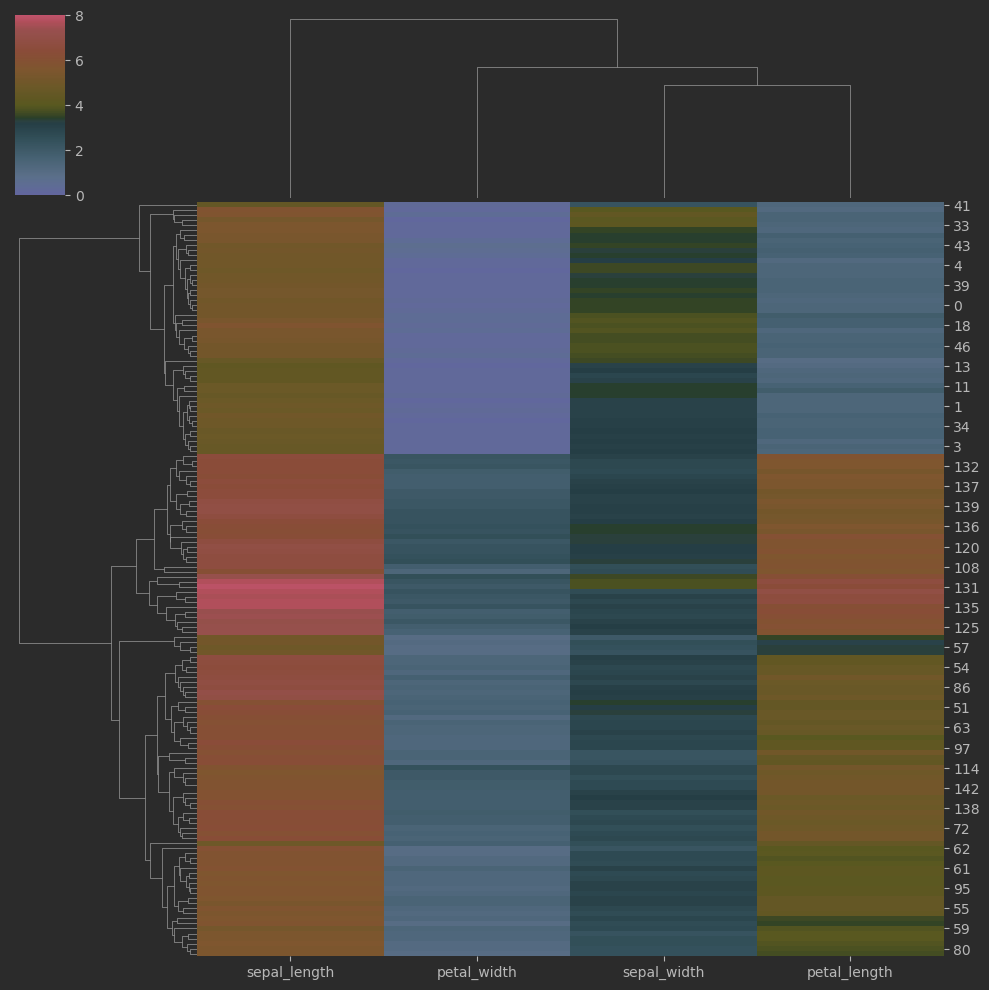
聚類熱圖
# 聚類熱圖
sns.clustermap(iris_sns.iloc[:,:-1], cmap = 'RdYlBu_r', vmin = 0, vmax = 8)
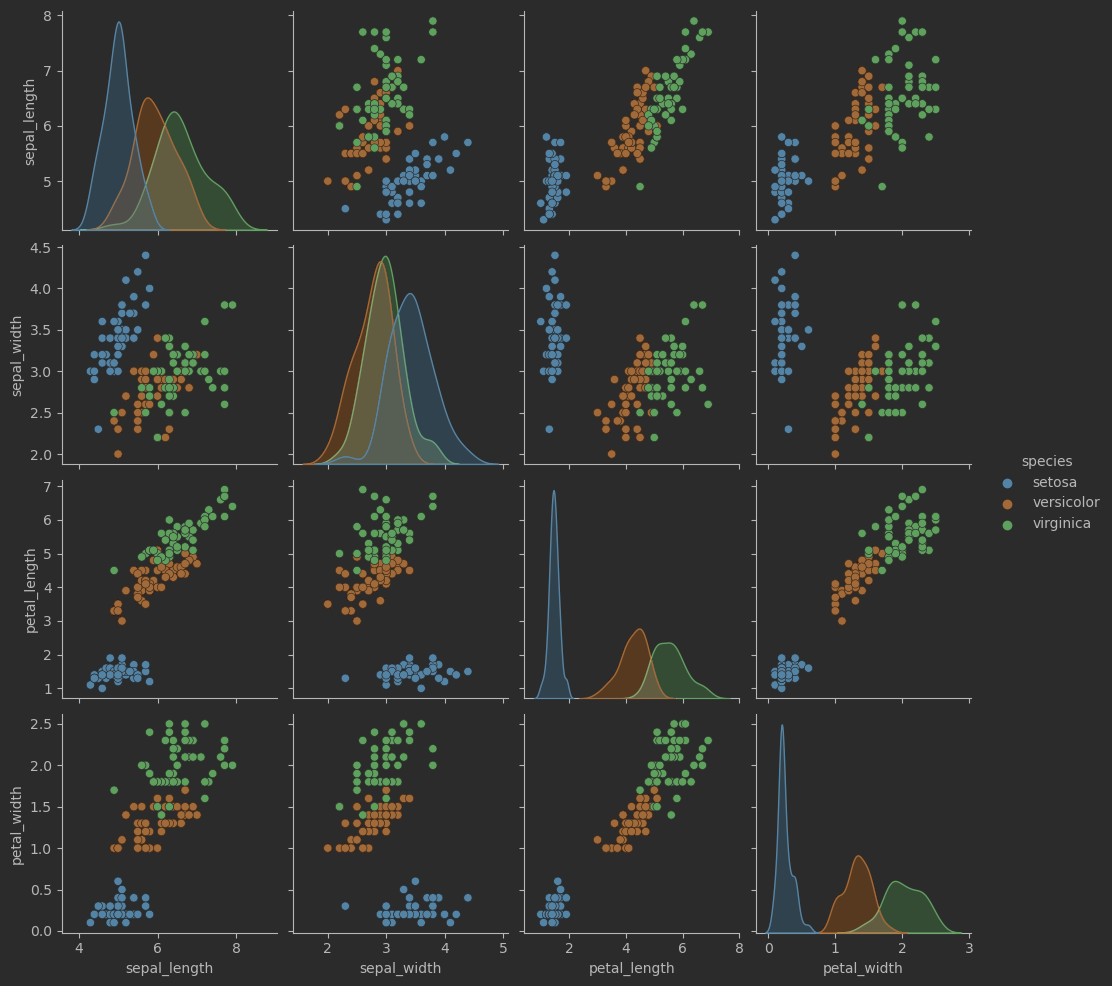
成對特征散點圖
sns.pairplot(iris_sns)
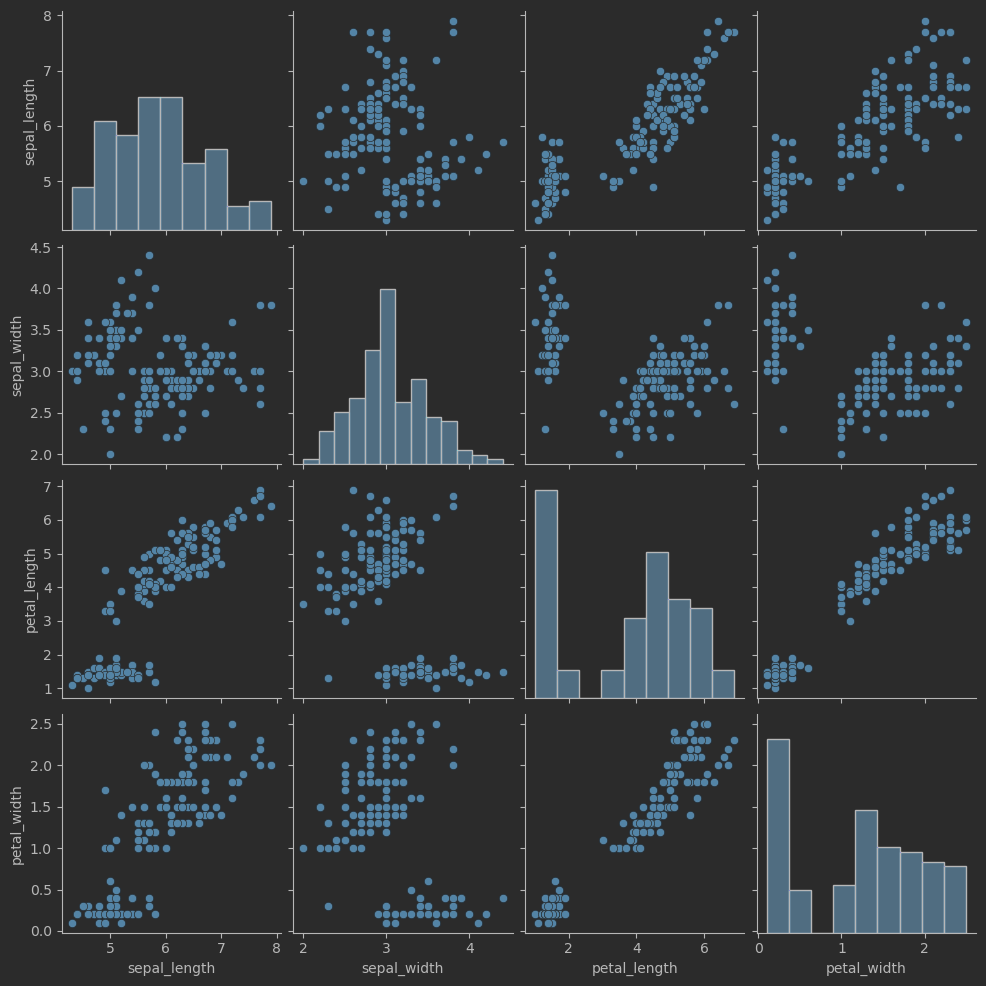
# 繪制成對特征散點圖
sns.pairplot(iris_sns, hue = 'species')
效果:
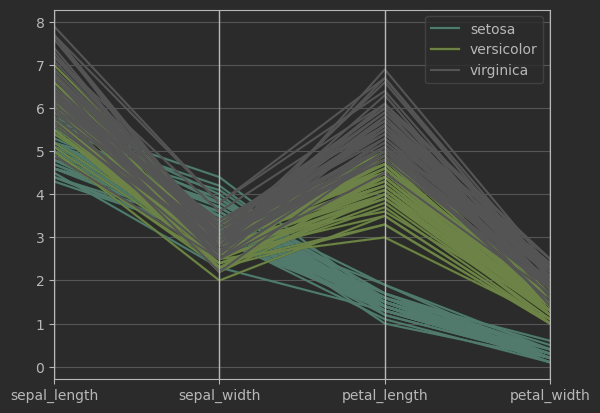
平行坐標圖
from pandas.plotting import parallel_coordinates
# 可視化函數來自pandas
parallel_coordinates(iris_sns, 'species', colormap=plt.get_cmap("Set2"))
plt.show()


)











:)




