說在前面
說起肺癌真的過去回憶歷歷在目,小編畢業后職業生涯的第一個項目——非小細胞肺癌預后有效靶點篩選。當時肝的是轉錄組預后+建模篩選。
做研發其實要求是遠遠高于發文章的,文章投不出去就降分,加工作量,做藥要是爛尾或者就算是有瑕疵直接寄,錢錢用之如泥沙。話不多說,欣賞一篇NSCLC的多組學分析
小編整理打包了文章多組學分析及copyKAT的代碼
文末公眾號后臺回復20240701lung即可領取項目代碼
今天給大家分享的一篇文章:Single-cell and spatial transcriptomics analysis of non-small cell lung cancer

- 標題:非小細胞肺癌的單細胞和空間轉錄組學分析
- 期刊名稱:Nature Communications
- 影響因子:14.7
- JCR分區:1區
- 中科院分區:綜合性期刊1區 Top
- 小類:綜合性期刊1區
摘要
肺癌是全球第二大最常見的癌癥,也是癌癥相關死亡的主要原因。腫瘤生態系統具有多種免疫細胞類型。髓系細胞尤其普遍,并且在促進疾病方面發揮著眾所周知的作用。在我們的研究中,我們通過單細胞和空間轉錄組學分析了 25 名未經治療的腺癌和鱗狀細胞癌患者的約 900,000 個細胞。我們注意到抗炎巨噬細胞與 NK 細胞/T 細胞之間存在反比關系,并且腫瘤內的 NK 細胞細胞毒性降低。雖然我們觀察到腺癌和鱗狀細胞癌中的細胞類型組成相似,但我們發現各種免疫檢查點抑制劑的共表達存在顯著差異。此外,我們還揭示了腫瘤中巨噬細胞轉錄“重編程”的證據,使其轉向膽固醇輸出并采用促進鐵流出的胎兒樣轉錄特征。我們的多組學資源提供了腫瘤相關巨噬細胞的高分辨率分子圖譜,增強了我們對其在腫瘤微環境中的作用的理解。
結果

- A 研究概覽。采用CD45+或CD235?富集的方法對切除的腫瘤組織、相鄰的非腫瘤組織(背景)和健康肺部的單細胞懸液進行了單細胞RNA測序(scRNA-seq)。使用新鮮、急速冷凍的腫瘤、背景和健康組織的冷凍切片進行了10x Visium空間轉錄組學研究。
- B 隊列概覽。符號代表單個病人和進行的分析。
- C UMAP投影顯示腫瘤和結合的背景+健康數據集。
- D 腫瘤樣本中用于廣泛細胞類型注釋的代表性基因的點圖。
- E 等高線圖展示AT2細胞(44,399個細胞)、CAMLs(2,520個細胞)和AIM?(16,120個細胞)中髓系(LYZ, CD68, MRC1)和上皮(EPCAM)基因的共表達。標準化、縮放和對數變換后的基因表達。
- F 盒形圖展示AT2細胞、CAMLs和AIM?中髓系(LYZ, APOE, CD68, MRC1)和上皮(EPCAM, KRT8, KRT19)基因的標準化、縮放和對數變換后的基因表達。

- A 熱圖顯示了每種免疫細胞類型(在CD235?富集中計算得到的)相對細胞類型豐度之間的皮爾遜相關系數。顏色表示皮爾遜相關系數值,星號表示雙側關聯測試的顯著性水平,該測試基于皮爾遜乘積矩相關系數計算。
- B 熱圖顯示LUAD(左)和LUSC(右)中所有細胞類型之間的LR(配體-受體)相互作用次數,通過廣泛的細胞注釋進行總結。行使用歐幾里得距離的完全連接方法進行了層次聚類。
- C Sankey圖顯示了cellphoneDB檢測到的LUAD和LUSC中選定的ICI(免疫檢查點抑制劑)的腫瘤特異性相互作用。線條顏色標識每種細胞類型之間的LR相互作用是僅在LUAD中發現(橙色)、僅在LUSC中發現(綠色)還是在兩種腫瘤類型中都有(藍色)。
- D 點圖顯示了?中突出顯示的ICI基因和細胞類型,按腫瘤類型分開。每個點的大小代表該簇中表達該基因的細胞百分比,而顏色表示每組中每個基因的平均標準化縮放對數變換表達量。
- E Sankey圖顯示了cellphoneDB檢測到的LUAD和LUSC中VEGFA/B相互作用者的腫瘤特異性相互作用。線條顏色標識每種細胞類型之間的LR相互作用是僅在LUAD中發現(橙色)、僅在LUSC中發現(綠色)還是在兩種腫瘤中都有(藍色)。
- F Sankey圖顯示了cellphoneDB檢測到的LUAD和LUSC中EGFR相互作用者的腫瘤特異性相互作用。線條顏色標識每種細胞類型之間的LR相互作用是僅在LUAD中發現(橙色)、僅在LUSC中發現(綠色)還是在兩種腫瘤中都有(藍色)。

- A 空間圖像展示了用cell2location估計的代表性腫瘤切片上AT2細胞、AIM?和Tregs的細胞豐度。
- B 使用cell2location對腫瘤和背景切片的細胞豐度估計,計算了免疫(左側)和非免疫(右側)細胞類型的相對比例。免疫細胞按照寬泛的注釋分組。箭頭表示與背景相比,在腫瘤中的增加(↑)或減少(↓)。使用雙側Wilcoxon秩次檢驗和Bonferroni校正進行了成對比較。
- C 空間LR共定位的熱圖。在每個斑點上估計了所有切片的LR基因對共表達,使用χ2檢驗并隨后進行Bonferroni多重比較校正,比較了腫瘤和背景中共定位與非共定位斑點的頻率。深灰色磚塊表示腫瘤和背景切片中共定位基因對的頻率顯著不同。綠色列注釋表示至少在八個病人中四個以上的LR對顯著。行注釋指示腫瘤類型。
- D 盒形圖顯示在每個分析的切片中,共定位LR對在腫瘤與背景間頻率顯著不同的情況。N=8個病人。盒形圖使用Python Seaborn包的默認設置繪制,即盒子顯示四分位數,胡須長度是四分位距的1.5倍。源數據提供為源數據文件。
- E 空間圖像展示了在腫瘤(頂部)和背景(底部)中發現LR對共表達的斑點的位置,對于NRP1-VEGFA, NECTIN2-TIGIT, PD1-PDL1, CD96-NECTIN1 和 HAVCR2-LGALS9。來自一位病人的代表性切片。
-
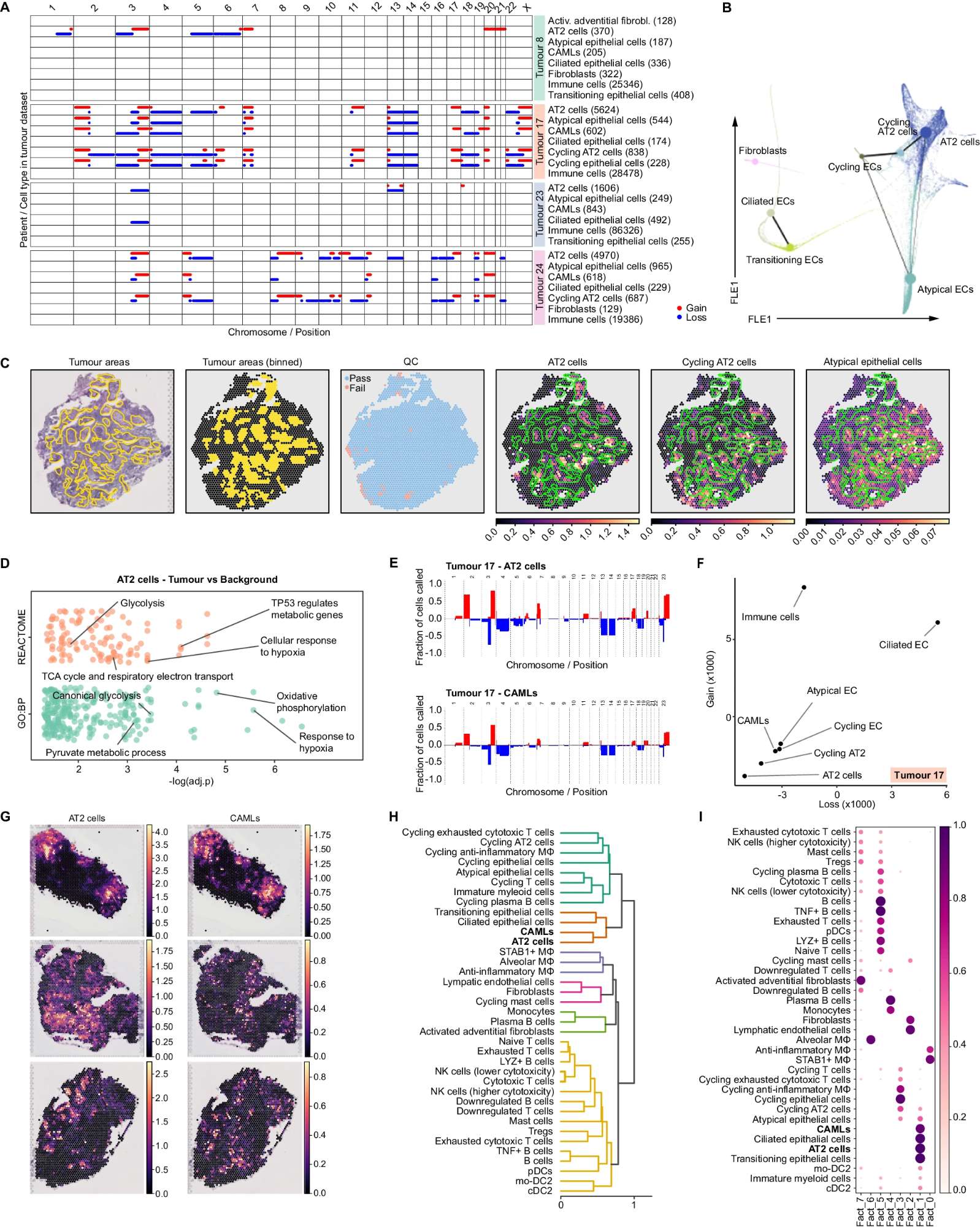
A CNA分析: 圖表展示了通過CopyKat估計的每個染色體臂上不同細胞類型和患者的染色體增益(紅線)和損失(藍線)。所有免疫細胞類型為繪圖目的被分組在一起。
-
B PAGA圖層:在腫瘤中非免疫細胞類型的擴散圖(FLE嵌入)上疊加PAGA圖。
-
C 盲注釋:前三個面板—資深病理學家標注的腫瘤浸潤區域(左),Visium點上的腫瘤區域分桶(中)和通過QC的點(右)。后三個面板—在同一切片上的cell2location估計的AT2細胞(左)、周期性AT2細胞(中)和非典型上皮細胞(右),疊加了病理學家對腫瘤浸潤的注釋(綠色輪廓)。
-
D 基因本體和REACTOME數據庫的超表達分析:使用clusterProfiler R包,基于AT2細胞在腫瘤與背景中上調的差異表達基因(DEGs),對生物過程進行超表達分析。提供源數據文件。
-
E 一個代表性患者腫瘤中AT2和CAMLs的CNA詳細概覽:條形圖顯示特定染色體區域中攜帶染色體增益(紅色條)或損失(藍色條)的細胞頻率。
-
F 散點圖顯示KL散度:用于計算腫瘤數據集中每種細胞類型的增益和損失分布之間的KL散度,損失在x軸,增益在y軸。所有免疫細胞類型為繪圖目的被分組在一起。
-
G 空間圖像:展示cell2location估計的三個代表性腫瘤切片上AT2細胞和CAMLs的細胞豐度。
-
H 層次聚類:計算通過QC的所有腫瘤切片中點的細胞類型組成(由cell2location估計)的相關距離。
-
I 非負矩陣分解:建立在通過QC的所有腫瘤切片中點的細胞類型豐度的q05估計上,使用cell2location估計。
-
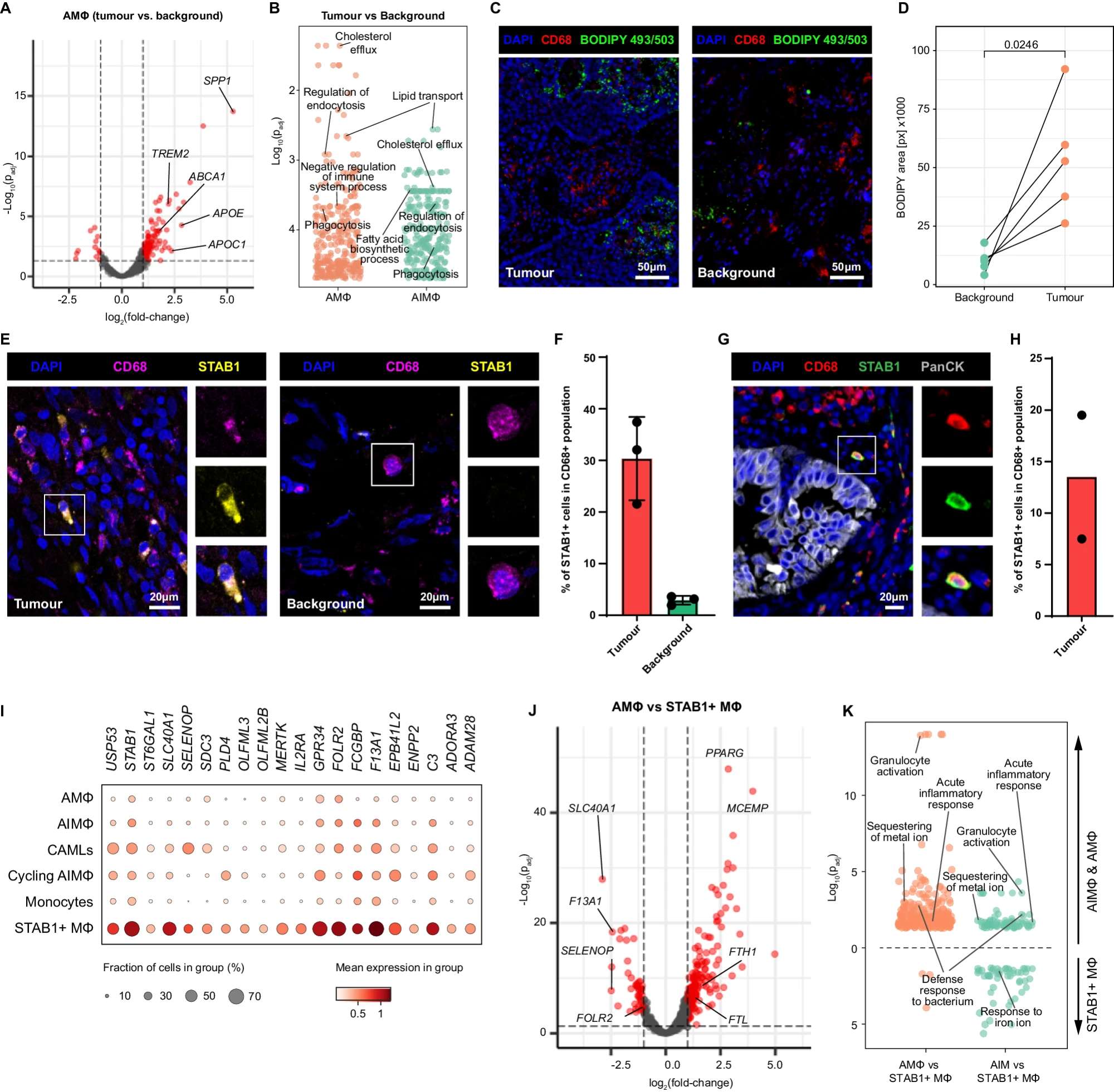
A 火山圖分析:使用py_DESeq2包提取的AIM?在腫瘤與背景比較中的差異表達基因(DEGs,紅色表示)的火山圖。
-
B 基因本體超表達分析:使用clusterProfiler R包對Alveolar M?和AIM?在腫瘤與背景中上調的DEGs進行基因本體—生物過程數據庫的超表達分析。提供源數據文件。
-
C 免疫組化染色(IHC):在腫瘤和背景組織切片上對CD68和中性脂類(BODIPY 493/503)進行免疫組化染色。Z堆疊的最大強度投影。標尺為50微米。
-
D BODIPY信號覆蓋面積分析:比較腫瘤與背景切片中BODIPY信號的覆蓋面積,使用配對的雙側t檢驗,匹配同一患者的腫瘤和背景切片。N=5個患者。提供源數據文件。
-
E CD68和STAB1的免疫組化染色:在腫瘤(左)和背景(右)組織切片上進行CD68和STAB1的免疫組化染色。Z堆疊的最大強度投影。小插圖顯示單個細胞的詳細放大。標尺為20微米。
-
F STAB1+細胞的定量:顯示CD68+巨噬細胞群體中STAB1+細胞的比例,以CD68+區域的百分比表示。數據以平均值和標準偏差表示(n=3個生物重復)。提供源數據文件。
-
G CD68, STAB1和PanCK的染色:在腫瘤組織切片上進行CD68, STAB1和PanCK的染色。Z堆疊的最大強度投影。小插圖顯示單個細胞的詳細放大。標尺為20微米。
-
H STAB1+ CD68+細胞的定量:在非小細胞肺癌(NSCLC)中,定量CD68+巨噬細胞群體中的STAB1+ CD68+細胞。數據以平均值和單個數據點表示(n=2個生物重復)。提供源數據文件。
-
I 點圖:顯示腫瘤中所有巨噬細胞亞群和CAMLs中的“STAB1標志基因”表達。
-
J 火山圖分析:由py_DESeq2識別的Alveolar M? vs STAB1 M?在腫瘤中的DEGs的火山圖(紅色表示)。
-
K 基因本體超表達分析:使用clusterProfiler R包對Alveolar M? vs STAB1 M?(頂部)和AIM? vs STAB1 M?(底部)在腫瘤中的DEGs進行基因本體—生物過程數據庫的超表達分析,分別顯示STAB1 M?上調(左側)和Alveolar M?或AIM?上調(右側)的DEGs。提供源數據文件。
-
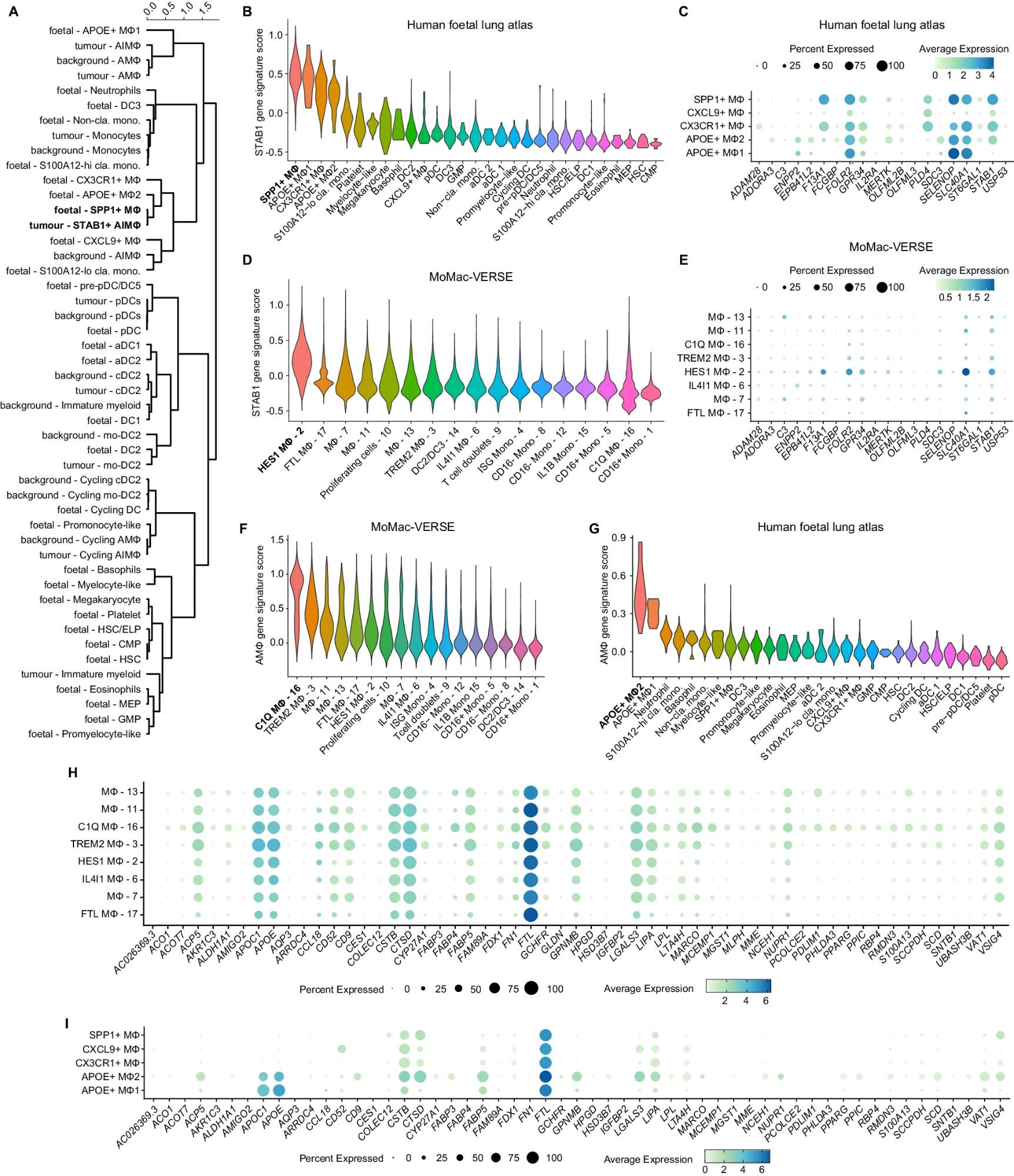
A 層次聚類分析:對在調和后的PC空間(腫瘤髓系 + 背景髓系 + 胎兒肺髓系)的每個細胞計算的相關距離進行層次聚類。
-
B 小提琴圖:顯示在公開可用的人類胎兒肺圖譜中識別的髓系細胞和祖細胞群體中“STAB1基因標志”的表達水平。
-
C 點圖:顯示選定的胎兒肺巨噬細胞群體中“STAB1基因標志”的每個基因的表達。每個點的大小代表群體中表達該基因的細胞百分比,顏色代表每個群體中每個基因的平均表達水平。
-
D 小提琴圖:顯示在公開可用的MoMac-VERSE數據集中識別的群體中“STAB1基因標志”的表達水平。
-
E 點圖:顯示MoMac-VERSE中選定的巨噬細胞群體中“STAB1基因標志”的每個基因的表達。每個點的大小代表群體中表達該基因的細胞百分比,顏色代表每個群體中每個基因的平均表達水平。
-
F 小提琴圖:顯示在公開可用的“MoMac-VERSE”數據集中識別的髓系細胞和祖細胞群體中“AM?基因標志”的表達水平。
-
G 小提琴圖:顯示在公開可用的人類胎兒肺圖譜中識別的髓系細胞和祖細胞群體中“AM?基因標志”的表達水平。
-
H 點圖:顯示“MoMac-VERSE”數據集中識別的選定巨噬細胞群體中“AM?基因標志”的每個基因的表達。每個點的大小代表群體中表達該基因的細胞百分比,顏色代表每個群體中每個基因的平均表達水平。
-
I 點圖:顯示在選定的胎兒肺巨噬細胞群體中“AM?基因標志”的每個基因的表達。每個點的大小代表群體中表達該基因的細胞百分比,顏色代表每個群體中每個基因的平均表達水平。
小結
- 主要數據及方法:
| Types | Notes |
|---|---|
| 分析數據 | BioStudies數據庫:E-MTAB-13526和E-MTAB-13530,其他的在補充文件里 |
| 分析方法 | 單細胞標準流程(從定量到注釋);差異分析;scArches聚類;CellPhoneDB;CopyKAT拷貝數分析;PAGA軌跡分析;cell2location空轉標準流程 |
| 實驗技術 | 流式;免疫組化和染色;多重免疫熒光 |
 葉子游戲新聞)
)






的語法及在對應不同需求下應如何使用)

)






-指針)

