最近需要仿真研究T1結構像+RS-fMRI影像融合處理輸出目標坐標的路線可行性。就此機會記錄下來。
為了完成驗證目標處理,首先需要有數據,然后需要準備對應的處理平臺和工具箱,進行一系列。那么開始記錄~
前言:
為了基于種子點的功能連接分析,找出與種子點的最相關坐標。如下是嚴老師的培訓文檔,正是我所需要的。鏈接如下:
https://d.rnet.co/Course/V3.0EN/4_R-fMRI_Data_Processing_DPARSFA.pdf
https://d.rnet.co/Course/V3.0EN/4_R-fMRI_Data_Processing_DPARSFA.mp4


確實做功能連接是我這次處理的目的,我需要先完成預處理,后才基于種子ROI去做功能連接分析,既可以分析種子ROI和目標腦區的連接情況,也可以分析種子ROI與全腦的功能連接情況。但是可以想象,兩個計算量肯定有明顯的差異。
萬里長征第一步,還是開始嘗試吧。
正文:
(1)下載滿足要求的開源數據
去OpenNEURO https://openneuro.org/datasets/ds002422/versions/1.1.0
下載數據做仿真驗證夠夠的了,有46個被試,每個被試下面有多個數據記錄,完全符合我的需要,有這個做仿真研究真的是太方便了。
(sub-01_T1W.nii就是T1結構像,sub-01_task-rest_bold.nii.gz就是我要的RS-fMRI)
多下載幾個數據處理驗證沒得問題。

也可以去嚴老師的網站下載數據,不同類別的data可供下載,可以直接下載Demo Data for DPARSF。
https://rfmri.org/content/demonstrational-data-resting-state-fmri
(2)準備數據處理平臺
按照如下準備先按照到位,這些是處理和查看處理過程的利器,作為研究核磁影像的人少不了。

安裝完Matlab以后,除了MRIcron 是獨立軟件,其他就都是工具箱。工具箱軟件需要先下載到本地,后解壓縮放到Matlab 安裝路徑的toolbox 文件夾下,并打開matlab 界面,從設置路徑處導入。這里我是遇到了一個問題,到底是 添加文件夾 還是 添加并包含子文件夾?

那么看下面

澄清一下:如果打算使用DPABI 做預處理,那么這個地方的SPM12 和DPABI的設置路徑的方式一定要參考嚴老師的官網
使用DPABI 一定要去看:http://www.rfmri.org/DPABIErrorHandling

REST 是DPABI 的一個子模塊。
(3)實施處理
都說DPABI 是“傻瓜版”的SPM,但是這個用起來還是要花時間去仔細啄米…
先說一下數據擺放路徑,非常容易出錯,可以先去閱讀網站 http://www.rfmri.org/DPABIErrorHandling
如果用DPABI 來做預處理,那數據擺放路徑就是講究,路徑關系如果層級發生錯誤,就會報各種奇怪的錯誤。
首先新建一個路徑文件夾,如202504_MRI是我的文件夾,文件夾下面需要新建兩個子文件夾:
如果原始數據都是Dicom 格式,新建T1Raw 和FunRaw 文件夾,并將各被試的T1像拷貝到T1Raw文件夾,RS-fMRI 像拷到FunRaw文件夾。
如果T1是nii格式,RS-fMRI 是.nii.gz 格式就新建T1Img 和FunImg文件夾,并將3個被試的T1像拷貝到T1Img文件夾,RS-fMRI 像拷到FunImg文件夾,我這次的數據就是這一種。
另外,一定要注意,如果使用DPABI進行預處理,這些原始數據的父文件夾是T1Img 和FunImg文件夾,或T1Raw 和FunRaw 文件夾,我曾經將sub當父文件夾出現了難以接受的錯誤。正確的數據層級關系如下:
當原始數據是.nii+.nii.gz 格式時:

當原始數據是Dicom格式時:

這里插一個處理步驟,因為我看有人講一個4D.nii.gz 容易報錯,需要轉化成多個3D ni。
我的fMRI 影像是一個文件4D.nii.gz 文件,然后我通過手動解壓以后變成了只變成了一個nii。如果想變成多個3D nii,還是需要工具或者代碼實現。下面提供一段可靠的代碼4D.nii.gz 轉成多個3D nii的matlab:
clc;clear;
fileName = 'sub-03_task-rest_bold.nii.gz';%需解壓的fMRI 的原始4D 文件壓縮格式文件名,
filepath = 'E:/202504_MRI/';%fMRI 的原始4D 文件路徑
gz_file = fullfile(filepath,fileName);tempniiPath = 'E:/202504_MRI/temp/';%初解壓縮的nii文件存儲路徑,臨時的,每完成一個數據的解壓以后都要建立一個temp
gunzip(gz_file,tempniiPath);%手動初解壓縮fileList = dir(fullfile(tempniiPath,'*.nii*'));
outFile = fileList.name;%fMRI 手動初步解壓縮后的文件名
inputFilename = fullfile(tempniiPath ,outFile);%解壓后的完整文件名outDir = 'E:/202504_MRI/FunImg/sub3/';%解壓成3D nii 的存儲路徑spm('Defaults','fMRI');
spm_jobman('initcfg');
% 創建 batch job
matlabbatch{1}.spm.util.split.vol = {inputFilename};
matlabbatch{1}.spm.util.split.outdir = {outDir}; % 輸出目錄
matlabbatch{1}.spm.util.split.prefix = 'vol_'; % 輸出文件名前綴
spm_jobman('run',matlabbatch);
(2)預處理步驟雖多,但是處理的過程有講究

在matlab 窗口輸入 DPARSFA(是DPARSF advance 版本,支持T1+fMRI 聯合分析)后即可進入DPARSFA 交互界面
①模版參數
選中Blank,其實就是空白模版,其他的都有很多默認的參數和處理步驟被選中是有特定的處理,作為新手來說,先從0開始進入吧,選中blank就相當于空白開始,滅有任何的默認選中處理步驟,需要自己去嘗試設定探索;
這個地方參數模版,是一些供選擇的處理方案,感覺是處理步驟流程模版化,選擇不同的模版會在界面上出現對應選中處理步驟,估計也會存在一些差異性的處理流程步驟,如果很清楚自己的處理目標和步驟,就可以直接選中適合的一個,后面只要把數據掃描關聯的參數填下就可以run了。
但是這個地方要結合自己的處理目標去分析。
如果只需要做預處理,那只能選擇blank,其他的選項中不管是在MNI空間還是original空間 都是默認選中了預處理+后處理。

②工作路徑
這個工作路徑需要選中建立的文件夾,是一個總的大路徑,不要出現中文字符;
③掃描參數
Time Points:采集的時間點數,應該是個整數,預估100~300,具體需要查看數據
TR:是重復時間,這個也是掃描參數,一般這兩個參數需要基于fMRI 數據去查看,如果已知就直接填寫
這兩個參數必須要準確,如果不知道,可以通過軟件查看,實在不行就空著,軟件會在處理的過程中讀取到。
建議在matlab 中通過命令得到點數,這個是.nii.gz 的獲取方式
nii=niftiinfo("my-rs-fMRI.nii.gz");
TimePoints = nii.ImageSize(4);
disp(['時間點總數:',num2str(TimePoints)]);dicom也有其獲取參數的方式,比如先轉成nii查看,或者用MRIcron/MRIcronGL/3D slicer/FSL /等軟件打開查看。
TR(s)這個參數重要,就是重復時間,這個需要基于掃描json文件去查看,也可以用別的軟件工具去查看。
④就是將dicom轉成nifti格式,由于我的輸入就不是dicom,這一步完全不用選。

上面沒有標出來,Apply Mats,如果輸入是dicom 格式數據,那就選中吧,如果是我這樣的nii格式就不選
⑤ 去除因設備開機不穩的時間點,一般會去掉開始的5~10個時間點的數據。這里,我沒有去,因為我下載的數據中已經在json中說了手動去掉了3個時間點,我就不再去了,直接填了0,也可以不選中,省的有效數據變短。
⑥時間層矯正,因為功能像是隔層掃描,做這個處理就是要重新把時間對齊,必須要選中;
⑦功能像的掃描層數,就是ImageSize(3),我下載的數據是64X64X36X200,我這里就是36層
后面的Slice Order:掃描次序,我填的[2:2:36,1:2:35](我看了我下載的json文件,我的是隔層掃描,先掃偶數層,后掃奇數層)
⑧參考層,一般用正中間的掃描時刻對應的層作為參考層,也有TR/2最接近的時刻處的層作為參考
我的掃描時間序列
“SliceTiming”: [1.5375,0,1.6225,0.085,1.7075,0.1725,1.7925,0.2575,1.8775,0.3425,1.9625,0.4275,2.05,0.5125,2.135,0.5975,2.22,0.6825,2.305,0.7675, 2.39,0.855,2.475,0.94,2.56,1.025,2.645,1.11,2.7325,1.195,2.8175,1.28,2.9025,1.365,2.9875,1.4525 ],
可以看出0時刻在第2幀,而且偶數層比奇數層的時刻小,說明先偶數后奇數。
⑨ 表明數據開始處理的路徑這樣可以附加到工作路徑文件夾。
⑩ 運行時間層矯正
看看做完時間層矯正后的結果,可以看到matlab 窗口中輸出說明我的參數設置正確了,正確完整了時間層矯正,而且還是3個sub都處理了


頭動矯正

Realign:必須要選中
Voxel-Specific Head Motion :因為我計算的目標是坐標靶點,對空間定位和連接精度非常敏感,我這里也需要選中,相當于是需要做一個更高級一些些的頭動矯正。普通的核磁處理可以不選中。有人說這一步時間長作用不大,我還是先處理看看。
根據上面,我處理的輸入是要基于時間層矯正的結果,所以我界面的這個輸入文件夾是時間層矯正的輸出
處理完以后輸出又多了更多文件夾

影像重定向及分割配準

(11 Reorient Fun : 處理fMRI,就是是的頭的朝向變正(因為很多情況下存在數據歪斜)
手動調整功能像歪斜,此處需要手動確立AC點。
(12 AutoMask :處理fMRI 自動掩膜生成,就是基于T1結構像,去掉不是大腦區域的部分,生產掩膜圖像;
這個步驟其實是對頭動矯正以后的fMRI 影像的平均圖像進行處理的,可以得到平均圖像去除顱骨以后模版mask。
(13 Crop T1: 就是去掉T1中的無關區域,比如脖子,背景、空氣、頭皮,從而減少圖像中的無效空白部分;
(14 Reorient T1: 處理結構像數據,結構像圖像重新定向,矯正朝向和歪斜
需要在彈窗中手動標記AC 點。
(15 Bet :T1 結構像處理,剔除顱骨,提取大腦區域
(16 T1 coreg to Fun: 對齊結構像和功能像,用coregistration 將fMRI 與T1 進行對齊,其實就是將T1結構像配準到fMRI 空間,從而實現兩者空間對齊。
這個步驟完成了不少內容,首先講去掉顱骨的的T1,批準到了去掉頭皮的fMRI 上,并得到了中間計算參數,可以講帶顱骨的T1配準大片fMRI 空間。
(17 segment :分割,將T1結構像分割成灰質、白質、腦脊液,segment 是傳統的分割方法,New segment+DARTEL 是一種新的分割方法(如果T1結構像的質量很好建議選擇這個方法)
這個處理步驟,其實是先將已配準到fMRI 平均影像的T1 又配準到標準空間,這樣得到了中間形變參數。同時對標準空間下的T1進行分割處理,分割出腦灰質、腦白質、腦脊液等掩膜圖像。
(18 分割處理的模版選擇,East Asian 是東亞模版比較適合處理東方人的數據(基于東亞數據得到),European是歐美數據模版(基于歐美數據得到,其實就是MNI)
我這個數據下載的,查閱以后還是先選擇歐美模版。

注意11和14都是需要手動調整數據朝向和歪斜的,新手還無從下手,暫時先不選。另外如果數據本來就很正,AC-PC都有差不多水平了,而且也沒有左右歪斜就要不選。如果要矯正就要研究其選中后的彈窗頁面的調整,等我學會了,我再來補充一下。
11和14選中以后都需要手動去差點參考點,會彈窗讓手動操作選擇。
先看看AC-PC:



上面圖示中給出了AC和PC應該落點的位置,可見AC和PC在上下方向幾乎是等高的。
在彈出的頁面,手動標記參考時,需要講AC作為參考點,先理解了AC和PC的結構特點就好落點了。
這一步的輸出:
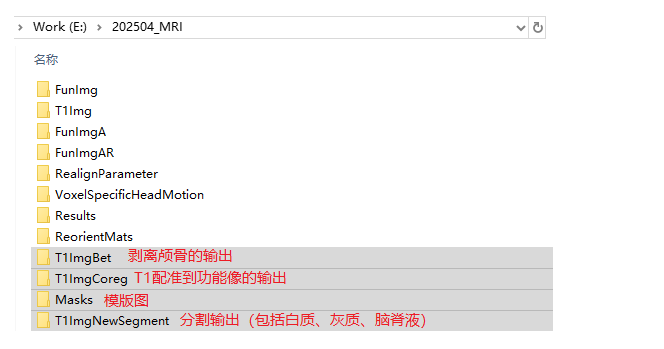
下面用軟件逐一打開查閱
T1ImgBet 就是T1結構像做了顱骨剔除以后的大腦區域的圖像。
如下圖可以看出sub1剔除的的比較差,在小腦下還包含了一些其他的組織,sub2有少許雜質組織,sub3剔除的最好,如果演示我肯定用sub3被試的處理結果去演示。

T1ImgCoreg:
這個是T1配準到fMRI 以后的結果,我也打開看看影像的變化
很遺憾,這個文件夾中的數據跟T1Img 中的數據大小和空間方位還有可視化一模一樣,說明T1 Coreg to Fun 是不成功了,為啥不成功,我要重新跟蹤步驟檢查。
這一步要重新到退處理檢查了,這一次不能跳過任何的彈窗和提示。
我查閱資料科普一番以后,發現T1 Coreg to Fun,就是T1結構像配準到去頭動后的fMRI 的平均圖像中,因為我的fMRI 圖像是沒有做顱骨剝離的,所以此處T 1 Coreg to Fun 輸出的應該還是帶顱骨的圖。跟我原來的理解有出入,先記錄一下。
T1ImgNewSegment 文件家中放的是分割的腦灰質、白質、腦脊液文件。

再看看預處理的后半部分,

這部分主要是4個功能:
①用來處理完成頭動矯正的fMRI 影像的,就是基于頭動還有基線去做回歸,去噪
②是標準化
也是用來處理fMRI影像的,在T1配準到fMRI 空間后,其具備了與fMRI 影像同坐標系空間特點,后 在segment 時其又配準到了標準空間,其轉換到標準空間的轉換參數也代表了fMRI 到標準空間的參數,基于此對fMRI 影像完成標準化。與此同時還做了基于體素大小的重采樣;
③空間平滑處理,還是對fMRI 影像處理,FWHM是高斯窗。如果要計算局部一致性REHo,則平滑處理應該放在計算REHO之后。
④帶通濾波,兩個是一個意思,只是因為濾波既可以先做,也可以后做。因為如果要計算ALFF 和fALFF 就要后做,選后面那個。
因為我只在意最后用來分析計算功能連接,沒有計算ALFF/fALFF和分析局部一致性,預處理部分我都是嚴格按照上面的步驟來的。
感覺計算這兩個參數的話,濾波和平滑就要選擇后面的了,不能早早選中。
理解了所有的預處理以后確實沒怎么報錯,只是這個單純的處理框架看起來很離散,每個處理步驟都有輸入和輸出還有做的目的都需要分析。

分析三天也才get 到一條初步的處理流程圖吧,一直都在邊處理邊理順邊驗證才得到的一張圖,也許這條路到最后發現有冗余,但是真的是要吃透才能簡化和完善。可以說預處理和準備分析工作占據了80%,后面的功能連接好像沒有那么多步驟。以后在具體實施中去應該吧。功能連接分析的路估計就是這么走了。真的挺長的,后面還要好好消化吸收。



)

)


:標準庫代碼示例)
與VGG16在圖像識別中的實驗設計與思路)

)
)

—— 打造直覺化交互體驗)


)


