本文歸納了常見的肌肉信號預處理流程,方便EMG信號的后續分析。使用pyemgpipeline庫 來進行信號的處理。文中使用了 UC Irvine 數據庫的下肢數據。
目錄
1 使用wrappers 定義數據類,來進行后續的操作
2 肌電信號DC偏置去除
3 帶通濾波器處理
4 對肌電信號進行全波整流
5 肌電信號線性包絡
6 幅度歸一化 (已知最大收縮的幅度)
7 分割得到一部分時間段的信號
8 得到最后處理好的數據并且保存下來
1 使用wrappers 定義數據類,來進行后續的操作
import os
import numpy as np
from matplotlib.figure import SubplotParams
import pyemgpipeline as pepdata_folder = 'uci_lower_limb/A_TXT'
data_filename = '3Asen.txt'
trial_name = 'Sit'
channel_names = ['rectus femoris', 'biceps femoris', 'vastus internus', 'semitendinosus']
sample_rate = 1000def load_uci_lower_limb_txt(_filepath):with open(_filepath) as fp:collect_values = np.array([])lines = fp.readlines()for line in lines[7:]: # first few lines are data descriptionitems = [float(e) for e in line.split('\t')[:4] if e != ''] # last column is not EMG dataif len(items) != 4: # last few rows might not have EMG datacontinuecollect_values = np.concatenate((collect_values, np.array(items)))_data = collect_values.reshape(-1, 4)return _datafilepath = os.path.join(data_folder, data_filename)
data = load_uci_lower_limb_txt(filepath)
dataprint('data shape:', data.shape)emg_plot_params = pep.plots.EMGPlotParams(n_rows=4,fig_kwargs={'figsize': (8, 6),'dpi': 80,'subplotpars': SubplotParams(wspace=0, hspace=0.6),},line2d_kwargs={'color': 'red',}
)m = pep.wrappers.EMGMeasurement(data, hz=sample_rate, trial_name=trial_name,channel_names=channel_names, emg_plot_params=emg_plot_params)m.plot()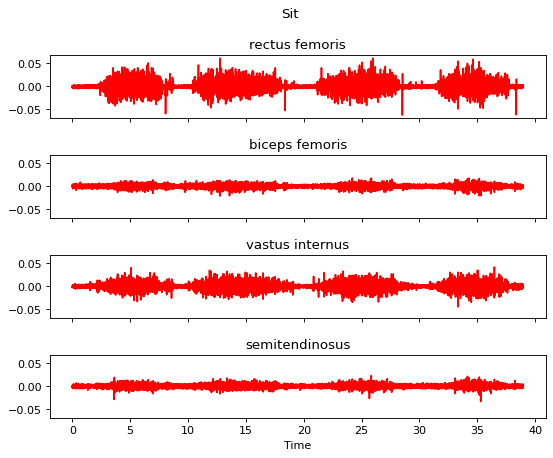
原始肌電信號
2 肌電信號DC偏置去除
m.apply_dc_offset_remover()
m.plot()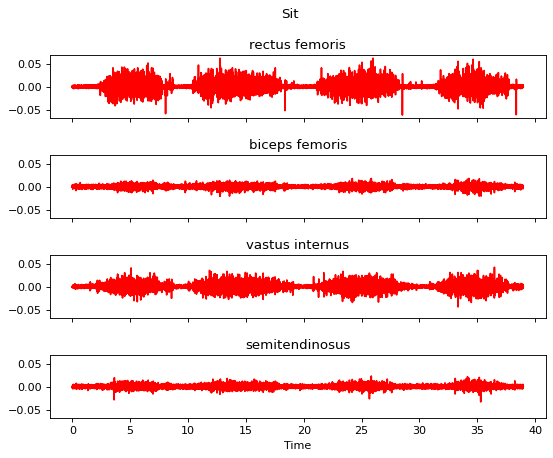
DC偏置去除的結果圖
3 帶通濾波器處理
m.apply_bandpass_filter(bf_order=4, bf_cutoff_fq_lo=10, bf_cutoff_fq_hi=450)
m.plot()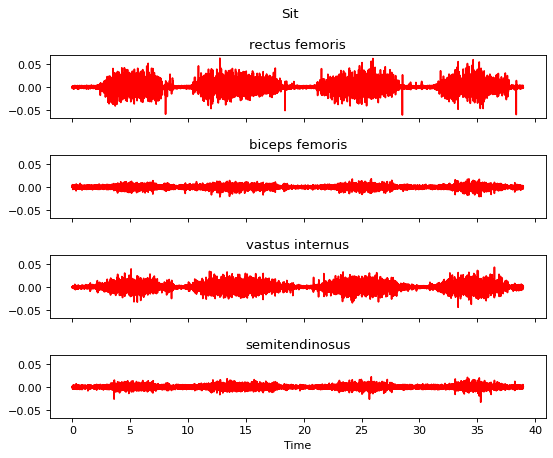
帶通濾波器處理的結果圖
4 對肌電信號進行全波整流
m.apply_full_wave_rectifier()
m.plot()
全波整流處理肌電信號
5 肌電信號線性包絡
m.apply_linear_envelope(le_order=4, le_cutoff_fq=6)
m.plot()
肌電信號線性包絡處理
6 幅度歸一化 (已知最大收縮的幅度)
max_amplitude = [0.043, 0.069, 0.364, 0.068] # assume the MVC is known
m.apply_amplitude_normalizer(max_amplitude)
m.plot()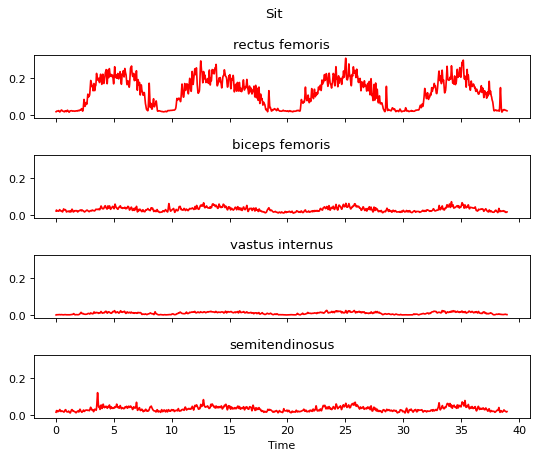
幅度歸一化結果?
?
7 分割得到一部分時間段的信號
m.apply_segmenter(20.5, 29.5)
m.plot()
分割結果
8 得到最后處理好的數據并且保存下來
m.datam.timestampm.export_csv('ex1_processed.csv')



)


![[SIGGRAPH-23] 3D Gaussian Splatting for Real-Time Radiance Field Rendering](http://pic.xiahunao.cn/[SIGGRAPH-23] 3D Gaussian Splatting for Real-Time Radiance Field Rendering)



![[ 云計算 | AWS 實踐 ] 基于 Amazon S3 協議搭建個人云存儲服務](http://pic.xiahunao.cn/[ 云計算 | AWS 實踐 ] 基于 Amazon S3 協議搭建個人云存儲服務)




圖像處理的基本操作)


