基于R 4.2.2版本演示
一、寫在前面
有不少大佬問做機器學習分類能不能用R語言,不想學Python咯。
答曰:可!用GPT或者Kimi轉一下就得了唄。
加上最近也沒啥內容寫了,就幫各位搬運一下吧。
二、R代碼實現KNN分類
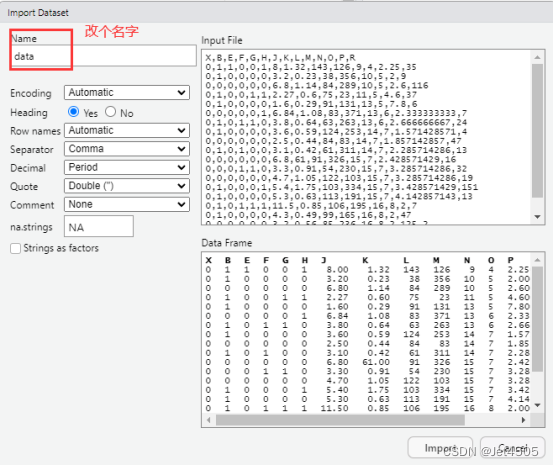
(1)導入數據
我習慣用RStudio自帶的導入功能:
(2)建立KNN模型
# Load necessary libraries
library(caret)
library(pROC)
library(ggplot2)# Assume 'data' is your dataframe containing the data
# Set seed to ensure reproducibility
set.seed(123)# Split data into training and validation sets (80% training, 20% validation)
trainIndex <- createDataPartition(data$X, p = 0.8, list = FALSE)
trainData <- data[trainIndex, ]
validData <- data[-trainIndex, ]# Convert the target variable to a factor for classification
trainData$X <- as.factor(trainData$X)
validData$X <- as.factor(validData$X)# Define control method for training with cross-validation
trainControl <- trainControl(method = "cv", number = 10)# Fit KNN model on the training set
model <- train(X ~ ., data = trainData, method = "knn", trControl = trainControl, preProcess = "scale")# Predict on the training and validation sets
trainPredict <- predict(model, trainData, type = "prob")[,2]
validPredict <- predict(model, validData, type = "prob")[,2]# Convert true values to factor for ROC analysis
trainData$X <- as.factor(trainData$X)
validData$X <- as.factor(validData$X)# Calculate ROC curves and AUC values
trainRoc <- roc(response = trainData$X, predictor = trainPredict)
validRoc <- roc(response = validData$X, predictor = validPredict)# Plot ROC curves with AUC values
ggplot(data = data.frame(fpr = trainRoc$specificities, tpr = trainRoc$sensitivities), aes(x = 1 - fpr, y = tpr)) +geom_line(color = "blue") +geom_area(alpha = 0.2, fill = "blue") +geom_abline(slope = 1, intercept = 0, linetype = "dashed", color = "black") +ggtitle("Training ROC Curve") +xlab("False Positive Rate") +ylab("True Positive Rate") +annotate("text", x = 0.5, y = 0.1, label = paste("Training AUC =", round(auc(trainRoc), 2)), hjust = 0.5, color = "blue")ggplot(data = data.frame(fpr = validRoc$specificities, tpr = validRoc$sensitivities), aes(x = 1 - fpr, y = tpr)) +geom_line(color = "red") +geom_area(alpha = 0.2, fill = "red") +geom_abline(slope = 1, intercept = 0, linetype = "dashed", color = "black") +ggtitle("Validation ROC Curve") +xlab("False Positive Rate") +ylab("True Positive Rate") +annotate("text", x = 0.5, y = 0.2, label = paste("Validation AUC =", round(auc(validRoc), 2)), hjust = 0.5, color = "red")# Calculate confusion matrices based on 0.5 cutoff for probability
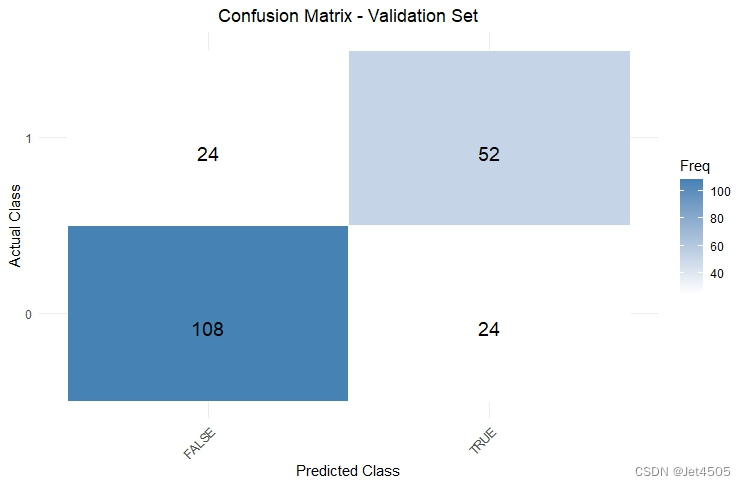
confMatTrain <- table(trainData$X, trainPredict >= 0.5)
confMatValid <- table(validData$X, validPredict >= 0.5)# Function to plot confusion matrix using ggplot2
plot_confusion_matrix <- function(conf_mat, dataset_name) {conf_mat_df <- as.data.frame(as.table(conf_mat))colnames(conf_mat_df) <- c("Actual", "Predicted", "Freq")p <- ggplot(data = conf_mat_df, aes(x = Predicted, y = Actual, fill = Freq)) +geom_tile(color = "white") +geom_text(aes(label = Freq), vjust = 1.5, color = "black", size = 5) +scale_fill_gradient(low = "white", high = "steelblue") +labs(title = paste("Confusion Matrix -", dataset_name, "Set"), x = "Predicted Class", y = "Actual Class") +theme_minimal() +theme(axis.text.x = element_text(angle = 45, hjust = 1), plot.title = element_text(hjust = 0.5))print(p)
}
# Now call the function to plot and display the confusion matrices
plot_confusion_matrix(confMatTrain, "Training")
plot_confusion_matrix(confMatValid, "Validation")# Extract values for calculations
a_train <- confMatTrain[1, 1]
b_train <- confMatTrain[1, 2]
c_train <- confMatTrain[2, 1]
d_train <- confMatTrain[2, 2]a_valid <- confMatValid[1, 1]
b_valid <- confMatValid[1, 2]
c_valid <- confMatValid[2, 1]
d_valid <- confMatValid[2, 2]# Training Set Metrics
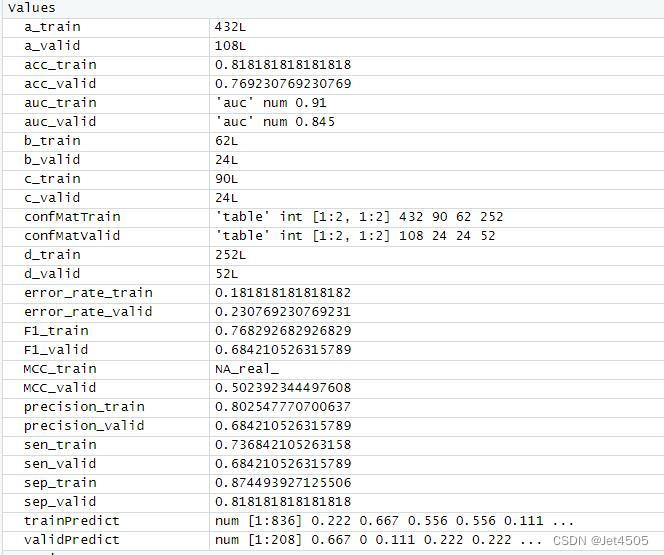
acc_train <- (a_train + d_train) / sum(confMatTrain)
error_rate_train <- 1 - acc_train
sen_train <- d_train / (d_train + c_train)
sep_train <- a_train / (a_train + b_train)
precision_train <- d_train / (b_train + d_train)
F1_train <- (2 * precision_train * sen_train) / (precision_train + sen_train)
MCC_train <- (d_train * a_train - b_train * c_train) / sqrt((d_train + b_train) * (d_train + c_train) * (a_train + b_train) * (a_train + c_train))
auc_train <- roc(response = trainData$X, predictor = trainPredict)$auc# Validation Set Metrics
acc_valid <- (a_valid + d_valid) / sum(confMatValid)
error_rate_valid <- 1 - acc_valid
sen_valid <- d_valid / (d_valid + c_valid)
sep_valid <- a_valid / (a_valid + b_valid)
precision_valid <- d_valid / (b_valid + d_valid)
F1_valid <- (2 * precision_valid * sen_valid) / (precision_valid + sen_valid)
MCC_valid <- (d_valid * a_valid - b_valid * c_valid) / sqrt((d_valid + b_valid) * (d_valid + c_valid) * (a_valid + b_valid) * (a_valid + c_valid))
auc_valid <- roc(response = validData$X, predictor = validPredict)$auc# Print Metrics
cat("Training Metrics\n")
cat("Accuracy:", acc_train, "\n")
cat("Error Rate:", error_rate_train, "\n")
cat("Sensitivity:", sen_train, "\n")
cat("Specificity:", sep_train, "\n")
cat("Precision:", precision_train, "\n")
cat("F1 Score:", F1_train, "\n")
cat("MCC:", MCC_train, "\n")
cat("AUC:", auc_train, "\n\n")cat("Validation Metrics\n")
cat("Accuracy:", acc_valid, "\n")
cat("Error Rate:", error_rate_valid, "\n")
cat("Sensitivity:", sen_valid, "\n")
cat("Specificity:", sep_valid, "\n")
cat("Precision:", precision_valid, "\n")
cat("F1 Score:", F1_valid, "\n")
cat("MCC:", MCC_valid, "\n")
cat("AUC:", auc_valid, "\n")在R語言中,caret包提供了一個通用的接口來訓練KNN模型。使用caret的train函數來訓練KNN模型時,可以調整多種參數來優化模型的性能:
基本參數:
①formula: 指定模型的公式,如Y ~ .,表示使用數據框中的所有其他變量來預測Y。
②data:?提供包含訓練數據的數據框。
③method:?對于KNN模型,這個參數應設置為"knn"。
④preProcess: 預處理步驟,常用的包括標準化("scale")和中心化("center"),對于KNN這一步非常重要因為KNN依賴于變量的距離度量。
⑤trControl: 一個trainControl對象,定義了模型訓練的各種控制策略,如交叉驗證的類型和重復次數。
trainControl 函數的參數:
①method: 訓練的方法,如交叉驗證("cv"),重復交叉驗證("repeatedcv"),留一交叉驗證("LOOCV")等。
②number: 對于"cv"和"repeatedcv",這個參數定義了折數。
③repeats: 當使用"repeatedcv"時,定義重復的次數。
④search: 參數搜索方法,默認為"grid"。也可以設置為"random"進行隨機搜索。
⑤savePredictions: 是否保存預測結果,通常用于后續分析。
模型性能調整參數:
使用KNN時,最關鍵的參數之一是鄰居的數量(K值)。這可以通過train函數的以下參數來調整:
①tuneLength: 這個參數決定了在參數搜索中考慮多少個不同的K值。
②tuneGrid: 這是一個數據框,可以自定義K值的具體范圍,例如expand.grid(k = c(1, 5, 10))
結果輸出(默認參數):



三、KNN調參方法
如前所述,KNN的關鍵參數就是K值,所以可以對其進行一個暴力測試,比如取值1到10:
# 定義交叉驗證的控制方法,啟用網格搜索
trainControl <- trainControl(method = "cv", number = 10)
# 定義K值的網格搜索范圍
tuneGrid <- expand.grid(k = 1:10)
# 在訓練集上擬合KNN模型,指定網格搜索的K值
model <- train(X ~ ., data = trainData, method = "knn", trControl = trainControl,tuneGrid = tuneGrid, preProcess = "scale")
# 查看模型結果,找出最優的K值
print(model)解讀:
①定義交叉驗證的控制方法:使用trainControl函數設定交叉驗證的詳細參數。
②定義K值的網格:使用tuneGrid參數在train函數中指定K值的范圍。
③擬合模型:使用train函數訓練模型,同時應用預處理步驟(比如標準化數據),以確保每個特征在距離計算中具有等同的權重。
結果輸出:
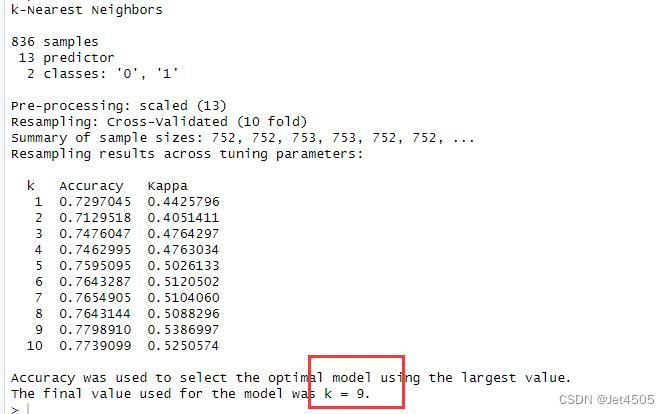
注意:用了caret包的train函數,并且通過網格搜索指定了一系列的參數(如K值的范圍),那么這個函數會自動選擇表現最好的參數配置來訓練最終的模型。train函數的輸出即是基于你提供的訓練數據和參數搜索范圍內表現最優的模型。因此,當你調用predict函數進行預測時,使用的就是這個最優化的模型。所以,下面的代碼不變。
結果吧,跟之前的完全一樣:
因為caret包對于KNN模型默認進行一系列的K值嘗試,通常這個范圍是1到最多的鄰居數,但具體的最大K值依賴于caret的內部設置。在大多數情況下,它會嘗試如1, 5, 7, 9等常用的K值。所以,我們默認參數的時候,其實軟件自動給我們尋找最優K值了。可以用這個代碼輸出最有K值:
# Print the best K value used by the model
best_k <- model$bestTune$k
cat("The best K value found is:", best_k, "\n")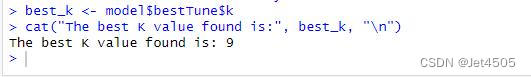
K值就是9,跟我們自行調參的一致。
那我們猛點,把K的范圍設置的寬一些:
# 定義交叉驗證的控制方法,啟用網格搜索
trainControl <- trainControl(method = "cv", number = 10)
# 定義K值的網格搜索范圍
tuneGrid <- expand.grid(k = 1:20)
# 在訓練集上擬合KNN模型,指定網格搜索的K值
model <- train(X ~ ., data = trainData, method = "knn", trControl = trainControl,tuneGrid = tuneGrid, preProcess = "scale")
# 查看模型結果,找出最優的K值
print(model)結果:
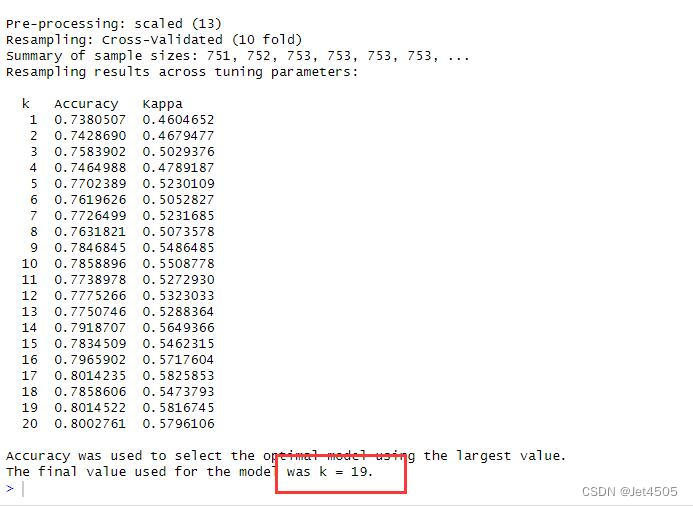
K=19,性能指標如下,似乎大同小異:
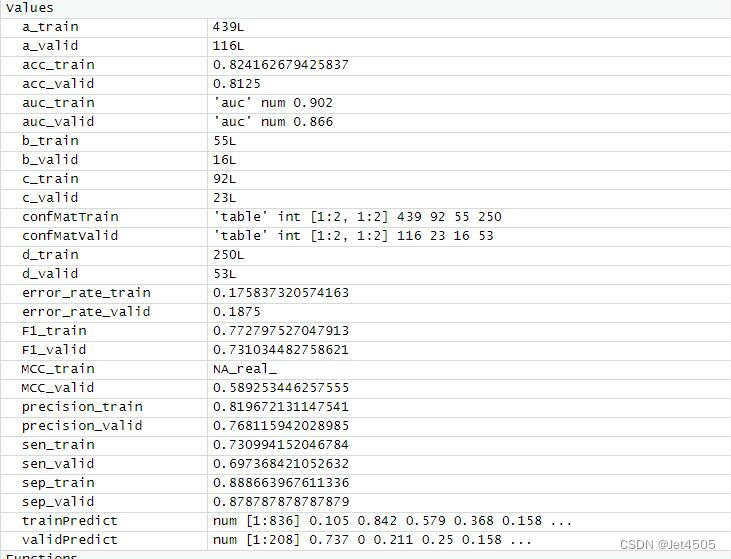
四、最后
數據嘛:
鏈接:https://pan.baidu.com/s/1rEf6JZyzA1ia5exoq5OF7g?pwd=x8xm
提取碼:x8xm










——文件保存后打開呈現亂碼問題)




(一):基本概念)
)


