本文先重點介紹了過采樣的原理是實現。 由于醫學數據相對缺乏,過采樣是解決數據問題的方法之一。? 后續寫一篇搭建神經網絡的說明
目錄
概述
導入必要的庫
數據加載和預處理函數
處理樣本不均衡函數
構建改進的 CNN 模型函數
主函數
數據生成器generator:高效助力電腦學習X光片
為啥需要這個 “服務員”?
數據生成器具體干了啥?
?Python 中對 X 光片標簽的統計操作
1. 先理解 y 是什么
2. np.sum(y == 0) 是什么意思?
3. np.sum(y == 1) 同理
總結
將 X 中的 X 光片從正方形重塑為長條形以適配處理工具
先看 X 是什么
X.reshape(...) 是在 “重塑形狀”
變成 “長條形” 后是什么樣?
為啥要這么做?
總結
隨機過采樣器
ros = RandomOverSampler(...) 是啥?
參數 random_state=42 是啥意思?
總結
復制機平衡數據
先回憶一下角色
這行代碼干了啥?
舉個生活例子
總結
使用過采樣解決訓練數據樣本不平衡問題
1. 初始化變量
2. 提取所有數據和標簽
3. 查看原始樣本分布
4. 數據展平(為過采樣做準備)
5. 過采樣少數類
6. 恢復圖像形狀
7. 查看過采樣后的分布
8. 返回處理后的數據
總結
?
import numpy as np
import matplotlib.pyplot as plt
from sklearn.model_selection import train_test_split
from sklearn.metrics import f1_score, roc_auc_score, roc_curve, confusion_matrix, classification_report
from imblearn.over_sampling import RandomOverSampler
import tensorflow as tf
from keras import layers
from keras import models
# 或者更常用的是直接導入Sequential類
from keras.models import Sequential
from keras.preprocessing.image import ImageDataGenerator
import os
import zipfile
import requests
from tensorflow.python.keras.callbacks import EarlyStopping# 數據加載和預處理
def load_data(train_dir, test_dir, val_dir, img_size=(150, 150), batch_size=32):# 數據增強器 - 僅用于訓練集train_datagen = ImageDataGenerator(rescale=1. / 255,rotation_range=10,width_shift_range=0.1,height_shift_range=0.1,shear_range=0.1,zoom_range=0.1,horizontal_flip=True)# 驗證集和測試集只需要重新縮放val_test_datagen = ImageDataGenerator(rescale=1. / 255)# 加載訓練數據train_generator = train_datagen.flow_from_directory(train_dir,target_size=img_size,batch_size=batch_size,class_mode='binary',classes=['NORMAL', 'PNEUMONIA'],shuffle=True)# 加載驗證數據val_generator = val_test_datagen.flow_from_directory(val_dir,target_size=img_size,batch_size=batch_size,class_mode='binary',classes=['NORMAL', 'PNEUMONIA'],shuffle=False)# 加載測試數據test_generator = val_test_datagen.flow_from_directory(test_dir,target_size=img_size,batch_size=batch_size,class_mode='binary',classes=['NORMAL', 'PNEUMONIA'],shuffle=False)return train_generator, val_generator, test_generator# 處理樣本不均衡(過采樣)
def handle_imbalance(generator):# 提取特征和標簽X, y = [], []num_batches = len(generator)# 重置生成器以確保從開始獲取數據generator.reset()for i in range(num_batches):batch_x, batch_y = generator.next()X.append(batch_x)y.append(batch_y)X = np.concatenate(X)y = np.concatenate(y)# 打印原始分布print(f"原始樣本分布: 正常={np.sum(y == 0)}, 肺炎={np.sum(y == 1)}")# 展平特征用于過采樣X_flat = X.reshape(X.shape[0], -1)# 過采樣少數類ros = RandomOverSampler(random_state=42)X_resampled, y_resampled = ros.fit_resample(X_flat, y)# 恢復圖像形狀X_resampled = X_resampled.reshape(-1, *X.shape[1:])print(f"過采樣后分布: 正常={np.sum(y_resampled == 0)}, 肺炎={np.sum(y_resampled == 1)}")return X_resampled, y_resampled, y# 構建改進的CNN模型
def build_model(input_shape):model = models.Sequential([# 第一個卷積塊layers.Conv2D(32, (3, 3), activation='relu', input_shape=input_shape),layers.BatchNormalization(),layers.MaxPooling2D((2, 2)),layers.Dropout(0.2),# 第二個卷積塊layers.Conv2D(64, (3, 3), activation='relu'),layers.BatchNormalization(),layers.MaxPooling2D((2, 2)),layers.Dropout(0.3),# 第三個卷積塊layers.Conv2D(128, (3, 3), activation='relu'),layers.BatchNormalization(),layers.MaxPooling2D((2, 2)),layers.Dropout(0.4),# 第四個卷積塊layers.Conv2D(256, (3, 3), activation='relu'),layers.BatchNormalization(),layers.MaxPooling2D((2, 2)),layers.Dropout(0.5),# 分類器layers.Flatten(),layers.Dense(512, activation='relu'),layers.BatchNormalization(),layers.Dropout(0.5),layers.Dense(1, activation='sigmoid')])# 使用更穩定的優化器optimizer = tf.keras.optimizers.Adam(learning_rate=0.0001)model.compile(optimizer=optimizer,loss='binary_crossentropy',metrics=['accuracy',tf.keras.metrics.Precision(name='precision'),tf.keras.metrics.Recall(name='recall'),tf.keras.metrics.AUC(name='auc')])return model# 主函數
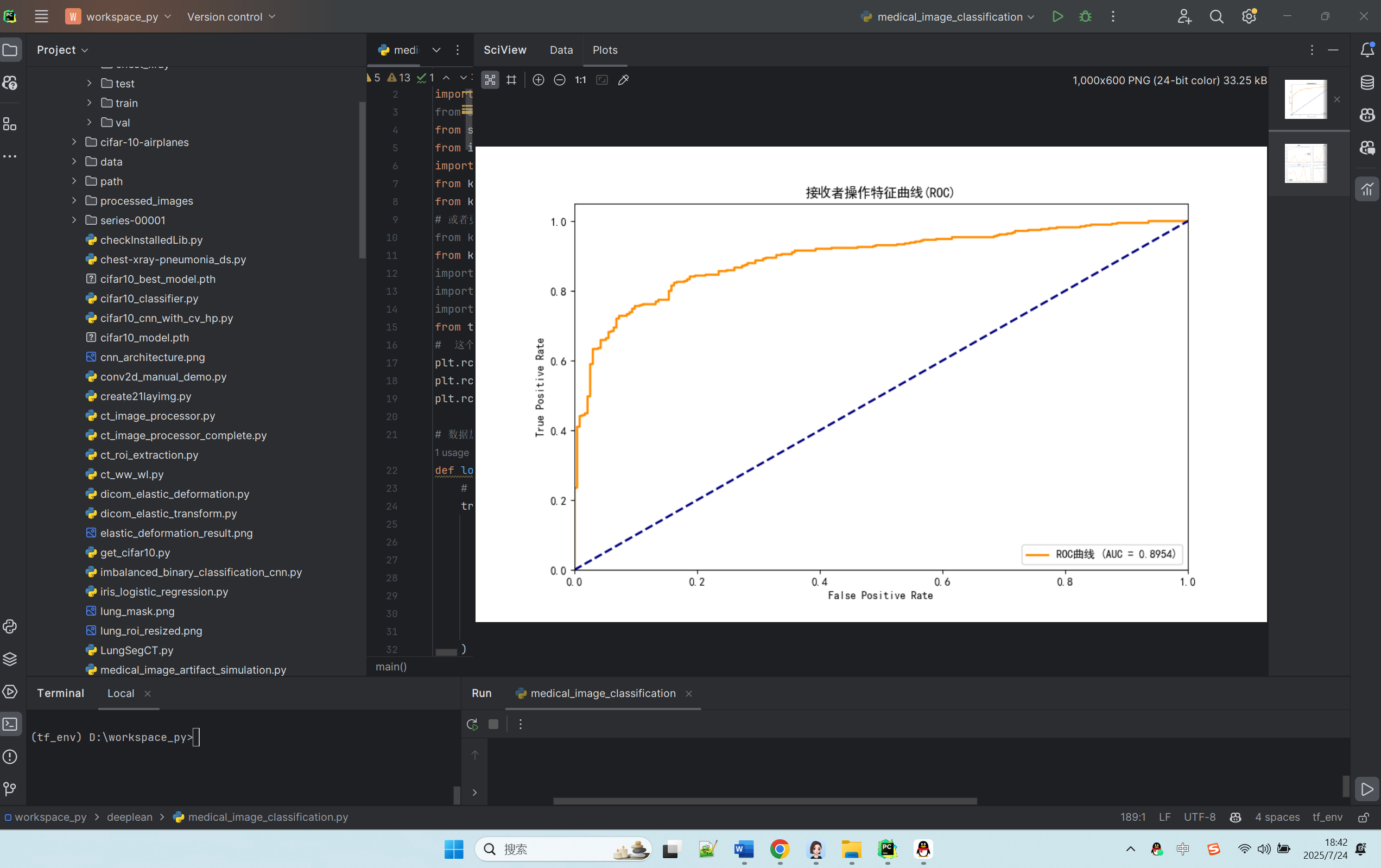
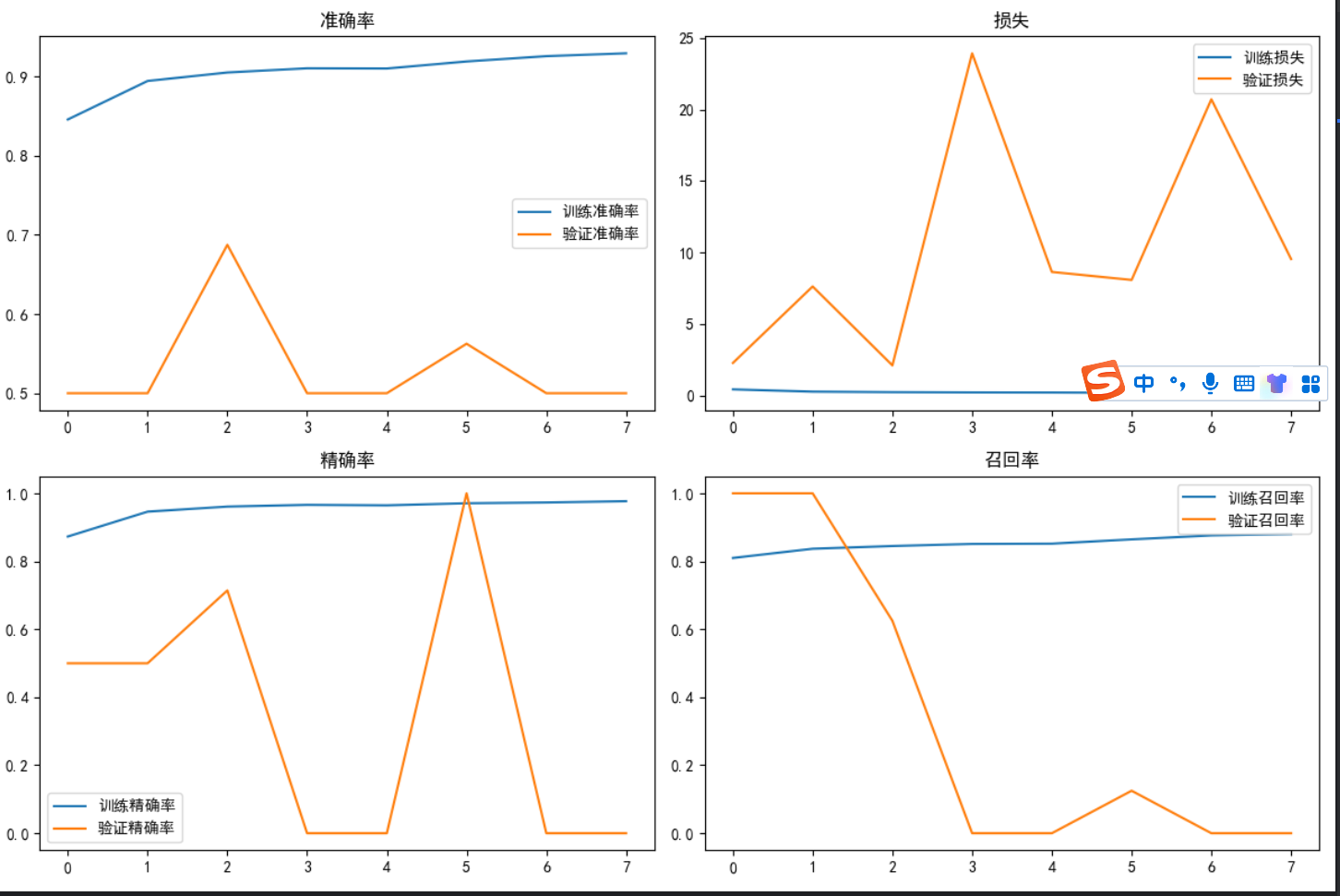
def main():# 假設數據集已經手動下載并解壓train_dir = "chest_xray/train"test_dir = "chest_xray/test"val_dir = "chest_xray/val"# 加載數據img_size = (150, 150)batch_size = 32train_generator, val_generator, test_generator = load_data(train_dir, test_dir, val_dir, img_size, batch_size)# 處理樣本不均衡X_train, y_train_resampled, y_train_original = handle_imbalance(train_generator)# 計算類別權重(基于原始分布)n_normal = np.sum(y_train_original == 0)n_pneumonia = np.sum(y_train_original == 1)total = n_normal + n_pneumoniaweight_for_normal = (1 / n_normal) * (total / 2.0)weight_for_pneumonia = (1 / n_pneumonia) * (total / 2.0)class_weights = {0: weight_for_normal, 1: weight_for_pneumonia}print(f"類別權重: 正常={weight_for_normal:.2f}, 肺炎={weight_for_pneumonia:.2f}")# 構建模型model = build_model((*img_size, 3))model.summary()# 提前停止回調early_stopping = EarlyStopping(monitor='val_loss',patience=5,restore_best_weights=True,verbose=1)# 訓練模型history = model.fit(X_train, y_train_resampled,epochs=30,batch_size=32,validation_data=val_generator,class_weight=class_weights,callbacks=[early_stopping],verbose=1)# 評估模型 - 使用完整測試集test_generator.reset()test_steps = len(test_generator)test_results = model.evaluate(test_generator, steps=test_steps, verbose=1)print("\n測試集評估結果:")print(f"準確率: {test_results[1]:.4f}")print(f"精確率: {test_results[2]:.4f}")print(f"召回率: {test_results[3]:.4f}")print(f"AUC: {test_results[4]:.4f}")# 獲取測試集所有預測結果test_generator.reset()y_true = []y_pred_prob = []for i in range(test_steps):batch_x, batch_y = test_generator.next()y_true.extend(batch_y)batch_pred = model.predict(batch_x, verbose=0).ravel()y_pred_prob.extend(batch_pred)y_true = np.array(y_true)y_pred_prob = np.array(y_pred_prob)y_pred = (y_pred_prob > 0.5).astype(int)# 計算額外指標f1 = f1_score(y_true, y_pred)auc = roc_auc_score(y_true, y_pred_prob)print(f"\nF1-score: {f1:.4f}")print(f"AUC-ROC: {auc:.4f}")# 分類報告print("\n分類報告:")print(classification_report(y_true, y_pred, target_names=['NORMAL', 'PNEUMONIA']))# 混淆矩陣cm = confusion_matrix(y_true, y_pred)print("混淆矩陣:")print(cm)# 繪制ROC曲線fpr, tpr, _ = roc_curve(y_true, y_pred_prob)plt.figure(figsize=(10, 6))plt.plot(fpr, tpr, color='darkorange', lw=2, label=f'ROC曲線 (AUC = {auc:.4f})')plt.plot([0, 1], [0, 1], color='navy', lw=2, linestyle='--')plt.xlim([0.0, 1.0])plt.ylim([0.0, 1.05])plt.xlabel('False Positive Rate')plt.ylabel('True Positive Rate')plt.title('接收者操作特征曲線(ROC)')plt.legend(loc="lower right")plt.savefig('roc_curve.png', dpi=300)plt.show()# 繪制訓練歷史plt.figure(figsize=(12, 8))plt.subplot(2, 2, 1)plt.plot(history.history['accuracy'], label='訓練準確率')plt.plot(history.history['val_accuracy'], label='驗證準確率')plt.title('準確率')plt.legend()plt.subplot(2, 2, 2)plt.plot(history.history['loss'], label='訓練損失')plt.plot(history.history['val_loss'], label='驗證損失')plt.title('損失')plt.legend()plt.subplot(2, 2, 3)plt.plot(history.history['precision'], label='訓練精確率')plt.plot(history.history['val_precision'], label='驗證精確率')plt.title('精確率')plt.legend()plt.subplot(2, 2, 4)plt.plot(history.history['recall'], label='訓練召回率')plt.plot(history.history['val_recall'], label='驗證召回率')plt.title('召回率')plt.legend()plt.tight_layout()plt.savefig('training_history.png', dpi=300)plt.show()if __name__ == "__main__":main()?運行情況:
D:\ProgramData\anaconda3\envs\tf_env\python.exe D:\workspace_py\deeplean\medical_image_classification.py
Found 5216 images belonging to 2 classes.
Found 16 images belonging to 2 classes.
Found 624 images belonging to 2 classes.
原始樣本分布: 正常=1341, 肺炎=3875
過采樣后分布: 正常=3875, 肺炎=3875
類別權重: 正常=1.94, 肺炎=0.67
2025-07-24 09:21:36.980575: I tensorflow/core/platform/cpu_feature_guard.cc:182] This TensorFlow binary is optimized to use available CPU instructions in performance-critical operations.
To enable the following instructions: SSE SSE2 SSE3 SSE4.1 SSE4.2 AVX AVX2 FMA, in other operations, rebuild TensorFlow with the appropriate compiler flags.
Model: "sequential"
_________________________________________________________________Layer (type) Output Shape Param #
=================================================================conv2d (Conv2D) (None, 148, 148, 32) 896 batch_normalization (Batch (None, 148, 148, 32) 128 Normalization) max_pooling2d (MaxPooling2 (None, 74, 74, 32) 0 D) dropout (Dropout) (None, 74, 74, 32) 0 conv2d_1 (Conv2D) (None, 72, 72, 64) 18496 batch_normalization_1 (Bat (None, 72, 72, 64) 256 chNormalization) max_pooling2d_1 (MaxPoolin (None, 36, 36, 64) 0 g2D) dropout_1 (Dropout) (None, 36, 36, 64) 0 conv2d_2 (Conv2D) (None, 34, 34, 128) 73856 batch_normalization_2 (Bat (None, 34, 34, 128) 512 chNormalization) max_pooling2d_2 (MaxPoolin (None, 17, 17, 128) 0 g2D) dropout_2 (Dropout) (None, 17, 17, 128) 0 conv2d_3 (Conv2D) (None, 15, 15, 256) 295168 batch_normalization_3 (Bat (None, 15, 15, 256) 1024 chNormalization) max_pooling2d_3 (MaxPoolin (None, 7, 7, 256) 0 g2D) dropout_3 (Dropout) (None, 7, 7, 256) 0 flatten (Flatten) (None, 12544) 0 dense (Dense) (None, 512) 6423040 batch_normalization_4 (Bat (None, 512) 2048 chNormalization) dropout_4 (Dropout) (None, 512) 0 dense_1 (Dense) (None, 1) 513 =================================================================
Total params: 6815937 (26.00 MB)
Trainable params: 6813953 (25.99 MB)
Non-trainable params: 1984 (7.75 KB)
_________________________________________________________________
Epoch 1/30
243/243 [==============================] - 134s 541ms/step - loss: 0.4104 - accuracy: 0.8552 - precision: 0.8934 - recall: 0.8067 - auc: 0.9327 - val_loss: 8.2165 - val_accuracy: 0.5000 - val_precision: 0.5000 - val_recall: 1.0000 - val_auc: 0.5000
Epoch 2/30
243/243 [==============================] - 132s 545ms/step - loss: 0.2674 - accuracy: 0.8968 - precision: 0.9505 - recall: 0.8372 - auc: 0.9627 - val_loss: 9.0454 - val_accuracy: 0.5000 - val_precision: 0.5000 - val_recall: 1.0000 - val_auc: 0.5000
Epoch 3/30
243/243 [==============================] - 144s 594ms/step - loss: 0.2277 - accuracy: 0.9112 - precision: 0.9674 - recall: 0.8511 - auc: 0.9713 - val_loss: 2.9472 - val_accuracy: 0.6250 - val_precision: 0.5833 - val_recall: 0.8750 - val_auc: 0.6719
Epoch 4/30
243/243 [==============================] - 133s 545ms/step - loss: 0.2208 - accuracy: 0.9165 - precision: 0.9676 - recall: 0.8619 - auc: 0.9735 - val_loss: 1.9898 - val_accuracy: 0.6250 - val_precision: 0.5833 - val_recall: 0.8750 - val_auc: 0.7188
Epoch 5/30
243/243 [==============================] - 128s 525ms/step - loss: 0.2024 - accuracy: 0.9228 - precision: 0.9688 - recall: 0.8738 - auc: 0.9754 - val_loss: 2.0641 - val_accuracy: 0.6875 - val_precision: 0.6154 - val_recall: 1.0000 - val_auc: 0.6875
Epoch 6/30
243/243 [==============================] - 133s 548ms/step - loss: 0.1924 - accuracy: 0.9227 - precision: 0.9691 - recall: 0.8733 - auc: 0.9783 - val_loss: 12.3177 - val_accuracy: 0.5000 - val_precision: 0.0000e+00 - val_recall: 0.0000e+00 - val_auc: 0.3984
Epoch 7/30
243/243 [==============================] - 128s 528ms/step - loss: 0.1849 - accuracy: 0.9303 - precision: 0.9728 - recall: 0.8854 - auc: 0.9796 - val_loss: 3.6844 - val_accuracy: 0.6250 - val_precision: 0.5714 - val_recall: 1.0000 - val_auc: 0.7500
Epoch 8/30
243/243 [==============================] - 129s 531ms/step - loss: 0.1561 - accuracy: 0.9334 - precision: 0.9784 - recall: 0.8865 - auc: 0.9849 - val_loss: 2.7532 - val_accuracy: 0.5000 - val_precision: 0.0000e+00 - val_recall: 0.0000e+00 - val_auc: 0.3438
Epoch 9/30
243/243 [==============================] - 128s 529ms/step - loss: 0.1646 - accuracy: 0.9356 - precision: 0.9739 - recall: 0.8952 - auc: 0.9839 - val_loss: 1.7896 - val_accuracy: 0.6875 - val_precision: 0.6364 - val_recall: 0.8750 - val_auc: 0.7031
Epoch 10/30
243/243 [==============================] - 125s 515ms/step - loss: 0.1542 - accuracy: 0.9385 - precision: 0.9778 - recall: 0.8973 - auc: 0.9847 - val_loss: 2.2632 - val_accuracy: 0.6250 - val_precision: 1.0000 - val_recall: 0.2500 - val_auc: 0.6797
Epoch 11/30
243/243 [==============================] - 130s 534ms/step - loss: 0.1450 - accuracy: 0.9432 - precision: 0.9810 - recall: 0.9040 - auc: 0.9862 - val_loss: 5.9280 - val_accuracy: 0.5000 - val_precision: 0.0000e+00 - val_recall: 0.0000e+00 - val_auc: 0.3906
Epoch 12/30
243/243 [==============================] - 129s 532ms/step - loss: 0.1435 - accuracy: 0.9422 - precision: 0.9793 - recall: 0.9035 - auc: 0.9866 - val_loss: 2.1806 - val_accuracy: 0.5000 - val_precision: 0.0000e+00 - val_recall: 0.0000e+00 - val_auc: 0.6562
Epoch 13/30
243/243 [==============================] - 132s 543ms/step - loss: 0.1348 - accuracy: 0.9475 - precision: 0.9793 - recall: 0.9143 - auc: 0.9876 - val_loss: 0.9206 - val_accuracy: 0.7500 - val_precision: 0.8333 - val_recall: 0.6250 - val_auc: 0.8203
Epoch 14/30
243/243 [==============================] - 133s 547ms/step - loss: 0.1261 - accuracy: 0.9520 - precision: 0.9853 - recall: 0.9177 - auc: 0.9893 - val_loss: 0.4002 - val_accuracy: 0.8125 - val_precision: 0.7778 - val_recall: 0.8750 - val_auc: 0.9062
Epoch 15/30
243/243 [==============================] - 129s 532ms/step - loss: 0.1259 - accuracy: 0.9507 - precision: 0.9821 - recall: 0.9182 - auc: 0.9890 - val_loss: 0.6035 - val_accuracy: 0.6875 - val_precision: 0.7143 - val_recall: 0.6250 - val_auc: 0.8281
Epoch 16/30
243/243 [==============================] - 128s 527ms/step - loss: 0.1224 - accuracy: 0.9525 - precision: 0.9851 - recall: 0.9190 - auc: 0.9896 - val_loss: 1.0697 - val_accuracy: 0.6875 - val_precision: 1.0000 - val_recall: 0.3750 - val_auc: 0.8750
Epoch 17/30
243/243 [==============================] - 124s 509ms/step - loss: 0.1145 - accuracy: 0.9556 - precision: 0.9838 - recall: 0.9265 - auc: 0.9907 - val_loss: 0.3882 - val_accuracy: 0.8125 - val_precision: 0.7778 - val_recall: 0.8750 - val_auc: 0.9062
Epoch 18/30
243/243 [==============================] - 122s 503ms/step - loss: 0.1106 - accuracy: 0.9583 - precision: 0.9863 - recall: 0.9295 - auc: 0.9911 - val_loss: 1.0384 - val_accuracy: 0.6250 - val_precision: 1.0000 - val_recall: 0.2500 - val_auc: 0.8594
Epoch 19/30
243/243 [==============================] - 124s 508ms/step - loss: 0.1084 - accuracy: 0.9561 - precision: 0.9852 - recall: 0.9262 - auc: 0.9923 - val_loss: 2.7370 - val_accuracy: 0.5000 - val_precision: 0.0000e+00 - val_recall: 0.0000e+00 - val_auc: 0.8125
Epoch 20/30
243/243 [==============================] - 124s 508ms/step - loss: 0.1044 - accuracy: 0.9574 - precision: 0.9863 - recall: 0.9277 - auc: 0.9920 - val_loss: 1.6090 - val_accuracy: 0.6250 - val_precision: 0.5714 - val_recall: 1.0000 - val_auc: 0.8828
Epoch 21/30
243/243 [==============================] - 123s 508ms/step - loss: 0.0952 - accuracy: 0.9634 - precision: 0.9875 - recall: 0.9386 - auc: 0.9932 - val_loss: 2.9180 - val_accuracy: 0.5625 - val_precision: 0.5333 - val_recall: 1.0000 - val_auc: 0.6875
Epoch 22/30
243/243 [==============================] - ETA: 0s - loss: 0.0913 - accuracy: 0.9635 - precision: 0.9891 - recall: 0.9373 - auc: 0.9937Restoring model weights from the end of the best epoch.
243/243 [==============================] - 124s 508ms/step - loss: 0.0913 - accuracy: 0.9635 - precision: 0.9891 - recall: 0.9373 - auc: 0.9937 - val_loss: 1.3448 - val_accuracy: 0.5625 - val_precision: 1.0000 - val_recall: 0.1250 - val_auc: 0.8281
Epoch 00022: early stopping
20/20 [==============================] - 5s 221ms/step - loss: 0.2983 - accuracy: 0.8990 - precision: 0.9247 - recall: 0.9128 - auc: 0.9554測試集評估結果:
準確率: 0.8990
精確率: 0.9247
召回率: 0.9128
AUC: 0.9554F1-score: 0.9187
AUC-ROC: 0.9568分類報告:precision recall f1-score supportNORMAL 0.86 0.88 0.87 234PNEUMONIA 0.92 0.91 0.92 390accuracy 0.90 624macro avg 0.89 0.89 0.89 624
weighted avg 0.90 0.90 0.90 624混淆矩陣:
[[205 29][ 34 356]]概述
這段代碼是一個用于胸部 X 光圖像肺炎分類的深度學習項目。
導入必要的庫
| import numpy as np import matplotlib.pyplot as plt from sklearn.model_selection import train_test_split from sklearn.metrics import f1_score, roc_auc_score, roc_curve, confusion_matrix, classification_report from imblearn.over_sampling import RandomOverSampler import tensorflow as tf from keras import layers from keras import models from keras.models import Sequential from keras.preprocessing.image import ImageDataGenerator import os import zipfile import requests from tensorflow.python.keras.callbacks import EarlyStopping |
- numpy 和 matplotlib.pyplot:用于數值計算和數據可視化。
- sklearn 相關模塊:用于數據劃分、模型評估等機器學習任務。
- imblearn 模塊:用于處理不平衡數據集的過采樣。
- tensorflow 和 keras 相關模塊:用于構建和訓練深度學習模型。
- os、zipfile、requests:用于文件操作和網絡請求
可以這樣理解:
- numpy和matplotlib:計算器和畫圖板,用來算數據、畫結果圖。
- sklearn:機器學習小助手,幫著評估電腦學得好不好。
- tensorflow和keras:深度學習的 “大腦”,負責讓電腦學會識別圖片。
- 其他工具:幫著讀圖片、處理文件的小幫手。
數據加載和預處理函數
| def load_data(train_dir, test_dir, val_dir, img_size=(150, 150), batch_size=32): ??? train_datagen = ImageDataGenerator( ??????? rescale=1. / 255, ??????? rotation_range=10, ??????? width_shift_range=0.1, ??????? height_shift_range=0.1, ??????? shear_range=0.1, ??????? zoom_range=0.1, ??????? horizontal_flip=True ??? ) ??? val_test_datagen = ImageDataGenerator(rescale=1. / 255) ??? train_generator = train_datagen.flow_from_directory( ??????? train_dir, ??????? target_size=img_size, ??????? batch_size=batch_size, ??????? class_mode='binary', ??????? classes=['NORMAL', 'PNEUMONIA'], ??????? shuffle=True ??? ) ??? val_generator = val_test_datagen.flow_from_directory( ??????? val_dir, ??????? target_size=img_size, ??????? batch_size=batch_size, ??????? class_mode='binary', ??????? classes=['NORMAL', 'PNEUMONIA'], ??????? shuffle=False ??? ) ??? test_generator = val_test_datagen.flow_from_directory( ??????? test_dir, ??????? target_size=img_size, ??????? batch_size=batch_size, ??????? class_mode='binary', ??????? classes=['NORMAL', 'PNEUMONIA'], ??????? shuffle=False ??? ) ??? return train_generator, val_generator, test_generator |
- 定義了一個 load_data 函數,用于加載和預處理胸部 X 光圖像數據。
- 使用 ImageDataGenerator 進行數據增強,包括旋轉、平移、縮放等操作,以增加訓練數據的多樣性。
- 分別為訓練集、驗證集和測試集創建數據生成器,從指定目錄加載圖像數據,并將圖像大小調整為 img_size,批量大小為 batch_size,類別模式為二分類('NORMAL' 和 'PNEUMONIA')。
可以這樣說:
- 數據分三類:train(訓練用,讓電腦學的)、test(考試用,最后打分的)、val(練習用,學的時候隨時糾錯的)。
- 給圖片做 “預處理”:比如把圖片統一改成 150x150 大小(方便電腦處理),訓練時還會故意旋轉、縮放圖片(讓電腦見多識廣,別認死理)。
- 告訴電腦:“NORMAL” 是正常,“PNEUMONIA” 是肺炎,這是兩類。
處理樣本不均衡函數
現實中,肺炎的片子可能比正常的多很多(比如 100 張肺炎 vs 20 張正常),電腦會學偏。這個函數就是 “找平”:
- 把少的那類(比如正常片)復制一些,讓兩類數量差不多(比如都變成 100 張)。
這樣電腦學的時候,不會因為某類片子多看就偏心。
| def handle_imbalance(generator): ??? X, y = [], [] ??? num_batches = len(generator) ??? generator.reset() ??? for i in range(num_batches): ??????? batch_x, batch_y = generator.next() ??????? X.append(batch_x) ??????? y.append(batch_y) ??? X = np.concatenate(X) ??? y = np.concatenate(y) ??? print(f"原始樣本分布: 正常={np.sum(y == 0)}, 肺炎={np.sum(y == 1)}") ??? X_flat = X.reshape(X.shape[0], -1) ??? ros = RandomOverSampler(random_state=42) ??? X_resampled, y_resampled = ros.fit_resample(X_flat, y) ??? X_resampled = X_resampled.reshape(-1, *X.shape[1:]) ??? print(f"過采樣后分布: 正常={np.sum(y_resampled == 0)}, 肺炎={np.sum(y_resampled == 1)}") ??? return X_resampled, y_resampled, y |
- 定義了一個 handle_imbalance 函數,用于處理樣本不均衡問題。
- 從數據生成器中提取特征和標簽,并將其展平用于過采樣。
- 使用 RandomOverSampler 對少數類進行過采樣,以平衡數據集。
構建改進的 CNN 模型函數
| def build_model(input_shape): ??? model = models.Sequential([ ??????? layers.Conv2D(32, (3, 3), activation='relu', input_shape=input_shape), ??????? layers.BatchNormalization(), ??????? layers.MaxPooling2D((2, 2)), ??????? layers.Dropout(0.2), ??????? layers.Conv2D(64, (3, 3), activation='relu'), ??????? layers.BatchNormalization(), ??????? layers.MaxPooling2D((2, 2)), ??????? layers.Dropout(0.3), ??????? layers.Conv2D(128, (3, 3), activation='relu'), ??????? layers.BatchNormalization(), ??????? layers.MaxPooling2D((2, 2)), ??????? layers.Dropout(0.4), ??????? layers.Conv2D(256, (3, 3), activation='relu'), ??????? layers.BatchNormalization(), ??????? layers.MaxPooling2D((2, 2)), ??????? layers.Dropout(0.5), ??????? layers.Flatten(), ??????? layers.Dense(512, activation='relu'), ??????? layers.BatchNormalization(), ??????? layers.Dropout(0.5), ??????? layers.Dense(1, activation='sigmoid') ??? ]) ??? optimizer = tf.keras.optimizers.Adam(learning_rate=0.0001) ??? model.compile( ??????? optimizer=optimizer, ??????? loss='binary_crossentropy', ??????? metrics=[ ??????????? 'accuracy', ??????????? tf.keras.metrics.Precision(name='precision'), ??????????? tf.keras.metrics.Recall(name='recall'), ??????????? tf.keras.metrics.AUC(name='auc') ??????? ] ??? ) ??? return model |
- 定義了一個 build_model 函數,用于構建一個改進的卷積神經網絡(CNN)模型。
- 模型包含多個卷積塊,每個卷積塊由卷積層、批量歸一化層、最大池化層和 dropout 層組成。
- 最后是全連接層和輸出層,使用 sigmoid 激活函數進行二分類。
- 模型使用 Adam 優化器,學習率為 0.0001,損失函數為 binary_crossentropy,評估指標包括準確率、精確率、召回率和 AUC。
主函數
| def main(): ??? train_dir = "chest_xray/train" ??? test_dir = "chest_xray/test" ??? val_dir = "chest_xray/val" ??? img_size = (150, 150) ??? batch_size = 32 ??? train_generator, val_generator, test_generator = load_data(train_dir, test_dir, val_dir, img_size, batch_size) ??? X_train, y_train_resampled, y_train_original = handle_imbalance(train_generator) ??? n_normal = np.sum(y_train_original == 0) ??? n_pneumonia = np.sum(y_train_original == 1) ??? total = n_normal + n_pneumonia ??? weight_for_normal = (1 / n_normal) * (total / 2.0) ??? weight_for_pneumonia = (1 / n_pneumonia) * (total / 2.0) ??? class_weights = {0: weight_for_normal, 1: weight_for_pneumonia} ??? print(f"類別權重: 正常={weight_for_normal:.2f}, 肺炎={weight_for_pneumonia:.2f}") ??? model = build_model((*img_size, 3)) ??? model.summary() ??? early_stopping = EarlyStopping( ??????? monitor='val_loss', ??????? patience=5, ??????? restore_best_weights=True, ??????? verbose=1 ??? ) ??? history = model.fit( ??????? X_train, y_train_resampled, ??????? epochs=30, ??????? batch_size=32, ??????? validation_data=val_generator, ??????? class_weight=class_weights, ??????? callbacks=[early_stopping], ??????? verbose=1 ??? ) ??? test_generator.reset() ??? test_steps = len(test_generator) ??? test_results = model.evaluate(test_generator, steps=test_steps, verbose=1) ??? print("\n測試集評估結果:") ??? print(f"準確率: {test_results[1]:.4f}") ??? print(f"精確率: {test_results[2]:.4f}") ??? print(f"召回率: {test_results[3]:.4f}") ??? print(f"AUC: {test_results[4]:.4f}") ??? test_generator.reset() ??? y_true = [] ??? y_pred_prob = [] ??? for i in range(test_steps): ??????? batch_x, batch_y = test_generator.next() ??????? y_true.extend(batch_y) ??????? batch_pred = model.predict(batch_x, verbose=0).ravel() ??????? y_pred_prob.extend(batch_pred) ??? y_true = np.array(y_true) ??? y_pred_prob = np.array(y_pred_prob) ??? y_pred = (y_pred_prob > 0.5).astype(int) ??? f1 = f1_score(y_true, y_pred) ??? auc = roc_auc_score(y_true, y_pred_prob) ??? print(f"\nF1-score: {f1:.4f}") ??? print(f"AUC-ROC: {auc:.4f}") ??? print("\n分類報告:") ??? print(classification_report(y_true, y_pred, target_names=['NORMAL', 'PNEUMONIA'])) ??? cm = confusion_matrix(y_true, y_pred) ??? print("混淆矩陣:") ??? print(cm) ??? fpr, tpr, _ = roc_curve(y_true, y_pred_prob) ??? plt.figure(figsize=(10, 6)) ??? plt.plot(fpr, tpr, color='darkorange', lw=2, label=f'ROC曲線 (AUC = {auc:.4f})') ??? plt.plot([0, 1], [0, 1], color='navy', lw=2, linestyle='--') ??? plt.xlim([0.0, 1.0]) ??? plt.ylim([0.0, 1.05]) ??? plt.xlabel('False Positive Rate') ??? plt.ylabel('True Positive Rate') ??? plt.title('接收者操作特征曲線(ROC)') ??? plt.legend(loc="lower right") ??? plt.savefig('roc_curve.png', dpi=300) ??? plt.show() ??? plt.figure(figsize=(12, 8)) ??? plt.subplot(2, 2, 1) ??? plt.plot(history.history['accuracy'], label='訓練準確率') ??? plt.plot(history.history['val_accuracy'], label='驗證準確率') ??? plt.title('準確率') ??? plt.legend() ??? plt.subplot(2, 2, 2) ??? plt.plot(history.history['loss'], label='訓練損失') ??? plt.plot(history.history['val_loss'], label='驗證損失') ??? plt.title('損失') ??? plt.legend() ??? plt.subplot(2, 2, 3) ??? plt.plot(history.history['precision'], label='訓練精確率') ??? plt.plot(history.history['val_precision'], label='驗證精確率') ??? plt.title('精確率') ??? plt.legend() ??? plt.subplot(2, 2, 4) ??? plt.plot(history.history['recall'], label='訓練召回率') ??? plt.plot(history.history['val_recall'], label='驗證召回率') ??? plt.title('召回率') ??? plt.legend() ??? plt.tight_layout() ??? plt.savefig('training_history.png', dpi=300) ??? plt.show() if __name__ == "__main__": ??? main() |
- 定義了一個 main 函數,作為程序的入口。
- 指定了訓練集、測試集和驗證集的目錄。
- 調用 load_data 函數加載數據,調用 handle_imbalance 函數處理樣本不均衡問題。
- 計算類別權重,用于處理樣本不均衡問題。
- 調用 build_model 函數構建模型,并打印模型摘要。
- 使用 EarlyStopping 回調函數,在驗證損失不再下降時停止訓練,并恢復最佳權重。
- 訓練模型,評估模型在測試集上的性能,并計算各種評估指標。
- 繪制 ROC 曲線和訓練歷史圖表,以可視化模型的性能。
總的來說,這段代碼實現了一個完整的胸部 X 光圖像肺炎分類系統,包括數據加載、預處理、模型構建、訓練和評估等步驟。
數據生成器generator:高效助力電腦學習X光片
數據生成器(generator)你可以理解成一個 “自動上菜的服務員”,專門給電腦 “喂” 數據的。
為啥需要這個 “服務員”?
如果你的電腦要學 10000 張 X 光片,這些片子加起來可能有幾個 G 大。如果一下子全塞進電腦內存(相當于 “一口氣把所有菜都端上桌”),內存可能裝不下,電腦會變慢甚至卡死。
這時候就需要 “數據生成器” 這個服務員:它不一次性把所有片子都拿出來,而是一批一批地給(比如一次給 32 張),電腦學完這 32 張,再給下 32 張,循環往復,直到學完所有。
數據生成器具體干了啥?
- 按批次取數據:比如你設定 “一批 32 張”,它就每次從文件夾里挑 32 張 X 光片。
- 順便做預處理:拿片子的時候,自動把它們改成統一大小(比如 150x150),或者旋轉、縮放一下(增加數據多樣性,讓電腦學得更靈活)。
- 給片子貼標簽:每張片子對應的 “正常” 或 “肺炎” 標簽,它也會一起拿給電腦,不用你手動對應。
def load_data(train_dir, test_dir, val_dir, img_size=(150, 150), batch_size=32)
def load_data(train_dir, test_dir, val_dir, img_size=(150, 150), batch_size=32):# 數據增強器 - 僅用于訓練集train_datagen = ImageDataGenerator(rescale=1. / 255,rotation_range=10,width_shift_range=0.1,height_shift_range=0.1,shear_range=0.1,zoom_range=0.1,horizontal_flip=True)# 驗證集和測試集只需要重新縮放val_test_datagen = ImageDataGenerator(rescale=1. / 255)# 加載訓練數據train_generator = train_datagen.flow_from_directory(train_dir,target_size=img_size,batch_size=batch_size,class_mode='binary',classes=['NORMAL', 'PNEUMONIA'],shuffle=True)# 加載驗證數據val_generator = val_test_datagen.flow_from_directory(val_dir,target_size=img_size,batch_size=batch_size,class_mode='binary',classes=['NORMAL', 'PNEUMONIA'],shuffle=False)# 加載測試數據test_generator = val_test_datagen.flow_from_directory(test_dir,target_size=img_size,batch_size=batch_size,class_mode='binary',classes=['NORMAL', 'PNEUMONIA'],shuffle=False)return train_generator, val_generator, test_generator在 load_data 函數里,我們創建了 train_generator(訓練用的服務員)、val_generator(驗證用的服務員):
| train_generator = train_datagen.flow_from_directory(...) |
這里的 flow_from_directory 就是 “從文件夾里取數據” 的意思,這個函數會生成一個 “服務員”,你調用它的 .next() 方法,它就給你一批處理好的片子和標簽:
| batch_x, batch_y = generator.next()? # 服務員,來一批32張片子和標簽~ |
數據生成器就像:
- 一個 “自助餐服務員”,每次給你端一小盤菜(一批數據),吃完再端,不浪費空間。
- 一個 “預處理小助手”,端菜前還會幫你把菜切好、擺盤(統一尺寸、增強數據)。
有了它,電腦就能高效地 “吃” 數據、學知識,不會因為數據太多噎著(內存不足)。
?Python 中對 X 光片標簽的統計操作
print(f"原始樣本分布: 正常={np.sum(y == 0)}, 肺炎={np.sum(y == 1)}")
這里的 y 不是集合,而是一個 “標簽列表”(更準確地說,是 NumPy 數組),里面裝的全是 0 和 1:0 代表 “正常” 的 X 光片,1 代表 “肺炎” 的 X 光片。
:
1. 先理解 y 是什么
假設我們有 10 張 X 光片,對應的標簽 y 可能是這樣的:
y = [0, 1, 1, 0, 1, 1, 1, 0, 1, 1]
(意思是:第 1、4、8 張是正常片,其余 7 張是肺炎片)
2. np.sum(y == 0) 是什么意思?
- y == 0:先檢查 y 里的每個元素是不是 0,會得到一個 “真假列表”。
比如上面的 y 會變成:
[True, False, False, True, False, False, False, True, False, False]
(True 表示這個位置是 0,False 表示不是)
- np.sum(...):把 “真假列表” 里的 True 算成 1,False 算成 0,然后求和。
上面的例子里,True 有 3 個,所以 np.sum(y == 0) = 3
(意思是:正常的片子有 3 張)
3. np.sum(y == 1) 同理
- y == 1:檢查每個元素是不是 1,得到另一個 “真假列表”:
[False, True, True, False, True, True, True, False, True, True]
- 求和后 np.sum(y == 1) = 7
(意思是:肺炎的片子有 7 張)
總結
這兩行代碼就是在 “數數量”:
np.sum(y == 0) → 數清楚 “正常” 的片子有多少張;
np.sum(y == 1) → 數清楚 “肺炎” 的片子有多少張。
最后用 print 顯示出來,就能直觀看到兩類片子的數量是否平衡啦~
將 X 中的 X 光片從正方形重塑為長條形以適配處理工具
這句話的作用是把所有 X 光片從 “正方形” 變成 “長條形”,方便后面的工具處理。
先看 X 是什么
X 里裝的是一堆 X 光 X 光片,假設是 3200 張,每張都是 150x150 像素的彩色圖(3 個顏色通道)。
它的形狀可以理解為:(3200, 150, 150, 3)
翻譯過來就是:3200 張圖,每張圖高 150、寬 150、3 個顏色層。
X.reshape(...) 是在 “重塑形狀”
X.reshape(X.shape[0], -1) 里的參數:
- X.shape[0]:表示 “保持樣本數量不變”(還是 3200 張)。
- -1:表示 “自動計算剩下的長度”(不用我們手動算)。
變成 “長條形” 后是什么樣?
原來每張圖是 150x150x3 的 “方塊”,展開成一條線的長度是:150×150×3 = 67500。
所以重塑后,X_flat 的形狀是 (3200, 67500):
- 3200 張圖不變;
- 每張圖從 “150x150x3 的方塊” 變成了 “67500 個數字排成的長條”。
為啥要這么做?
因為后面用來 “復制樣本” 的工具(RandomOverSampler)比較 “死板”,只認這種 “一行代表一個樣本” 的長條形數據,不認原來的 “方塊形” 圖片。
這一步就相當于把 “魔方” 拆成 “一條直線”,方便工具操作,后面用完了還會再拼回去。
總結
這行代碼就是:把所有圖片從二維的 “方塊” 展開成一維的 “長條”,目的是適配后面的處理工具,就像把衣服疊成特定形狀才能放進收納盒一樣。
隨機過采樣器
ros = RandomOverSampler(random_state=42)
這句話是在創建一個 “復制機”,專門用來復制少的那類數據,讓兩類數據數量一樣多。
ros = RandomOverSampler(...) 是啥?
- RandomOverSampler 翻譯過來是 “隨機過采樣器”,你可以理解成一個 “智能復制機”。
- 它的唯一任務:發現哪類數據少,就隨機復制少的那類,直到兩類數量一樣多(比如正常片少就復制正常片,肺炎片少就復制肺炎片)。
- ros 是給這個復制機起的 “小名”,方便后面調用它干活。
參數 random_state=42 是啥意思?
- 這個參數是給復制機設定 “復制規則”,保證每次復制的結果都一樣。
舉個例子:
如果沒有 random_state=42,復制機第一次可能復制第 1、3、5 張正常片,第二次可能復制第 2、4、6 張 —— 兩次結果不一樣,電腦學習效果也會波動。
加上 random_state=42 后,就像給復制機定了個 “固定菜譜”,每次都會按同樣的規則選要復制的片子,結果完全一樣。這樣實驗結果能重復,方便調試。
(為啥是 42?這是個常用的 “隨機種子” 數字,用其他數字比如 100、2024 也可以,只要固定就行。)
總結
- RandomOverSampler 是個 “復制機”,負責把少的樣本復制到和多的一樣多。
- random_state=42 是為了讓復制結果固定不變,保證實驗能重復。
就像你按同一個食譜做飯,每次味道都一樣,不會忽咸忽淡~
復制機平衡數據
X_resampled, y_resampled = ros.fit_resample(X_flat, y)
這句話的作用是:讓 “復制機” 開始工作,把少的那類 X 光片復制到和多的那類數量一樣多。
先回憶一下角色
- ros 是之前初始化的 “復制機”(RandomOverSampler),專門負責復制少的樣本。
- X_flat 是 “壓平成條” 的 X 光片(長條形,方便復制機操作)。
- y 是這些片子的標簽(0 = 正常,1 = 肺炎)。
這行代碼干了啥?
- 復制機先 “看” 數據:
復制機(ros)會先檢查 y 里的標簽,數數 0 和 1 各有多少。
比如發現:正常片(0)有 500 張,肺炎片(1)有 2500 張 —— 正常片太少了。
- 自動復制少的那類:
復制機只復制少的(這里是正常片),一直復制到兩類數量一樣多。
上面的例子里,會把正常片從 500 張復制到 2500 張,和肺炎片數量相同。
- 返回復制后的結果:
- X_resampled:復制后的 “長條形” X 光片(現在正常和肺炎各 2500 張,共 5000 張)。
- y_resampled:對應的標簽(也是 5000 個,0 和 1 各 2500 個)。
舉個生活例子
就像你有 5 顆草莓糖和 25 顆巧克力糖,想讓兩種糖數量一樣多:
- 復制機看到草莓糖少,就會復制 20 顆草莓糖(總共 25 顆)。
- 最后得到 25 顆草莓糖(X_resampled里的正常片)和 25 顆巧克力糖(X_resampled里的肺炎片),標簽也對應上。
總結
這行代碼就是啟動 “復制機”,自動把少的樣本復制到和多的樣本數量相同,讓兩類數據平衡,方便電腦公平學習。
使用過采樣解決訓練數據樣本不平衡問題
這個 handle_imbalance 函數的核心作用是解決訓練數據中不同類別的樣本數量不平衡問題(比如肺炎樣本遠多于正常樣本),通過 “過采樣” 讓兩類樣本數量趨于均衡,避免模型學習時偏向數量多的類別。
下面分步驟詳細解釋:
1. 初始化變量
| X, y = [], [] num_batches = len(generator) generator.reset() |
- X 用來存儲所有圖像數據,y 用來存儲對應的標簽(0 表示正常,1 表示肺炎)。
- num_batches 獲取數據生成器(generator)中的批次數量(比如每次生成 32 張圖,共 100 批,就是 3200 張圖)。
- generator.reset() 重置生成器,確保從第一批數據開始讀取,避免漏讀或重復。
2. 提取所有數據和標簽
| for i in range(num_batches): ??? batch_x, batch_y = generator.next()? # 獲取一批數據(圖像+標簽) ??? X.append(batch_x)??????????????????? # 把這批圖像加入X列表 ??? y.append(batch_y)??????????????????? # 把這批標簽加入y列表 X = np.concatenate(X)? # 把列表中的所有批次圖像合并成一個大數組 y = np.concatenate(y)? # 把列表中的所有批次標簽合并成一個大數組 |
- 數據生成器是 “分批” 提供數據的(比如一次給 32 張圖),這里通過循環把所有批次的數據合并成一個完整的數據集。
- 例如:原來分 10 批,每批 32 張圖,合并后 X 就是一個形狀為 (320, 150, 150, 3) 的數組(320 張圖,每張 150x150 像素,3 通道 RGB)。
3. 查看原始樣本分布
| print(f"原始樣本分布: 正常={np.sum(y == 0)}, 肺炎={np.sum(y == 1)}") |
- 統計并打印原始數據中 “正常”(標簽 0)和 “肺炎”(標簽 1)的樣本數量。
- 假設輸出為 原始樣本分布: 正常=1000, 肺炎=4000,說明兩類樣本比例是 1:4,不平衡問題明顯。
4. 數據展平(為過采樣做準備)
| X_flat = X.reshape(X.shape[0], -1) |
- 過采樣工具(RandomOverSampler)要求輸入的特征是 “二維數組”(樣本數 × 特征數),而圖像是四維數組(樣本數 × 高 × 寬 × 通道)。
- 這一步將每張圖像 “展平”:比如 150x150x3 的圖像會變成一個長度為 150×150×3=67500 的一維數組,最終 X_flat 形狀為 (樣本數, 67500)。
5. 過采樣少數類
| ros = RandomOverSampler(random_state=42)? # 初始化過采樣器(固定隨機種子,結果可重復) X_resampled, y_resampled = ros.fit_resample(X_flat, y)? # 對少數類進行過采樣 |
- RandomOverSampler 的作用是:復制少數類的樣本,讓兩類樣本數量相同。
- 以上面的例子(1000 正常 vs 4000 肺炎),過采樣后會變成 4000 正常 vs 4000 肺炎(通過復制 3000 個正常樣本實現)。
6. 恢復圖像形狀
| X_resampled = X_resampled.reshape(-1, *X.shape[1:]) |
- 過采樣后的 X_resampled 是展平的一維數組,需要恢復成圖像的原始形狀(高 × 寬 × 通道),方便后續輸入模型訓練。
- 例如:從 (8000, 67500) 恢復為 (8000, 150, 150, 3)。
7. 查看過采樣后的分布
| print(f"過采樣后分布: 正常={np.sum(y_resampled == 0)}, 肺炎={np.sum(y_resampled == 1)}") |
- 打印過采樣后的樣本數量,確認兩類已平衡(比如 正常=4000, 肺炎=4000)。
8. 返回處理后的數據
| return X_resampled, y_resampled, y |
- 返回 3 個結果:
- X_resampled:過采樣后的圖像數據(平衡后)。
- y_resampled:過采樣后的標簽(平衡后)。
- 原始標簽 y:用于后續計算類別權重等。
總結
這個函數的核心邏輯是:
- 從數據生成器中提取所有原始數據。
- 用過采樣方法(復制少數類)平衡兩類樣本數量。
- 恢復圖像形狀,方便模型使用。
通過這一步處理,模型在訓練時不會因為某類樣本多就 “偏愛” 它,能更公平地學習兩類特征,提高分類準確性。


是什么)









)






