這篇論文提出了ThermoSeek,一個綜合性的網絡資源,用于分析來自嗜熱和嗜冷物種的蛋白質序列和結構。具體來說,
- 數據收集:從美國國家生物技術信息中心(NCBI)的基因組數據庫中收集了物種的分類ID,并根據“溫度范圍”標記為嗜熱、超嗜熱、嗜冷或冷適應。使用MMseqs2對蛋白質序列進行聚類和冗余消除,生成一個包含130,825個超嗜熱蛋白、566,619個嗜熱蛋白、486,139個嗜冷蛋白和19,793個冷適應蛋白的綜合數據庫。
- 序列比對:使用NCBI BLAST 2.13.0+和MMseqs2創建序列數據庫,并通過“mmseqs easy-search”和“blastp”進行序列搜索。
- 結構搜索:利用Foldseek算法將蛋白質結構編碼為20個離散值,表示二級結構特征和氨基酸之間的空間關系。使用MMseqs2進行結構搜索。
- 模體搜索:使用Fpocket v2.0識別超嗜熱和嗜熱蛋白質中的口袋,并將提取的蛋白質口袋編碼為自定義的二進制格式。使用Kruskal算法構建最小生成樹(MST),以優化搜索過程。
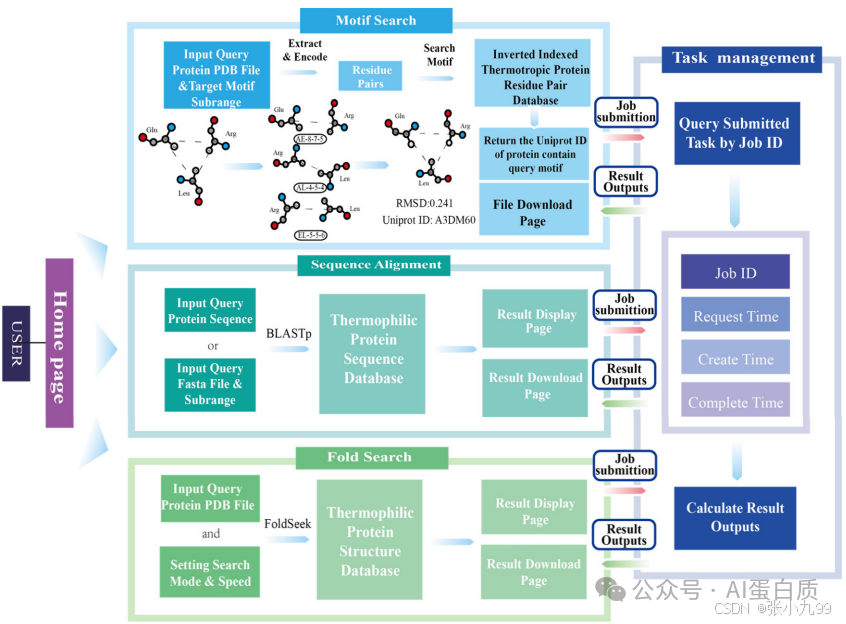
這篇論文提出了ThermoSeek,一個綜合性的網絡資源,用于分析來自嗜熱和嗜冷物種的蛋白質序列和結構。具體來說,
- 數據收集:從美國國家生物技術信息中心(NCBI)的基因組數據庫中收集了物種的分類ID,并根據“溫度范圍”標記為嗜熱、超嗜熱、嗜冷或冷適應。使用MMseqs2對蛋白質序列進行聚類和冗余消除,生成一個包含130,825個超嗜熱蛋白、566,619個嗜熱蛋白、486,139個嗜冷蛋白和19,793個冷適應蛋白的綜合數據庫。
- 序列比對:使用NCBI BLAST 2.13.0+和MMseqs2創建序列數據庫,并通過“mmseqs easy-search”和“blastp”進行序列搜索。
- 結構搜索:利用Foldseek算法將蛋白質結構編碼為20個離散值,表示二級結構特征和氨基酸之間的空間關系。使用MMseqs2進行結構搜索。
- 模體搜索:使用Fpocket v2.0識別超嗜熱和嗜熱蛋白質中的口袋,并將提取的蛋白質口袋編碼為自定義的二進制格式。使用Kruskal算法構建最小生成樹(MST),以優化搜索過程。















)

02)
之ShadowCaster)
![[MRCTF2020]Ez_bypass](http://pic.xiahunao.cn/[MRCTF2020]Ez_bypass)