在Jupyter-lab中使用RDKit畫分子2D圖
在做完分子對接后,想看看篩選后的分子的結構。因此想利用Jupyter-lab來畫分子的2D圖。
1. 安裝Jupyter-lab與RDKit
- 系統:Win11
- 已安裝conda
RDKit 是一個功能強大、靈活易用的化學信息學工具包,廣泛應用于藥物發現、化學生物學、材料科學等領域。RDKit 提供了豐富的化學信息可視化功能,用戶可以通過RDKit 生成分子結構圖、化學反應圖、藥物分子的三維結構等,方便進行結果展示和分析。
1.1 安裝RDKit
#創建并安裝RDKit的conda環境
conda create -c conda-forge -n my-rdkit-env rdkit#激活RDKit的虛擬環境
conda activate my-rdkit-env
不知道為啥,這一步很慢,不知道是不是conda channel的問題。但總歸是等了一段時間就成功了。參考自官方教程。
1.2 安裝Jupyter-lab
Jupyter Lab是Jupyter Notebook的升級版本,可以一個窗口中同時打開多個 Notebook、代碼編輯器、終端、文件瀏覽器等,更方便地進行多任務處理和工作流程管理。Jupyter Lab集成了豐富的文檔編輯器,包括 Markdown、LaTeX 等,用戶可以方便地編寫和編輯文檔,同時可以通過插件支持更多的文檔格式和擴展功能。最主要的是Jupyter Lab提供了豐富的可視化功能,用戶可以通過插件支持各種圖表庫和數據可視化工具,實現更豐富和復雜的數據分析和可視化。
使用pip安裝
pip install jupyterlab參考自官方安裝教程。
2. 小分子(SDF格式)作圖
2.1 畫單個SDF格式的分子圖
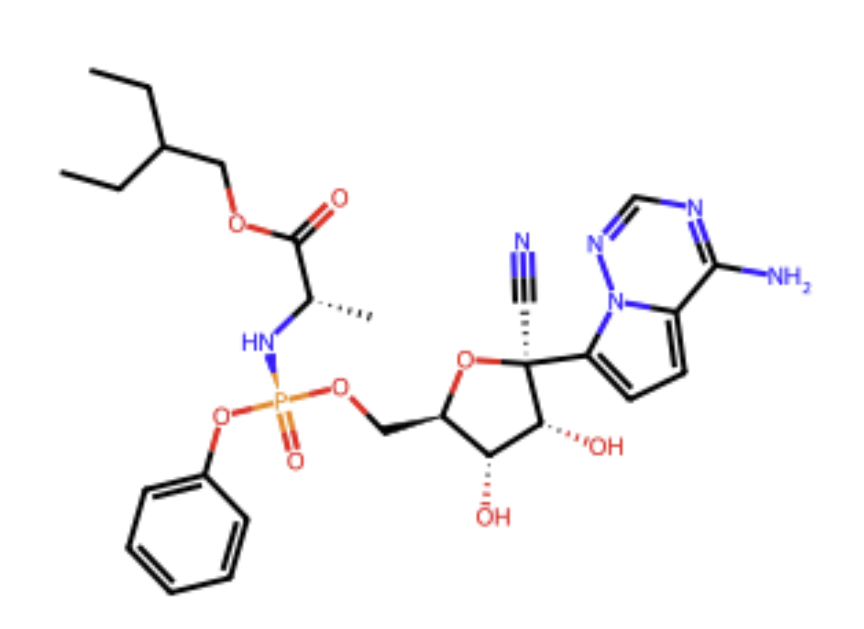
以他莫昔芬(Tamoxifen)小分子為例,首先從PubChem網站下載該分子的三維結構的SDF結構文件。
如法炮制,再下載其余三個小分子:阿司匹林(Aspirin),莫洛昔康(Molnupiravir),瑞德西韋(Remdesivir)。將四個分子保存在sdf/文件夾中。
在CMD中跳轉至cd sdf/文件夾路徑下,打開Jupyter-lab。
from rdkit import Chem
from rdkit.Chem import AllChem
from rdkit.Chem import Draw#讀取sdf文件
m1 = Chem.MolFromMolFile('sdf/Conformer3D_COMPOUND_CID_121304016.sdf')#通過調用AllChem.Compute2DCoords(m1)計算出m1分子相應的二維坐標,并將其更新到分子對象中
AllChem.Compute2DCoords(m1)#判斷m1分子是否讀取成功
m1 is None#Draw.MolToImage()函數畫出分子的2D圖像
Draw.MolToImage(m1)
運行后結果如下:
- 讀取單個的SDF文件可以用
Chem.MolFromMolFile(),盡管這個主要用于讀取MOL格式的分子文件。 - 讀取一個存有多個分子集的SDF文件,主要用
Chem.SDMolSupplier()。 Chem.MolFromMolFile()無法讀取PDB格式的分子文件,因此小分子文件讀取主推SDF格式。
2.2 畫多個SDF格式的分子圖
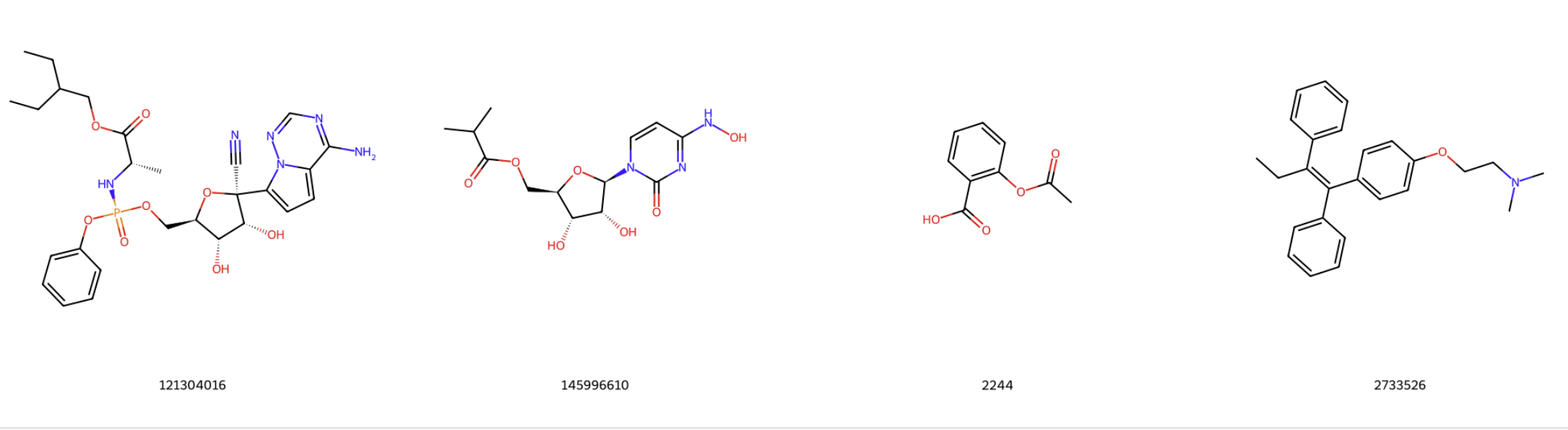
讀取sdf/文件夾下的多個SDF分子,且將分子的2D圖放在一張圖中。
import os
import glob# 指定小分子的存儲路徑
path = r'sdf/'# 使用os.path.join構建路徑,獲取所有.sdf文件的路徑列表
sdf_files = glob.glob(os.path.join(path, '*.sdf'))# 將所有.sdf文件保存在一個列表ms中
ms = []
for sdf_file in sdf_files:m = Chem.MolFromMolFile(sdf_file)ms.append(m)#計算出列表中分子的二維坐標,并將其更新到分子對象中
for m in ms: tmp=AllChem.Compute2DCoords(m)# MolsToGridImage()函數可以將4個分子畫在一張圖上
# molsPerRow=4參數設置4個小分子并排成一列
# subImgSize=(500,500)設置每個小分子的圖像尺寸為500x500大小
# legends=x.GetProp("_Name")獲取小分子的名稱作為圖例
img=Draw.MolsToGridImage(ms[:4],molsPerRow=4,subImgSize=(500,500),legends=[x.GetProp("_Name") for x in ms[:4]], returnPNG=False) # 保存分子圖像為PNG格式文件在sdf/文件夾下
img.save('./Mol_4.png')img
結果如下所示:
在Draw.MolsToGridImage函數中設置returnPNG=False參數,主要用于在保存畫出的圖片時img.save()的報錯:
AttributeError Traceback (most recent call last) Cell In[23], line 20
17 for m in ms: tmp=AllChem.Compute2DCoords(m)
19 img=Draw.MolsToGridImage(ms[:4],molsPerRow=4,subImgSize=(500,500),legends=[x.GetProp(“_Name”)
for x in ms[:4]])
—> 20 img.save(‘./Mol12_20.png’)
21 imgAttributeError: ‘Image’ object has no attribute ‘save’

-基礎tranform算子(一))














)


