Deep-joint-learning analysis model of single cell transcriptome and open chromatin accessibility data單細胞轉錄組和開放染色質可及性數據的深度聯合學習分析模型
在同一個細胞中同時分析轉錄組和染色質可及性信息為了解細胞狀態提供了前所未有的解決方案。然而,計算有效的方法,這些固有的稀疏和異構數據的整合是缺乏的。在這里,我們提出了一個單細胞多模態變分自動編碼器模型,它結合了三種類型的聯合學習策略與概率高斯混合模型,以學習準確代表這些多層配置文件的聯合潛在特征。對模擬數據集和真實的數據集的研究表明,該方法具有更好的能力:(i)在聯合學習空間中剖析細胞異質性;(ii)去噪和估算數據;(iii)構建多層組學數據之間的關聯,可用于理解轉錄調控機制。
介紹
基因表達是通過一組轉錄因子(TF)結合到其順式調控基因組區域來調節的。
scRNA-seq表征細胞的基因表達水平,而scATAC-seq等表觀基因組變化反映了附近基因中順式調控元件的開放性。這種兩組學數據的整合可以提供關于與細胞異質性相關的調控層的新見解[13]。許多集成工具都是為批量數據設計的[14]。
例如,主成分分析(PCA)的推廣MOFA被提出來處理批量數據,也可以應用于單細胞數據集[15]。IntNMF是非負矩陣因子分解(NMF)的擴展,用于整合疾病亞型分類的多組學數據,并評估其處理單細胞數據集[16,17]。然而,最近的研究發現,單細胞數據有其獨特的特點,不同于批量數據,因此需要開發新的方法[18]。單細胞多分析(PCA)的集成被提出來處理批量數據,也可以應用于單細胞數據集[15]。IntNMF是非負矩陣因子分解(NMF)的擴展,用于整合疾病亞型分類的多組學數據,并評估其處理單細胞數據集[16,17]。然而,最近的研究發現,單細胞數據有其獨特的特點,不同于批量數據,因此需要開發新的方法[18]。
單細胞多組學數據的整合仍然是一個巨大的挑戰,這是由于固有的高度稀疏性,由于測定噪聲導致的巨大異質性,scATAC-seq和scRNA-seq數據之間的巨大維度差異,大約10-20倍[19],以及越來越大規模的數據集[20]。已經開發了大量用于scRNA-seq數據整合的方法,然而,只有少數方法被提出用于整合單細胞多組學數據,并且這些方法是針對從不同細胞收集但從相同細胞群體提取的組學數據開發的[21-24]。例如,提出了耦合MMF,通過構建基因和順式調控元件的耦合非負矩陣來對scRNA-seq和scATACseq數據進行聚類[23]。MATCHER被提出來通過使用高斯過程潛變量模型來推斷每個細胞的偽時間來預測scRNA-seq和scATACseq之間的相關性[24]。最近,開發了Seurat(版本3)[25]和LIGER [22]用于整合scRNA-seq和scATAC-seq數據。這兩種方法都是先將scATAC-seq數據轉化為類似于基因表達數據的基因活性數據,然后通過在低維空間中相互比對來識別scRNA-seq數據和基因活性數據之間的錨點。然而,兩組學/兩層組學數據之間的比對效率通常需要來自兩種測量的相似聚類性能。由于scATAC-seq數據的極稀疏性(即sci-CAR-seq中超過99%為零),很難通過scATAC-seq數據定義細胞簇。因此,這兩種方法的不正確對齊可能會影響下游分析。
深度生成模型已經成為一個強大的框架來建模高維數據[26,27]。具體地,VAE通過編碼器從輸入數據學習低維特征,并通過解碼器恢復輸入數據,這可以通過最大化恢復的數據和輸入數據之間的似然性,并最小化學習的潛在特征和真實后驗之間的Kullback-Leibler(KL)發散來完成。最近,提出了采用標準VAE的單細胞變分推理(scVI)來分析scRNA-seq數據[26]。然而,標準的VAE在潛在變量上使用單一的各向同性多變量高斯分布,并且通常不適合稀疏數據[28]。SCALE適配使用高斯混合模型(GMM)作為潛在變量的先驗的VAE被提出來分析scATAC-seq數據,分析結果表明,集成VAE和GMM的框架可以用于處理高度稀疏的數據,并學習更分散和可解釋的潛在特征[27]。深度學習多模態技術[29,30]的最近快速發展以及在整合多視圖生物數據[31]方面的成功應用,證明了它們在解決當前單細胞多組學數據分析困難方面的巨大潛力。
在這里,我們提出了單細胞多模態變分自動編碼器(scMVAE),用于整合來自同一單細胞的scRNAseq和scATAC-seq數據,通過使用三種類型的聯合學習策略。scMVAE模型使用隨機優化和多模態編碼器,首先聚合兩種組學數據跨相似細胞和特征,以逼近具有GMM先驗的聯合潛在特征位置,然后通過每種組學數據的解碼器重構觀察到的表達值,同時考慮每種類型數據的歸一化,可用于訓練非常大的數據集。
特別是,通過無監督方式聯合學習兩種組學數據,scMVAE模型(i)產生具有生物意義的低維特征,同時表示這兩個多層剖面,允許細胞可視化和聚類;(ii)去噪和填充兩種組學數據;(iii)構建兩層數據之間的關聯,可用于推斷新的調控關系。為了證明其效率,我們將scMVAE模型和其他整合方法應用于模擬和真實數據集,結果表明scMVAE模型的性能優于當前的最先進方法。
方法
scMVAE概率模型
scMVAE通過三種聯合學習策略對來自同一細胞的scRNA-seq和scATAC-seq的分布進行建模:PoE推斷網絡(在材料S1中詳細描述)、神經網絡和直接連接兩種組學數據特征(圖1A–C)。為了平衡scRNA-seq和scATAC-seq數據之間的大尺度差異,我們將scATAC-seq數據的峰值水平計數矩陣轉換為類似于scRNA-seq數據的基因活性數據,建模每個組學數據均來自于一個零膨脹負二項(ZINB)分布。
具體而言,給定K個聚類,可以通過多組學編碼器網絡通過重新參數化獲得聯合學習特征z,其中c是一個概率離散的分類變量。p(z|c)是一個混合高斯分布,其參數是由在c條件下的均值向量μc和協方差矩陣σc參數化的。考慮到x、y和c在z條件下是獨立的,那么多模態聯合學習分布p(x、y、z、c、lx、ly),其中lx和ly分別是用作scRNA-seq和scATAC-seq數據的庫大小因子的一維高斯變量,可以分解為:
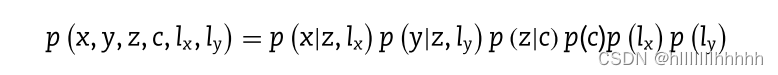


每個因子分解變量定義如下:
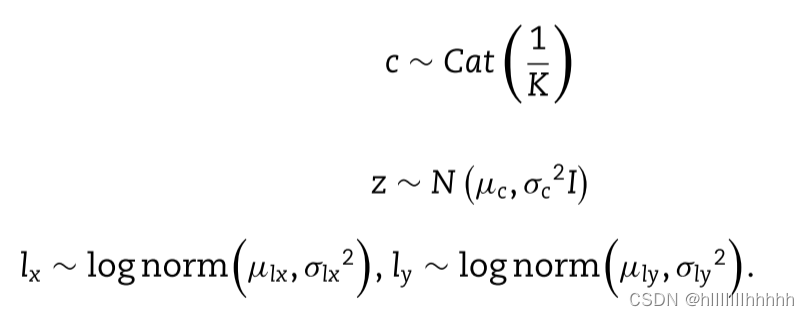
此外,x或y的每個基因表達水平獨立于以下生成過程:
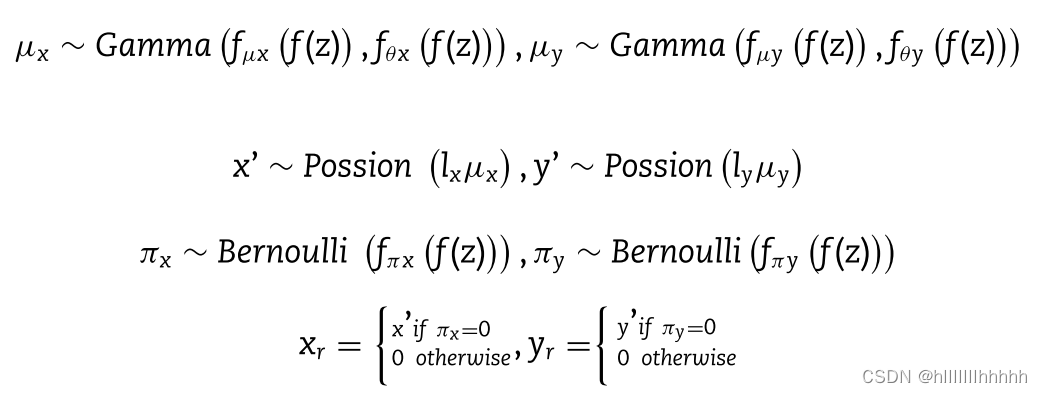
在MVAE中,z的GMM先驗被用來生成高度逼真的樣本,通過學習更加解耦和可解釋的潛在表示。這在先前的工作中分別應用于scRNA-seq和scATAC-seq[27, 32]。lx和ly被視為與經驗日志庫大小強相關的對數正態分布。fθx(f(z))和fθy(f(z))表示由變分貝葉斯推斷估計的特定特征的反比例。
在推斷期間,神經網絡fμx和fμy通過在最后一層使用‘softmax’激活函數被約束,以編碼一個細胞中所有基因的平均比例基因表達,分別用于scRNA-seq和scATAC-seq數據。神經網絡fπx和fπy通過在最后一層使用‘sigmoid’函數編碼每個基因是否因為捕獲效率和測序深度而被刪除,用于每個二組學數據。
scMVAE模型的訓練旨在最大化觀察到的scRNA-seq和scATAC-seq數據的對數似然,然而,由于這是不可解的,因此轉而優化證據下界(ELBO):
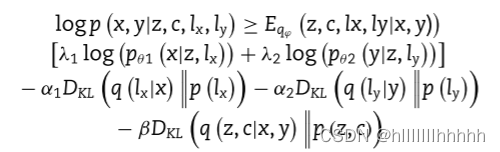
鼓勵使用與庫大小因子lx和ly相關的兩個重建項和KL散度的正則化項來進行數據歸一化、去噪和插值。潛在變量z的KL散度用于將其調節為GMM流形,以增強與多組學數據的關聯。參數q?、pθ1和pθ2分別是多模態編碼器、scRNA-seq數據的解碼器和scATAC-seq數據的解碼器。
所有神經網絡都使用了dropout正則化和批量歸一化。每個神經網絡都有一個或兩個全連接層,每層有128或256個節點。隱藏層之間的激活函數是'relu'函數。使用Adam優化器和1e-6的權重衰減來最大化ELBO。scMVAE模型使用pytorch軟件包實現,其中GMM是使用Python scikit-learn軟件包構建的。源代碼位于GitHub存儲庫:https://github.com/cmzuo11/scMVAE。
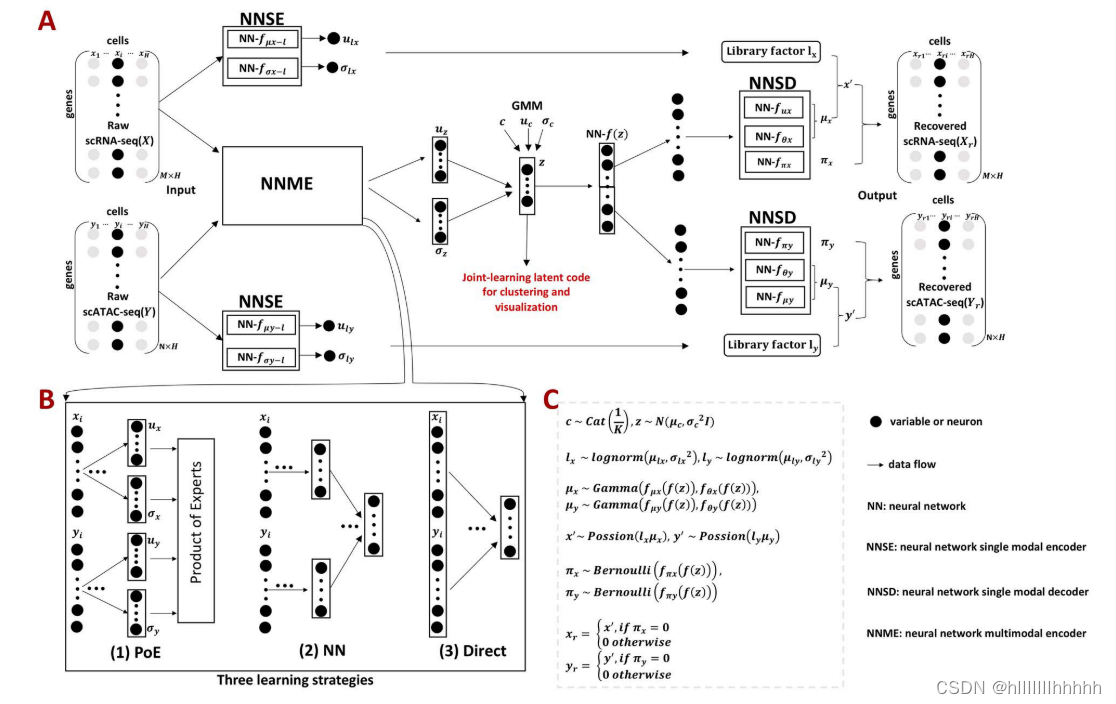
圖1. scMVAE模型的概述,包括三種聯合學習策略。
(A)scMVAE模型的總體框架。給定同一細胞i的scRNA-seq數據(具有M個變量的xi)和scATAC-seq數據(具有N個變量的yi)作為輸入,scMVAE模型通過一個具有三種學習策略的多模態編碼器學習了細胞的非線性聯合嵌入(z),該嵌入可用于多種分析任務(例如細胞聚類和可視化),然后通過解碼器對每個組學數據將其重構回原始維度作為輸出。注意:兩種組學數據的相同細胞順序確保了一個細胞對應于低維空間中的一個點。
(B)三種學習策略的示意模型:
(i)‘PoE’框架用于通過每個組學數據的后驗概率的乘積來估計聯合后驗(詳見材料S1),
(ii)‘NN’用于通過使用神經網絡來組合為每個層數據提取的特征來學習聯合學習空間
(iii)‘Direct’策略通過直接使用兩層數據的原始特征的串聯作為輸入一起學習。在這種學習條件下,神經網絡:NN?fμy?l,NN?fσy?l,NN?fμy,NN?fθy,NN?fπy已從總網絡中刪除。
(C)scMVAE模型中每個變量所屬的分布。每個組學數據都被建模為一個ZINB分布。有關每個變量的詳細描述,請參見數據集和預處理。






)

)



)



![latex中\documentclass[preprint,review,12pt]{elsarticle}的詳細解釋](http://pic.xiahunao.cn/latex中\documentclass[preprint,review,12pt]{elsarticle}的詳細解釋)

征稿開啟!)
