這一次我們從一些已經發表的文章拆解,我們來看看,你找到了一個核心基因以后,你可以怎么做呢?我們就不說那么多廢話了,直接用幾篇文章的解讀來帶著大家領會一下如何去進行下一步的分析。
Case1:預后標志物+免疫浸潤
第一篇文章是2019年發表在 Front Oncol(IF=4.1437)名為:UBASH3B Is a Novel Prognostic Biomarker and Correlated With Immune Infiltrates in Prostate Cancer的文章。
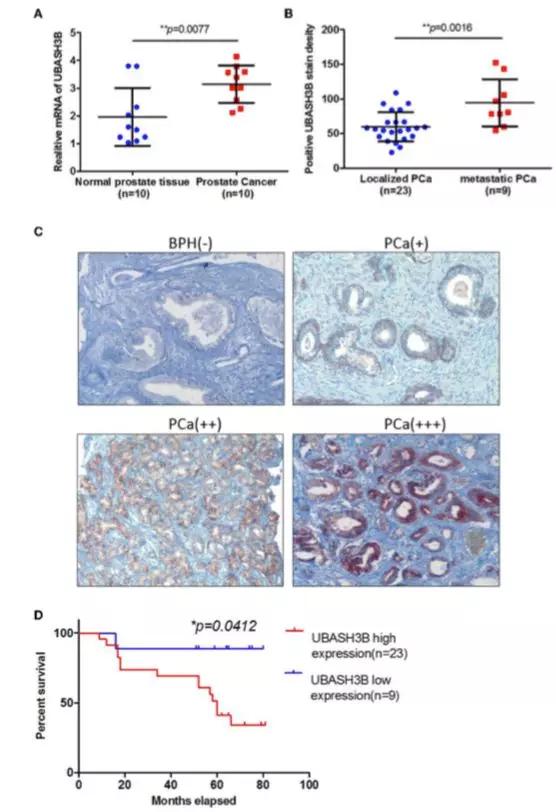
不得不說,隨著免疫檢查點抑制劑在各種腫瘤治療中大放異彩,和免疫檢查點抑制劑療效相關的一些標志物也一起受到了廣泛的關注,比如說我們的第一個case“免疫浸潤”就是一個很好的例子。
免疫浸潤不僅僅常用到腫瘤的免疫檢查點抑制劑療效中,還有其他的疾病的發生發展也都和“免疫浸潤”息息相關,學習方法和套路;然后從這個所謂的“套路思維”中跳出來,這才是希望大家可以達到的。
- 通過腫瘤組織和正常組織的mRNA表達:發現核心基因在腫瘤組織中顯著升高;
- 在轉移的組織和惡性程度更高的組織中核心基因也更高;
其實我覺得還不如用Cox來篩選來得好,有那么多基因都滿足上面的兩點,作者為什么要研究UBASH3B這個基因?或者是通過高通量篩選出只有UBASH3B滿足上面的兩個標準,這樣的方法更為嚴謹。
- 通過核心基因的高低表達把人群分成2組做差異分析并得到差異基因(DEGs)
- 通過上面的得到的DEGs進行通路富集來解釋為什么會引起生存差異
- 然后發現DEGs和顯著激活差異的通路和免疫浸潤相關
- 最后做了一下核心基因和免疫浸潤相關基因以及通路的相關性分析
這樣的思路的確是比較簡單粗暴的,肯定是可以這樣的做的,但是顯然也可以做得更好。
小結:
首先,可以建議大家可以補做CIBERSORT或者是xCell這樣的分析,不建議做Timer或者是Estimate,因為信息量太少了。其次,作者是有用到自己的數據的,但是如果能有20-30個樣本并且是帶隨訪的RNAseq數據來進行篩選,感覺說服力就強很多了,當然作者為了增強說服力,從多種維度進行驗證,彌補了缺陷。最后,如果大家能有現成的基因敲除鼠,或者是能夠有免疫重建的PDX模型,那么能在現在的基礎上補做一些機制實驗,在10分左右還是很有希望的。
Case2:單基因多組學驗證生存
第二篇文章是2020年發表在J Clin Med(IF=5.688)上名為Opposite Roles of BAP1 in Overall Survival of Uveal Melanoma and Cutaneous Melanoma的文章;
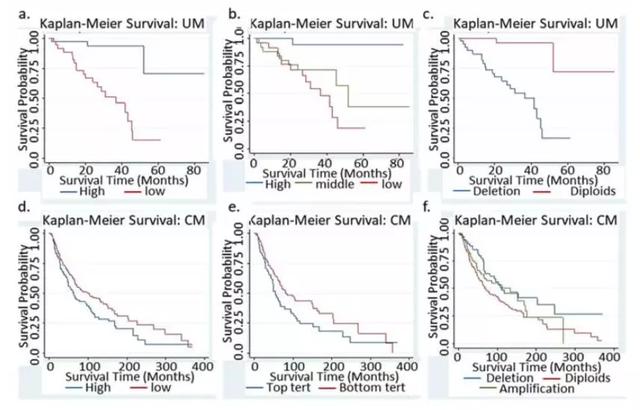
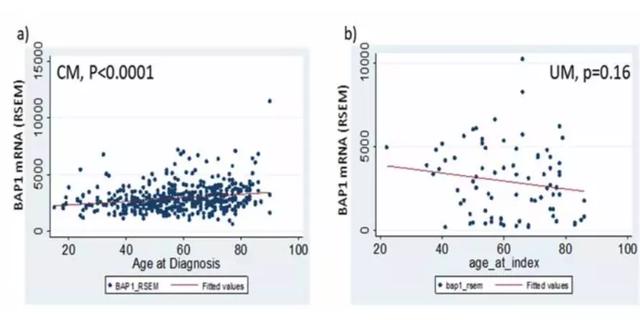
這篇文章很簡單,大致內容如下:
- 核心基因的低表達組和缺失組有著更壞的生存;
- 然后用Cox回歸分析也同樣證明了第一步的結論
- 擴展分析了核心基因的表達量和診斷年齡顯著相關
是的,就這樣戛然而止了,很詫異。反思了一下,這個可能是一個“約稿”,畢竟JCM是MDPI的雜志,他們雜志就愛搞這種“特刊+約稿”的事情。當然,按照題目,作者發現了核心基因分別在uveal melanoma (UM) and cutaneous melanoma (CM)的生存意義是不一樣的…這也算是一個重大發現吧…
那么我們從這篇文章的分析跳出來,看看我們還可以做哪些東西,從而做得更好呢?如果找到一個和生存相關的基因,接下來除了重復性質的驗證,你還可以做什么呢?
- 找到最合適的臨界值,建議xTile或者是tROC,而不是直接的median;
- 用GSEA或者是ssGSEA來解釋生存預后以及和核心基因的關系;
- 橫向分析有很多,除了作者做的年齡;還有可以想到的臨床分期分型,腫瘤大小,一些熱門的score;
- 從核心基因本身出發探尋通路和互作關系;
本來有一篇文章要分析,但是因為篇幅性質,就沒有放進來。那是講的篩選出來的基因是一個自噬相關的基因。篩選出那個基因了以后,就可以看看那個基因和自噬相關的通路那些是相關的;如果運氣好還可以在GEO看看,說不定能找到一些別人調控過該基因的分析;
當然還可以通過疾病類型進行擴展分析,比如說研究NSCLC的時候,把LUAD和LUSC分開來做亞組補充分析的信息量;
Case3:基因突變+TMB+免疫浸潤
第二篇文章是2020年發表在Aging(IF=5.515)上名為EP300 mutation is associated with tumor mutation burden and promotes antitumor immunity in bladder cancer patients的文章。
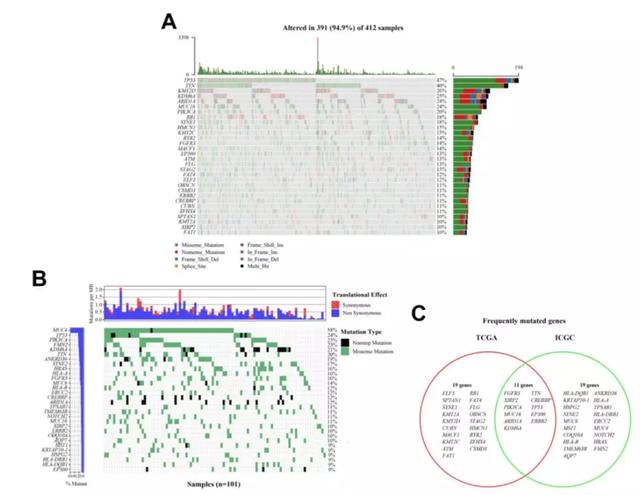
這篇文章的核心基因是EP300,但是它和上面的文章不一樣的是,它是做的基因突變,當然隨著多組學數據的開放,你可以做拷貝數變異,可以做蛋白芯片,可以做甲基化,可以做乙酰化等;RNA seq的數據最多并且干預調控比較容易,大部分的時候大家都還是以RNAseq的數據為核心方式進行展開自己的文章。
這篇文章為什么要研究EP300 mutation,作者一步一步篩選出來就很有邏輯,大家可以看一看:
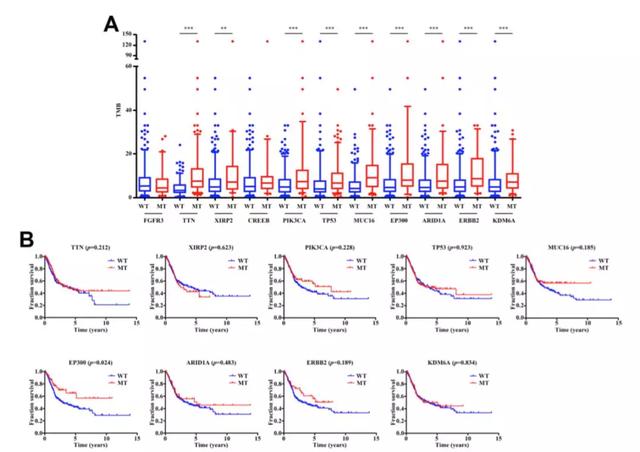
定義Frequently Mutated Gene(FMG)→把TCGA和ICGC兩個數據集中的FMGs取交集(11個基因)→TMB的差異分析(9個顯著)→生存分析中僅EP300 mutation顯著
那么全文思路大致如下:
- 篩選了核心基因:和TMB以及生存存在關聯;
- Cox回歸分析驗證核心基因和生存的關系;
- GSEA分析看核心基因突變與否和通路激活程度的關系;
- 核心基因突變與否和CIBERSORT的免疫浸潤之間的關系;
討論也比較簡單,這篇文章比較大的一個亮點是作者很有邏輯地篩選出來了EP300 mutation,至于后面的分析的確是比較少了;該作者既沒有看那個基因突變是否和其他組學的關聯,也沒有看是否有熱點的突變;擴展的分析也就止步于GSEA和CIBERSORT。
其實,我在想如果是能夠從GSEA分析中找到一類通路,然后和CIBERSORT的結合起來講一個故事可能會更好?或者是找到一些通路,進行干預他們對一些表型的關系?如果是有一些臨床的樣本,結合上現實生活中ICIs治療的案例,配上case report的一些描述和影像資料,我覺得文章還能再上一個檔次。
三個案例分享完了,希望大家能夠從里面學到很多新的東西,請點下右下角的在看喲,哈哈哈其實厲害的同學會說,這不就是所謂的單基因套路嗎?那我問你套路香嗎?
當然香呀!!!!!
并且套路都沒學會就開始高級創新了??????



















