一、概念
首先還是老規矩,處理一下概念問題,好將之后的問題進行分類和區分
單細胞轉錄組:指在單個細胞水平上對轉錄組(即細胞內所有轉錄出來的 RNA,主要是 mRNA)進行研究的學科或技術方向,核心是分析單個細胞中基因的表達情況(包括哪些基因被激活表達、表達量高低等),從而揭示細胞間的異質性、功能差異及分子調控機制。
????????可以簡單理解為:研究 RNA——>揭示細胞內群體異質性、定義細胞類型、細胞狀態和細胞的動態轉變
空間轉錄組(ST):是一種結合空間位置信息和基因表達數據的技術,能夠在組織切片上解析基因表達的空間分布模式,彌補了傳統單細胞轉錄組技術丟失空間信息的缺陷?。
????????空間轉錄組能夠將基因表達信息與細胞在組織中的具體位置相結合,從而揭示細胞類型的分布、細胞亞群的空間組織以及細胞間的相互作用。
二、具體應用
這個部分只專注于實驗設計和樣本,不關注研究內容
案例一:scRNA-seq+ST破譯結直腸癌干細胞異質性與轉移路徑
該研究通過scRNA-seq和空間轉錄組(ST)分析,揭示了結直腸癌中異質性癌癥干細胞樣細胞(CSCs)及其器官特異性轉移的機制。
?
樣本:8例原發性CRC、5例oCRC(卵巢轉移)、2例lCRC(肝臟轉移)、7例鄰近正常組織和7例外周血單核細胞(PBMC),共29例樣本。
?研究路線: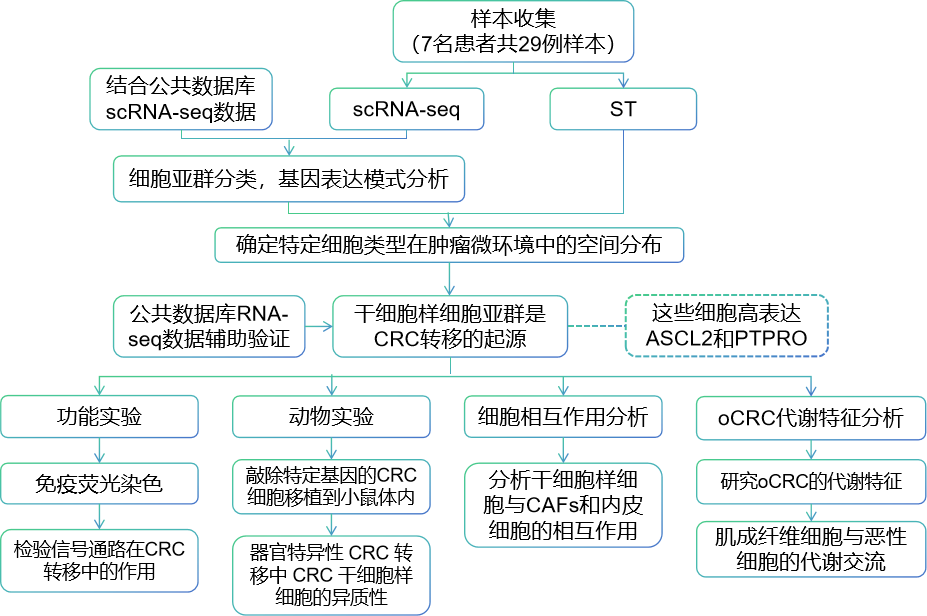
?案例二:scRNA-seq+ST表征缺血小鼠大腦的分子和細胞結構
本研究利用空間轉錄組學和scRNA-seq技術對缺血性腦卒中小鼠模型的大腦進行了深入分析,揭示了腦卒中后神經炎癥反應中的關鍵分子和細胞變化。

樣本:接受光栓塞手術組和假手術對照組的兩組小鼠的梗死周邊區域
研究路線:
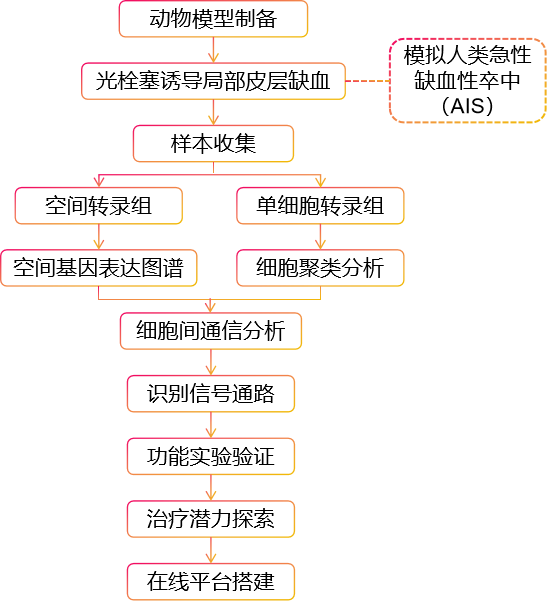
?案例三:scRNA-seq+ST揭示HNSCC中CAF亞群限制了CD8+T細胞的浸潤和抗腫瘤活性
本文為關于頭頸部鱗狀細胞癌(HNSCC)中癌癥相關成纖維細胞(CAF)亞群對CD8+ T細胞浸潤和抗腫瘤活性的影響。研究通過空間轉錄組和scRNA-seq技術,揭示了特定的CAF亞群與CD8+ T細胞浸潤限制和功能障礙相關。這些CAFs表達高水平的CXCL9、CXCL10、CXCL12和MHC-I分子,并且富含半乳糖結合蛋白-9(Gal9)。
樣本:來自HNSCC患者5個腫瘤組織和5個配對的鄰近正常組織
研究路線:
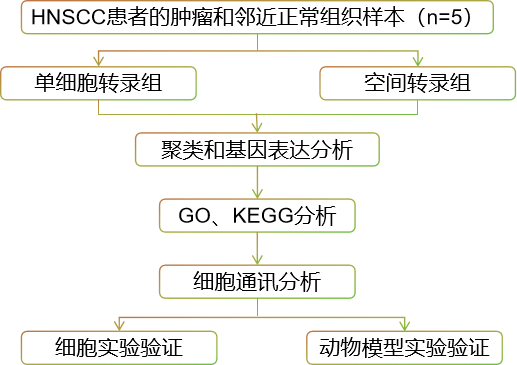
?三、整合方法分析方法總結
在《Nature reviews genetics》發表綜述文章,回顧了整合scRNA-seq與空間轉錄組學技術研究的嘗試和努力,包括新興的整合計算方法,并提出了有效結合當前方法的途徑。
參考文獻
Longo, S.K., Guo, M.G., Ji, A.L. et al. Integrating single-cell and spatial transcriptomics to elucidate intercellular tissue dynamics. Nat Rev Genet (2021). https://doi.org/10.1038/s41576-021-00370-8
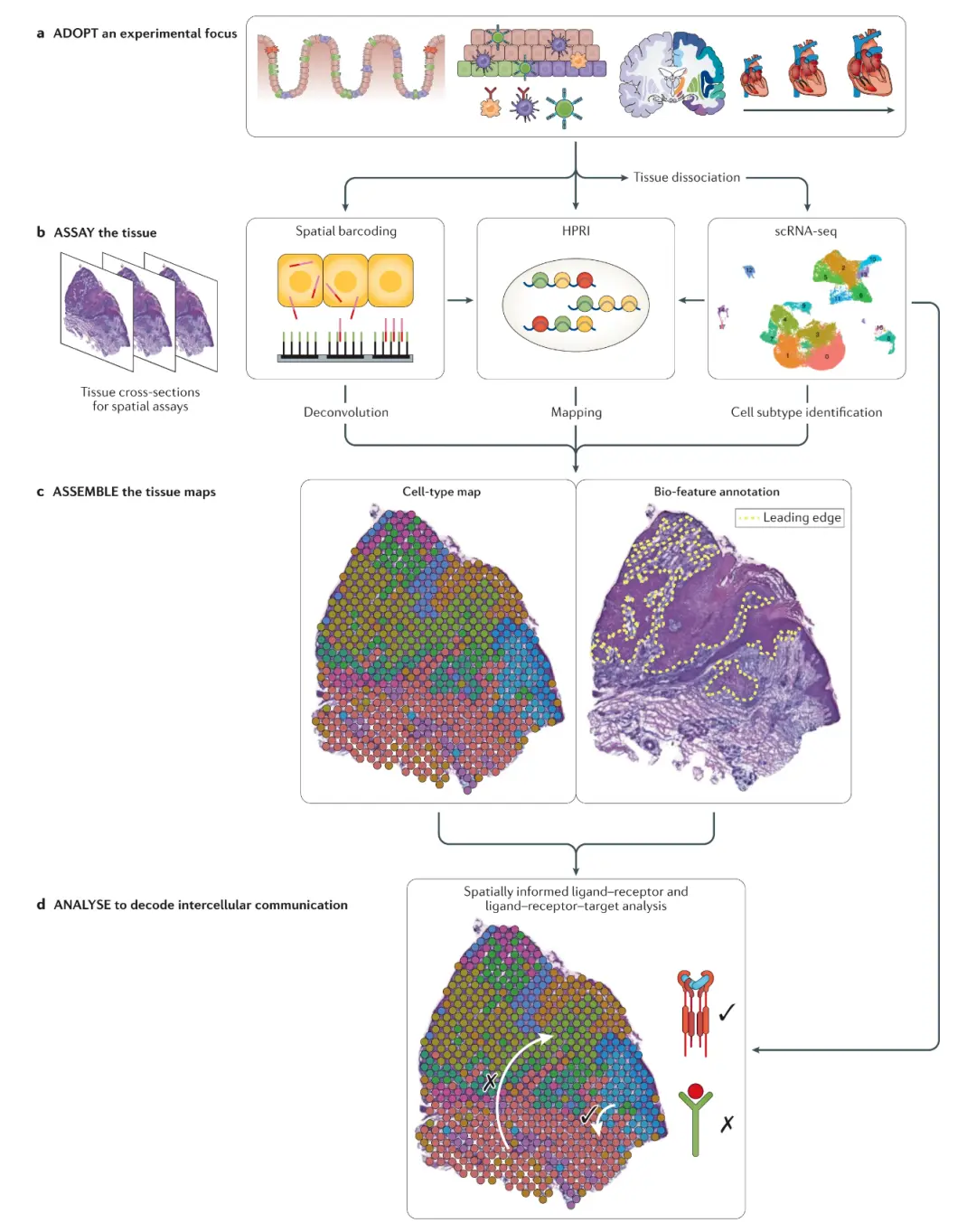
?????????整合scRNA-seq和空間轉錄組學研究的流程模式
核心流程(a→b→c→d)
- a ADOPT:確定實驗對象(如組織、器官、疾病模型),明確研究聚焦方向(分子、細胞、器官層面)。
- b ASSAY:組織分析環節,包含 3 類技術:
- Spatial barcoding:空間條形碼技術(如 10X Visium),通過 “空間反卷積(Deconvolution)” 解析混合細胞的空間分布;
- HPRI:高分辨率原位分析(High - Plex RNA Imaging),實現基因表達的空間 “映射(Mapping)”;
- scRNA - seq:單細胞轉錄組測序,用于 “細胞亞型鑒定(Cell subtype identification)”。
- c ASSEMBLE:整合分析結果,生成 2 類圖譜:
- Cell - type map:細胞類型空間分布圖(標注不同細胞類型的區域);
- Bio - feature annotation:生物特征注釋圖(如標記 “Leading edge” 等功能區域)。
- d ANALYSE:基于空間圖譜,解析 “細胞間通訊(intercellular communication)”,驗證配體 - 受體互作對(Ligand - receptor pairs)及下游調控關系。
?scRNA-seq+空間組學整合分析的研究進展(2021年止)
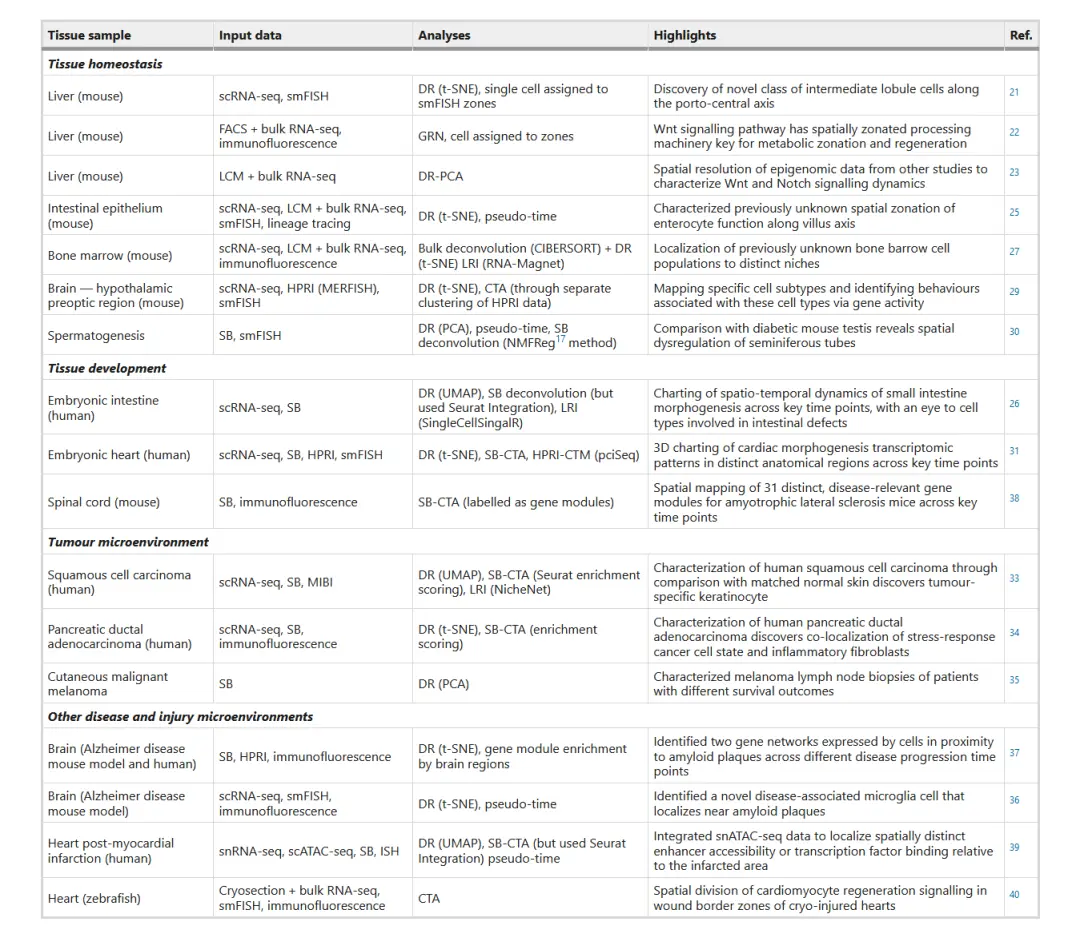
?解析scRNA-seq和空間轉錄組數據的研究
核心維度(列)
- Tissue sample:研究對象(組織 / 疾病模型),分 5 大類:組織穩態(Tissue homeostasis)、組織發育(Tissue development)、腫瘤微環境(Tumour microenvironment)、其他疾病微環境(Other disease and injury microenvironments)。
- Input data:實驗輸入數據類型,如單細胞測序(scRNA-seq)、單分子熒光原位雜交(smFISH)、流式分選(FACS)、激光捕獲顯微切割(LCM)等。
- Analyses:分析方法,涉及降維(DR,如 t-SNE、UMAP )、基因調控網絡(GRN)、反卷積(Deconvolution)、擬時序分析(pseudo-time )等。
- Highlights:研究亮點(核心發現),如揭示基因表達的空間分區(spatial zonation )、細胞亞型映射(cell subtypes mapping )、疾病相關分子網絡(gene networks )等。
- Ref.:文獻引用編號(對應研究的參考文獻)。
研究場景覆蓋
涵蓋正常組織穩態(如肝臟、腸道)、發育(胚胎腸道、心臟)、腫瘤(鱗狀細胞癌、胰腺癌)、疾病模型(阿爾茨海默病、心肌梗死)?等方向,體現空間組學在解析組織異質性、細胞功能、疾病機制中的應用。
scRNA-seq+空間組學整合分析的計算方法
整合scRNA-seq和空間轉錄組數據有兩種主要方法:去卷積(Deconvolution)和映射(Mapping)。去卷積旨在根據單細胞數據,從每個捕獲點的mRNA轉錄物的混合物中分離出離散的細胞亞群;映射有兩方面:將指定的基于scRNA的細胞亞型定位到HPRI圖譜上的每個細胞和將每個scRNA-seq細胞定位到組織的特定生態位或區域。
- 去卷積:被用于從混合細胞樣本中分離出不同細胞類型的表達模式。
- HPRI(High-plex RNA Imaging)?:高通量RNA成像技術,提供單細胞分辨率的空間數據,用于將scRNA-seq識別的細胞類型映射到組織中的實際位置。
- 映射:以單細胞分辨率創建空間分辨率的細胞類型映射。
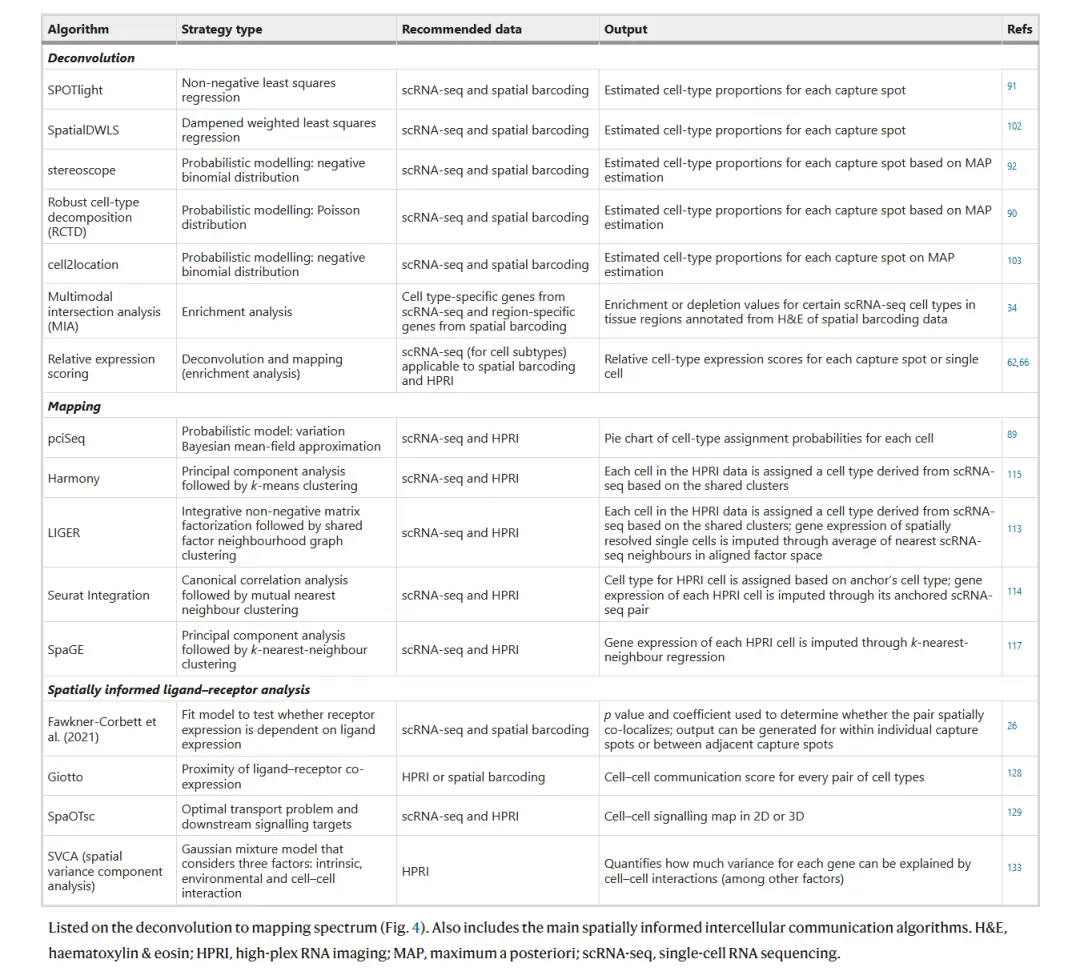
空間轉錄組與單細胞數據分析工具的分類對比表
核心維度(列)
- Algorithm:算法名稱(如 SPOTlight、stereoscope 等),覆蓋 3 類分析方向:
- Deconvolution(去卷積):解析混合信號的細胞類型比例(如 SPOTlight、SpatialDWLS );
- Mapping(映射):關聯單細胞與空間數據(如 scSeq、Harmony );
- Spatially informed ligand-receptor analysis(空間配體 - 受體分析):解析細胞間通訊(如 SpaOTsc、Giotto )。
- Strategy type:算法策略(如 Non-negative least squares regression、Probabilistic modelling )。
- Recommended data:推薦輸入數據類型(scRNA-seq、空間條形碼數據、HPRI 等)。
- Output:工具輸出結果(如細胞類型比例、配體 - 受體互作對 )。
- Refs:文獻引用(對應工具的方法學或應用文獻)。
價值與應用
這張表匯總了空間多組學分析的主流算法工具,覆蓋去卷積、數據整合、細胞通訊?3 大核心場景,可用于指導空間轉錄組 + 單細胞數據分析的工具選型(如解析細胞類型比例選 SPOTlight,做細胞通訊選 SpaOTsc )。
單細胞和空間轉錄組數據的整合策略
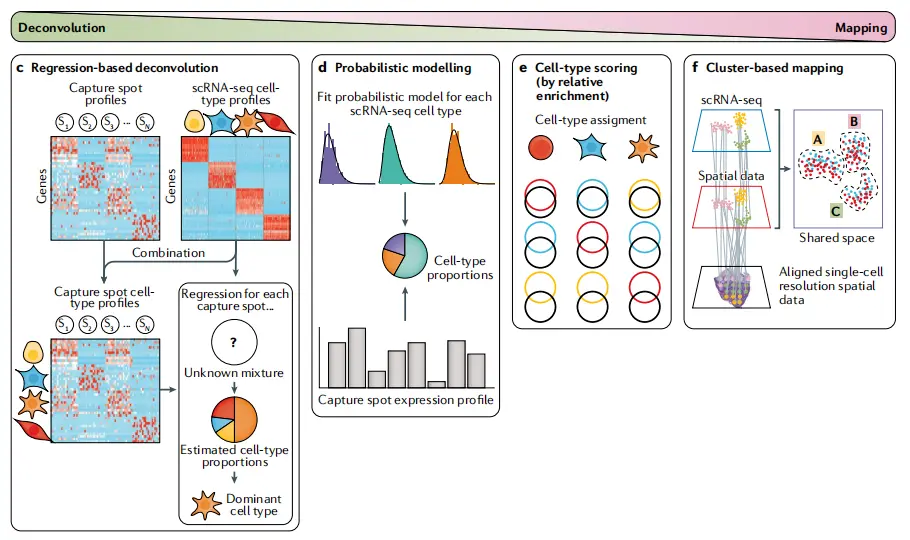
“去卷積(Deconvolution)與映射(Mapping)” 流程的示意圖
模塊拆解(左→右)
-
c Regression-based deconvolution(基于回歸的去卷積):
用單細胞轉錄組的細胞類型表達譜(scRNA-seq cell-type profiles),通過回歸模型(Regression)解析空間捕獲點(Capture spot)的混合表達信號,估算細胞類型比例,識別 “主導細胞類型(Dominant cell type)”。 -
d Probabilistic modelling(概率建模去卷積):
為每種單細胞類型擬合概率模型(如分布曲線),結合空間捕獲點的表達譜(Capture spot expression profile),推斷細胞類型比例(Cell-type proportions)。 -
e Cell-type scoring(細胞類型評分映射):
通過 “相對富集(relative enrichment)” 給細胞類型打分,完成 “細胞類型分配(Cell-type assignment)”,用不同符號 / 顏色標記空間點的細胞類型歸屬。 -
f Cluster-based mapping(基于聚類的映射):
將單細胞轉錄組(scRNA-seq)與空間數據(Spatial data)映射到 “共享空間(Shared space)”,通過聚類(Cluster)對齊,生成 “單細胞分辨率的空間數據(Aligned single-cell resolution spatial data)”,劃分 A、B、C 等區域。
核心邏輯
這是空間轉錄組與單細胞數據整合的典型方法,解決 **“單細胞類型的空間定位”** 問題 —— 先通過去卷積解析混合信號中的細胞類型比例,再通過映射(Mapping)將細胞類型與空間位置關聯,最終構建 “細胞類型 - 空間分布” 圖譜,常用于腫瘤微環境、發育生物學等研究。
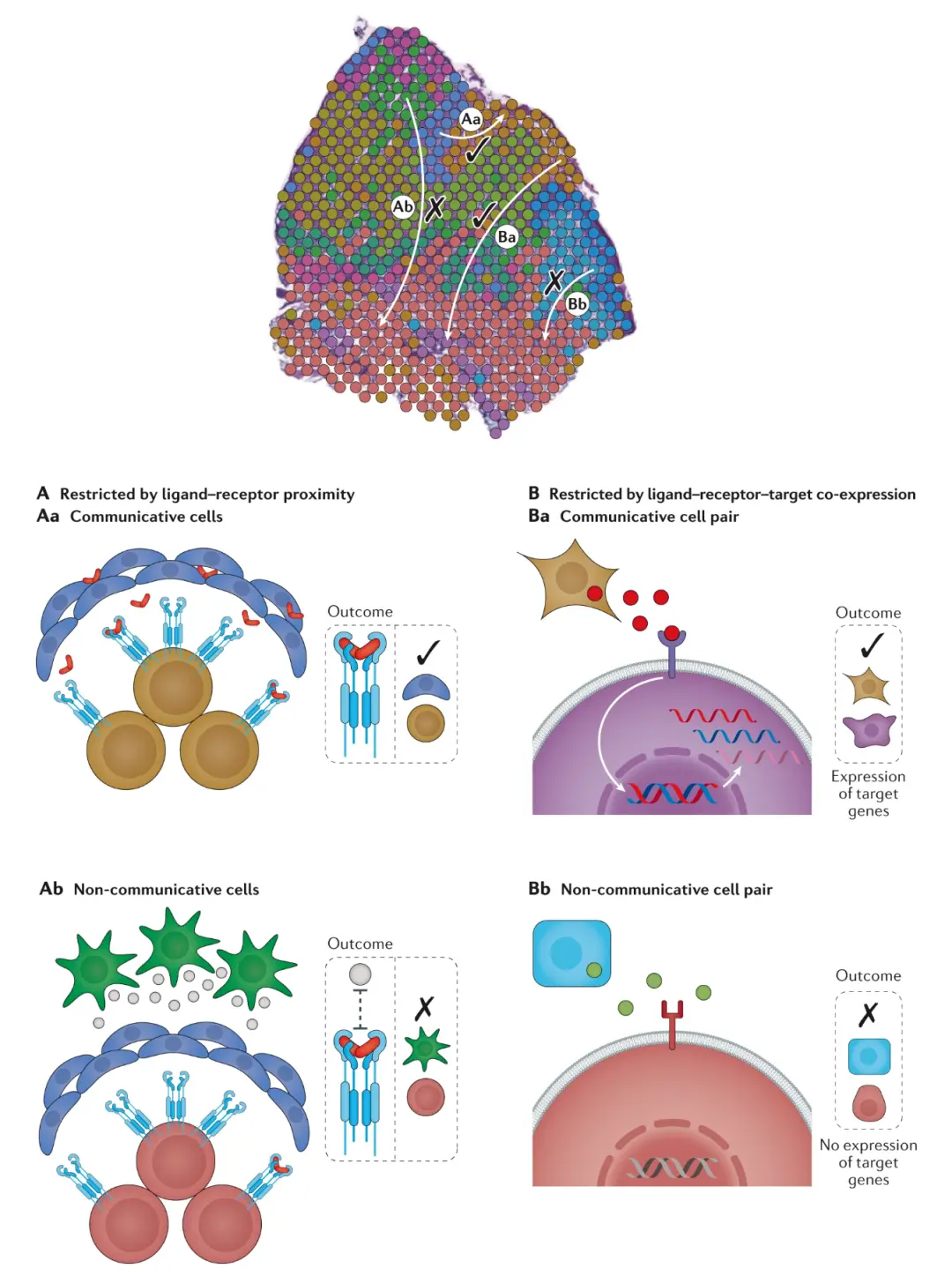
空間轉錄組學中細胞間通訊機制的示意圖
核心模塊
- 上方組織圖譜:標注不同細胞通訊狀態(Aa/Ba 為可通訊,Ab/Bb 為不可通訊 ),體現細胞通訊的空間特異性。
- 左側(A):受 “配體 - 受體 proximity(空間鄰近性)” 限制的細胞通訊:
- Aa:細胞空間鄰近 + 配體 - 受體結合→可通訊(Outcome:√ );
- Ab:細胞空間分散 / 配體 - 受體不結合→不可通訊(Outcome:× )。
- 右側(B):受 “配體 - 受體 - 靶基因共表達” 限制的細胞通訊:
- Ba:配體結合受體后,靶基因激活表達→可通訊(Outcome:√ );
- Bb:配體 - 受體未觸發靶基因表達→不可通訊(Outcome:× )。
價值
這是空間細胞通訊分析的典型模型,解釋 **“為什么有些細胞在空間上鄰近卻無法通訊”** —— 除了物理距離,還需滿足 “配體 - 受體結合” 和 “靶基因激活” 等條件,為解析腫瘤微環境、發育信號通路提供理論框架。
詳細文章鏈接:
熱點綜述 | 單細胞+空間轉錄組的整合分析方法總結_空間轉錄組細胞通訊方法-CSDN博客
單細胞轉錄組+空間轉錄組聯合應用,1+1>2-CSDN博客
)








)
)








)