前言
本文是基于 3D 的分子生成方法DeepFMPO v3D的介紹及安裝使用。
一、DeepFMPO v3D是什么?
github代碼
介紹文章
在藥物發現中,如何尋找具新穎性和結構多樣性的候選分子是頗受藥物設計科學家關注的問題。通過虛擬篩選的化學空間搜索往往會受限于篩選庫的大小和構建方式,而分子生成則擴大了化學空間搜索的自由度,近年來發展了不少AI分子生成模型。
來自阿斯利康的Jonas Bostr?m等人在Journal of Chemical Information and Modeling上發表的文章 “On the Value of Using 3D Shape and Electrostatic Similarities in Deep Generative Methods”,該團隊之前公開了一個基于片段的強化學習的二維分子生成工具DeepFMPO,本文中作者展示了一種基于 3D 的分子生成方法(DeepFMPO v3D),并開發了一個免費開源的python包(ESP-Sim)計算片段對之間的靜電勢 (ESP) 相似性的,從三維形狀和靜電勢相似性上改進特征,更準確地生成與輸入分子結構具有相似性的優化分子。
作者展示了一種基于 3D 的分子生成方法(DeepFMPO v3D),并開發了一個免費開源的python包(ESP-Sim)計算片段對之間的靜電勢 (ESP) 相似性的,從三維形狀和靜電勢相似性上改進特征,更準確地生成與輸入分子結構具有相似性的優化分子。

DeepFMPO v3D工作流程: 以片段取代連接點氫原子,采用RDKit為每個片段生成10個能量最低構象,生成能量最低構象,以連接點對齊保留相似性最高分子,從連接點移除相同起始部分,計算RESP等原子電荷,計算對齊構象的ESP相似性,ESP-Combo 打分相似性評價。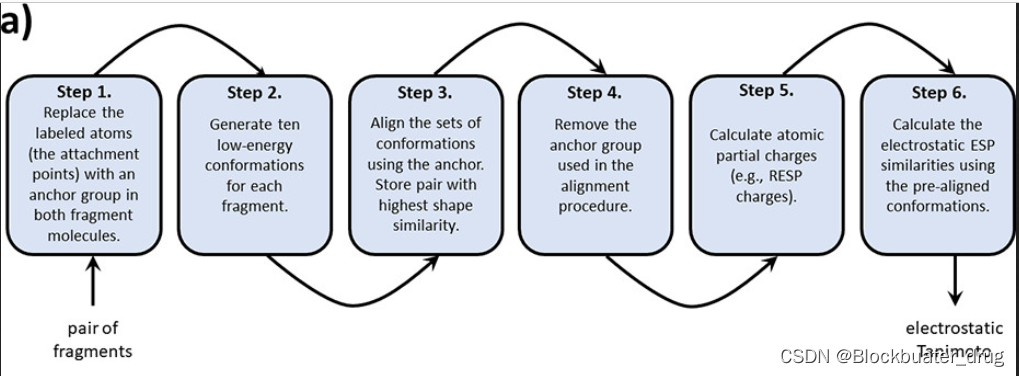
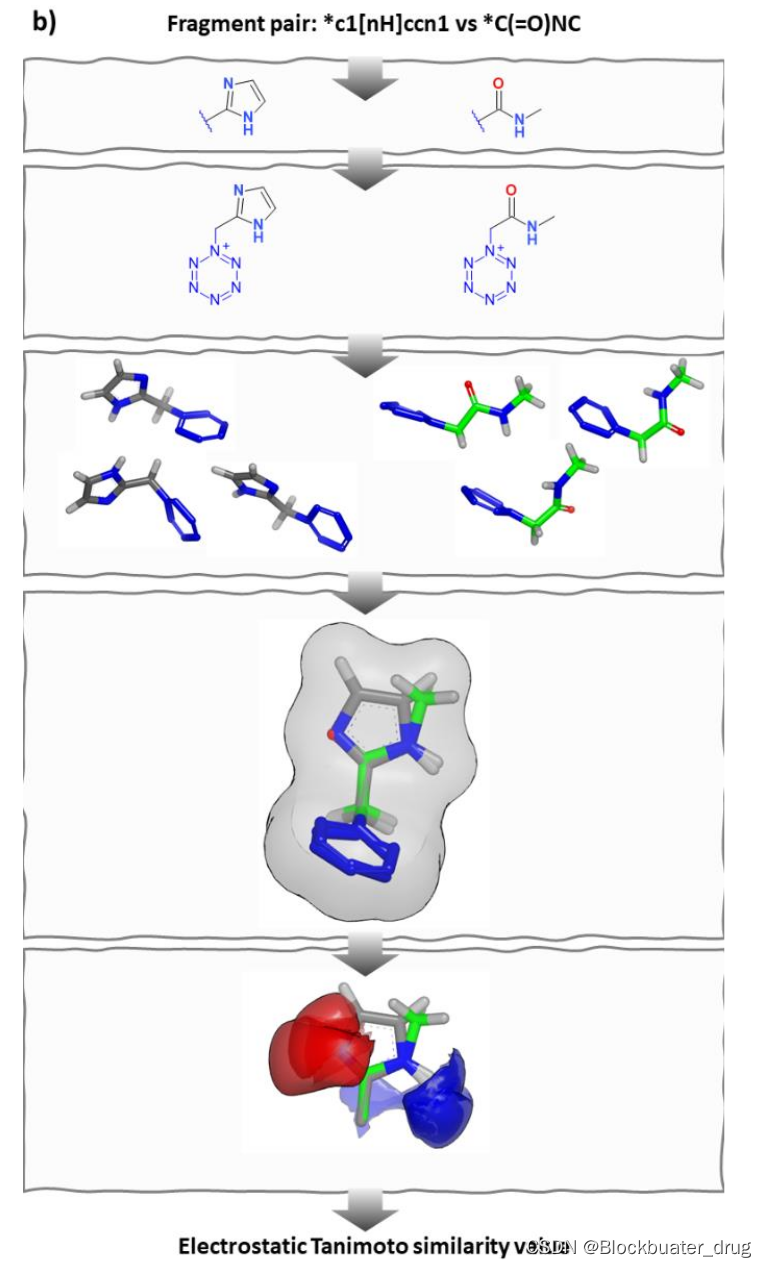
以上流程的具體示例:
ESP-Sim 基準測試:
為了評估使用不同部分電荷對ESP-Sim的影響,作者計算了相同分子相同幾何結構不同部分電荷的ESP相似性。選擇約3000個中性分子,使用高精度量子化學(QM)計算獲得的RESP電荷再現分子的靜電勢,將不同部分電荷獲得的靜電勢與QM獲得的RESP電荷進行相似性比較。作者評估了Gasteiger、MMFF94和AM1-BCC部分電荷,以及機器學習模型 (ML)得到的部分電荷。
下表概述了各個部分電荷與 RESP 電荷的平均絕對偏差,以及通過 Carbo 或 Tanimoto 相似性評估的ESP相似性。可以發現 AM1-BCC電荷重現QM 靜電勢的效果較好,其次是深度學習模型、MMFF 和 Gasteiger。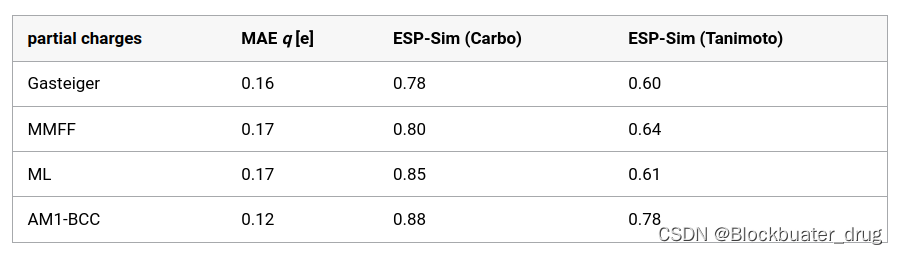
二、安裝步驟
安裝環境:Ubuntu 22.04,? CUDA runtime版本11.8。
安裝步驟:先安裝espsim,然后安裝DeepFMPO v3D
1. 創建DeepFMPO v3D運行的conda環境
為DeepFMPO v3D運行創建虛擬環境,包含espsim安裝。
下載deepFMPOv3D:
git clone https://github.com/giovanni-bolcato/deepFMPOv3D.git
cd deepFMPOv3D將以下內容保存為environment.yml:
?
name: deepFMPOv3D_envchannels:- rdkit- pytorch- conda-forgedependencies:- python=3.10- pytorch- numpy- scikit-learn- scipy- matplotlib- joblib- tqdm- keras- pandas- tensorflow- pip- pip:- resp- rdkit- python-Levenshtein- dask[dataframe]- bisect- git+https://github.com/hesther/chemprop-atom-bond.git創建環境:
?
conda env create -f environment.yml
如果pip部分不成功,提示 `CondaEnvException: Pip failed`,激活conda環境,單獨安裝pip包即可,如下:
pip install git+https://github.com/hesther/chemprop-atom-bond.git激活conda環境:
?
conda activate deepFMPOv3D_env
在環境中安裝TensorFlow,rdkit等模塊:
conda install numpy tensorflow -c conda-forgepip install resp rdkit pandas python-levenshtein dask[dataframe]?2. 安裝psi4:
conda install psi4 -c conda-forge3. 下載并安裝espsim
git clone https://github.com/hesther/espsim.git
cd espsimpip install -e .?
測試espsim安裝:
python scripts/test_imports.py返回以下即安裝正常:
Test passed, imports work fine.
??
測試espsim運行:
python scripts/test_esp_function.py返回:
三、運行分子生成
激活以上創建的環境,進入deepFMPOv3D目錄:
cd deepFMPOv3DUsage:
python deepFMPO.py -f your_fragment.smi -l your_lead.smi -o your_results.sdfpython deepFMPO.py -f ./Data/molecules.smi -l ./Data/lead.smi -o results.sdfpython decoding_to_sdf.py---
總結
本文是基于 3D 的分子生成方法DeepFMPO v3D的介紹及安裝使用。
???
參考資料
1. GitHub - giovanni-bolcato/deepFMPOv3D: Implementation of Shape and Electrostatic similarity metric in deepFMPO.
2. On the value of using 3D-shape and electrostatic similarities in deep generative methods | Theoretical and Computational Chemistry | ChemRxiv | Cambridge Open Engage








)







)


