禁止商業或二改轉載,僅供自學使用,侵權必究,如需截取部分內容請后臺聯系作者!
文章目錄
-
- 介紹
- 加載R包
- 模擬數據
- 構建網絡
- RMSE指數計算
- 畫圖
- 總結
- 系統信息
介紹
在生物信息學和生態學研究中,組學數據的分析越來越依賴于對微生物群落或基因表達數據中物種或基因間相關性的準確估計。傳統的相關性估計方法,如Pearson和Spearman相關系數,雖然在處理連續數據時表現良好,但在處理組成數據時可能會遇到挑戰。組成數據是由比例構成的,其總和固定,這使得數據的分布特性與傳統的正態分布假設不符,從而影響相關性估計的準確性。
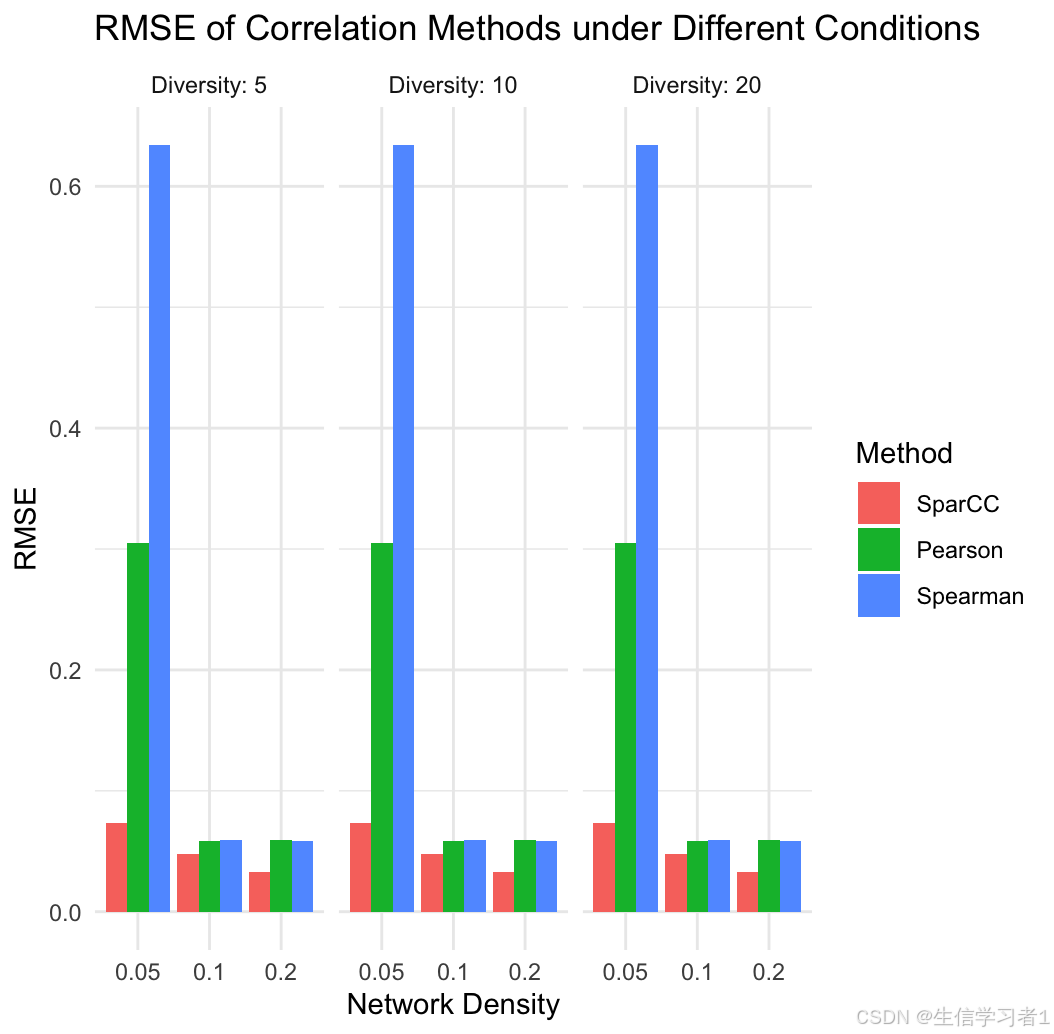
近年來,一種新的相關性估計方法——SparCC(Sparse Correlations for Compositional data)被提出,專門用于處理組成數據。SparCC方法通過稀疏表示和正則化技術,能夠在控制假陽性率的同時,準確地估計組成數據中的相關性。然而,SparCC方法在不同數據特性(如多樣性水平和網絡密度)下的表現如何,以及與傳統方法相比的優勢和局限性,仍需進一步研究。
本研究通過模擬不同多樣性水平和網絡密度下的組成數據,比較了SparCC、Pearson和Spearman三種相關性估計方法的表現。首先,我們生成了合成的組成數據,模擬了不同多樣性水平(5, 10, 20)和網絡密度(0.05, 0.1, 0.2)條件下的微生物群落數據。然后,使用SparCC、Pearson和Spearman方法估計這些數據的相關性,并計算每種方法估計的相關性與真實相關性


:I2C協議實現)





 微服務)

顯示器位置調節)

:SPI協議測試(使用W25Q64))




 high 調優一例(一))
![[Linux] Linux提權管理 文件權限管理](http://pic.xiahunao.cn/[Linux] Linux提權管理 文件權限管理)
pod的管理及優化)