禁止商業或二改轉載,僅供自學使用,侵權必究,如需截取部分內容請后臺聯系作者!
文章目錄
-
- 介紹
-
- 數據準備與模擬
- 相關性分析與邊表生成
- 網絡可視化
- 結果展示與討論
- 加載R包
- 模擬數據
- Spearman 相關 -> 邊表
- 畫圖
- 所有代碼
- 總結
- 系統信息
介紹
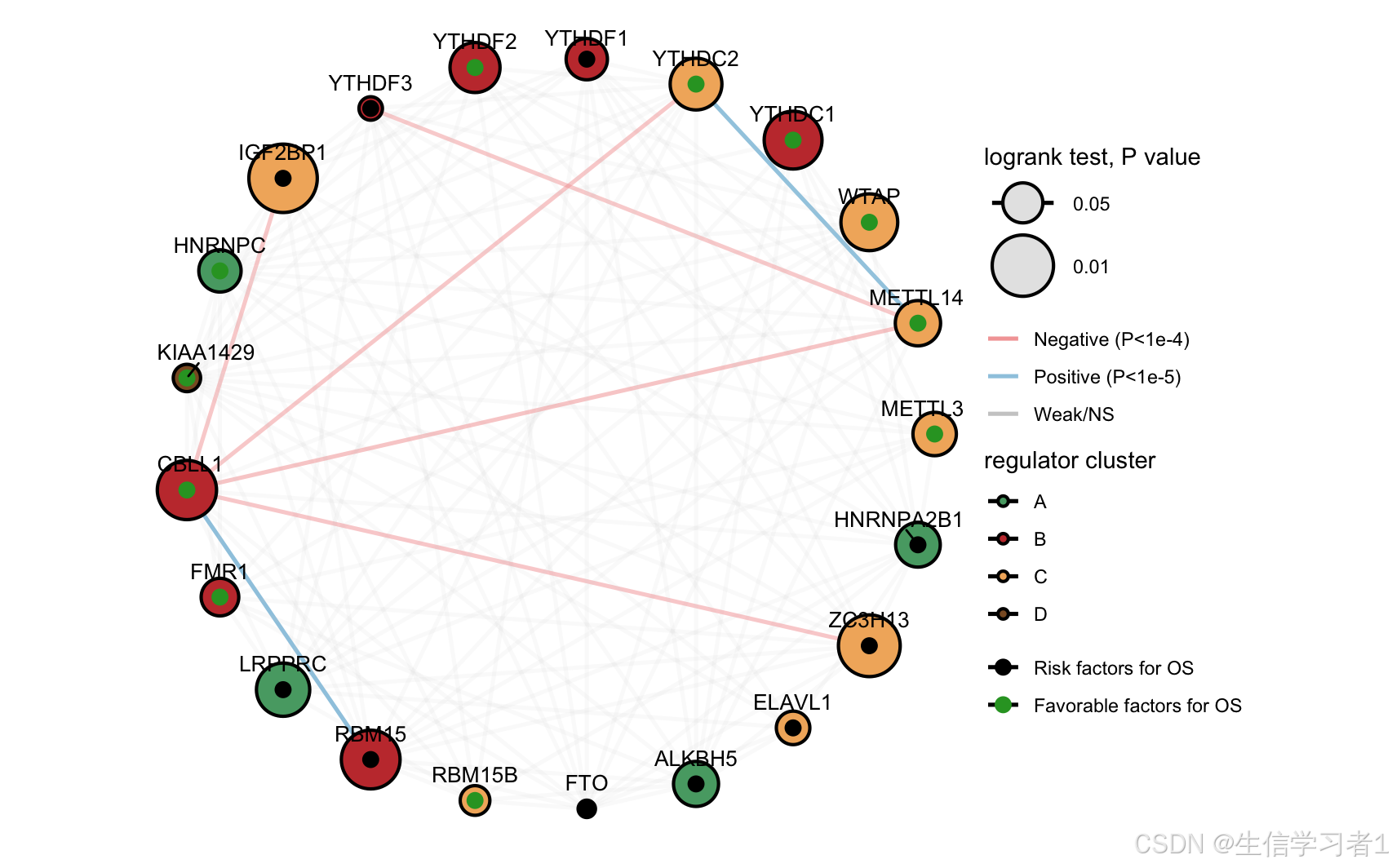
在生物醫學研究中,N?-甲基腺苷(m?A)修飾作為一種重要的表觀遺傳調控機制,近年來受到了廣泛關注。m?A 調控因子在多種生物學過程中發揮著關鍵作用,包括基因表達調控、細胞分化、免疫反應以及腫瘤發生等。為了深入理解這些調控因子之間的相互作用及其對細胞功能的影響,構建一個系統化的調控網絡顯得尤為重要。本研究通過模擬數據,展示了一個完整的分析流程,從基因表達矩陣的生成到基于 Spearman 相關性的網絡可視化,旨在為研究者提供一個可復現的分析模板。
數據準備與模擬
研究首先定義了 21 個 m?A 調控因子,包括 METTL3、METTL14、WTAP 等,這些因子被隨機分配到四個功能簇(A、B、C、D)中。每個基因還被賦予了生存效應(風險或有利)和 log-rank P 值,用于后續的節點屬性展示。為了模擬基因表達數據,研究生成了一個包含 120 個樣本的表達矩陣。通過潛在因子模型,確保了簇內基因表達的強正相關性和簇間基因表達的輕微負相關性,同時加入了隨機噪聲以模擬真實數據的復雜性。
相關性分析與邊表生成
利用 Hmisc 包中的 rcorr 函數,研究計算了基因表達矩陣的 Spearma



Jetson Nano 系統Ubuntu22.04安裝ROS 2 Humble版本)




-PingInfoView)










