概念
多模態空間組學:簡單來說,就是同時研究生物組織里的多種分子信息(比如基因表達、蛋白質、代謝物、表觀遺傳標記等),而且這些信息還帶有空間位置。
MISO(MultI-modal Spatial Omics)是這篇論文提出的一種算法,專門用來分析這些復雜的多模態空間組學數據。它的目標是把不同類型的數據(比如基因表達和組織切片的顯微鏡圖像)整合起來,找出組織里不同的功能區域(叫“空間域”),而且還能處理超大規模的數據,比如亞細胞級別的精細數據。
MISO算法的工作流程
MISO的工作流程(圖1)包括以下關鍵步驟:
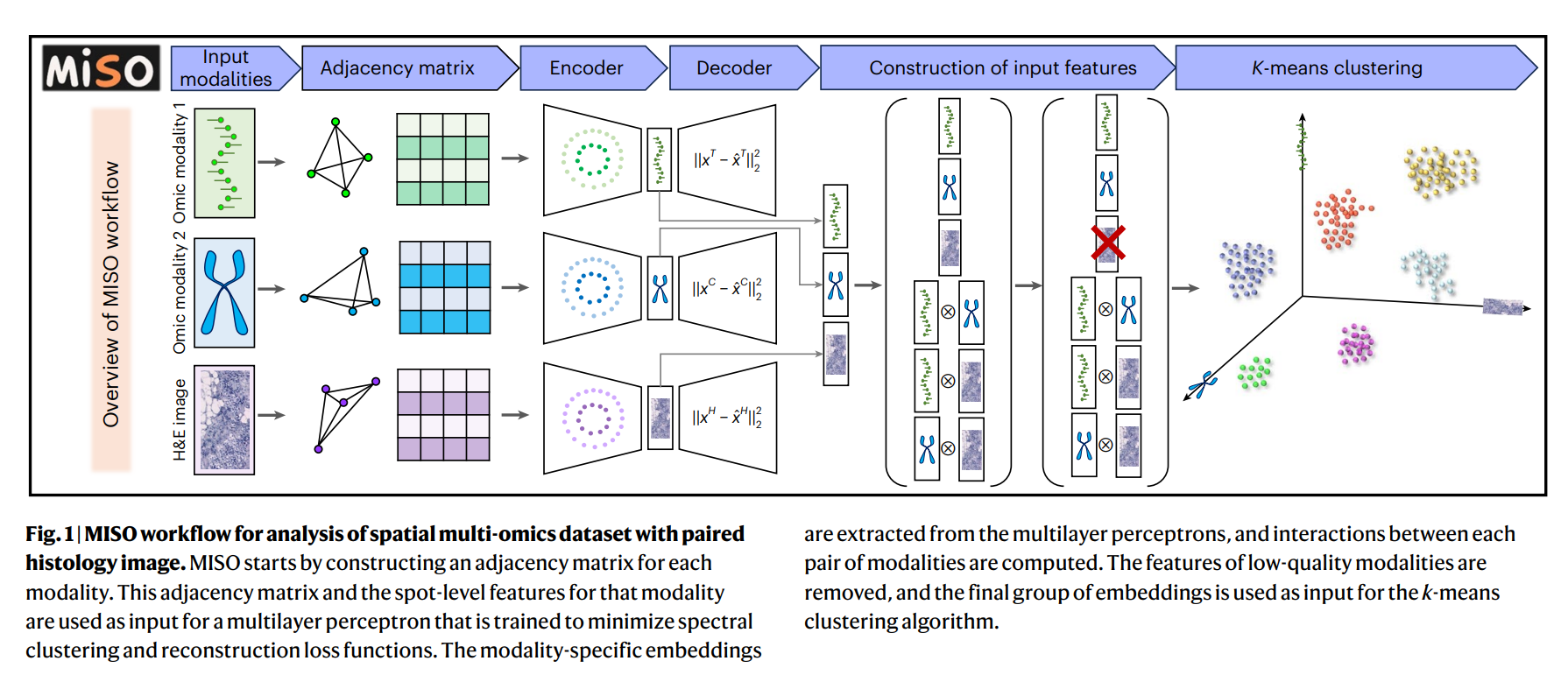
? ? ? ? ? ? ?2.構建模態特異性鄰接矩陣,捕捉數據點之間的相似性,利用神經網絡(SpectralNet啟? ? ? ? ? ? ? ? ? ? ? ?發)生成低維特征向量。
? ? ? ? ? ? ?3.對于組織學成像(如H&E染色圖像),使用預訓練的層次視覺變換器(ViT)模型提取? ? ? ? ? ? ? ? ? ? 局部和全局特征,生成每個點的576維嵌入。
? ? ? ? ? ? ? ? 對于組織學圖像,MISO用一個預訓練的視覺變換器(ViT)模型,分析圖像的局部和全局特征。比如,它會看一個細胞附近的小區域(局部特征),同時也考慮整個組織切片的模式(全局特征),最后給每個數據點生成一個576維的圖像特征向量。
特征提取(”精華提取“):
- 對每種模態使用特定的多層感知器(MLP(一種神經網絡))提取低維嵌入,基于光譜聚類和重構損失函數進行訓練。
????????“多層感知器”(MLP)的神經網絡,把高維數據(比如幾千個基因的表達量)壓縮成低維的“特征向量”,有點像把一大堆信息提煉成一個簡潔的“摘要”。
????????“光譜聚類”該方法,基于數據點之間的相似性(通過“鄰接矩陣”表示),確保提取的特征能捕捉數據的內在結構。
跨模態特征融合(”混出新味道“):
- 計算模態對之間的外積(”外積“一種數學操作),生成交互特征矩陣,捕捉模態間的相互作用(如基因表達與表觀遺傳數據的調控關系)。
? ? ? ? ?為什么還要計算模態對?因為光有單獨的特征還不夠,MISO還會看不同模態之間的“互動”。比如,基因表達和染色質開放性(ATAC-seq)可能一起決定某個細胞的功能。
2.通過主成分分析(PCA)將交互特征降維至32維,與模態特異性特征保持一致。然后和單獨模態的特征拼接在一起,形成一個包含所有信息的“綜合特征”。低質量模態過濾(”丟掉爛食材“):
- 用戶需通過視覺檢查(圖像模態)或定量指標(如基因表達的UMI計數、代謝物信號的空間分布)識別低質量模態,并決定是否納入聚類。
某些數據質量不好,比如圖像有模糊或染色不均,基因數據測序深度不夠,通過檢查將低質量的特征剔除,只保留高質量的特征和它們的交互特征。
K均值聚類(”分菜,裝盤“):
- 將模態特異性和交互特征向量拼接后,輸入K均值聚類算法,生成反映生物學特性的空間域。每個域代表一個生物學上獨特的功能區域,比如腫瘤區域、免疫細胞聚集區等。
大規模數據集優化:
- 對于大規模數據集(如MERFISH生成的數據),MISO通過稀疏鄰接矩陣(基于k近鄰,k=100)降低存儲需求,提高計算效率。
MISO的計算效率突出,可在單GPU上處理中等規模數據集(1,000-10,000個點)在1分鐘內完成,并能擴展至包含數十萬點的超大規模數據集。
應用案例與結果
文章通過16個數據集(涵蓋小鼠、人類、斑馬魚的多種組織和模態)展示了MISO的性能,以下是主要應用案例的總結:
膀胱癌10x Visium V2數據(利用istar工具識別微米級案例)(圖2)
總結:MISO像個“顯微鏡”,能從復雜的數據里精準挖出細小的HEV結構,其他方法要么太“粗糙”,要么直接“看不見”。
- 數據集:轉錄組+組織學成像(H&E),使用iStar增強分辨率至4×4 μm2超像素。
- 目標:識別三級淋巴結構(TLS)中的高內皮小靜脈(HEV)。
- 結果:MISO精確識別TLS1和TLS2中的HEV(誤分非HEV超像素僅10個和6個),靈敏度和特異性均高于SpatialGlue(誤分289和381個)和MUSE(無法生成HEV相關聚類)。
- 評估:通過類內相關系數(ICC)比較,MISO在RNA和組織學數據的聚類一致性上優于其他方法。
胃癌10x Xenium數據(數據龐大的案例)(圖3a-e)
- 數據集:單細胞分辨率轉錄組(377個基因,696,314個細胞)+組織學成像。
- 目標:區分病理學家標注的組織結構(如低黏附性癌、侵襲性分散腫瘤細胞、淋巴聚集等)。
- 結果:MISO準確區分低黏附性癌(聚類14)和分散腫瘤細胞(聚類5),并識別小規模淋巴聚集,減少手動標注時間。MISO利用組織學特征成功區分表達相似標記基因(如ERBB2)的癌和黏膜區域。
- 優勢:由于內存限制,MUSE和SpatialGlue無法處理此大規模數據集,而MISO展現了處理能力。
總結:MISO像個“超級大腦”,在數據量大、基因信息少的情況下,依然能精準找到復雜的組織結構,而其他方法直接“卡殼”了。
? ? 3.結直腸癌10x Visium HD數據(展示MISO處理亞細胞分辨率和全轉錄組數據的超強能力)(圖3f-i)
? ? ? ? ? ? ? 1.數據集:亞細胞分辨率全轉錄組(18,085個基因,545,913個8×8 μm2區域)+組織學成像。
? ? ? ? ? ? ? 2.目標:區分肌肉、正常結腸黏膜和侵襲性癌。
? ? ? ? ? ? ? 3.結果:MISO識別侵襲性癌的多個子類,其中聚類4與SPP1+巨噬細胞共定位,提示更具侵襲性的腫瘤細胞(SPP1在聚類4中上調,log2倍變化1.06,P=1.22×10???)。
? ? ? ? ? ? ? 4.優勢:MISO通過提取組織學特征,捕捉短程和遠程依賴,優于MUSE和SpatialGlue(后者因內存限制無法分析)。
總結:MISO可以找到癌區,同時進一步將癌區分成幾個子類,并與SPP1+巨噬細胞共定位,從而找到更侵襲性的癌細胞亞型。
? 4.小鼠胚胎(E13)空間ATAC-RNA-seq數據(涉及兩種復雜的模態(轉錄組和表觀遺傳組))(圖4)
? ? ? ? ? ? ? 1.數據集:轉錄組+染色質可及性+組織學成像,聚焦前腦端腦區域。
? ? ? ? ? ? ? 2.目標:區分端腦的腦室區(VZ)、底板層和皮質層。
? ? ? ? ? ? ? 3.結果:MISO唯一區分了VZ(SOX2高表達,聚類3)、底板層(GAD2高表達,聚類1)和皮質層(TBR1高表達,聚類6),驗證了其整合復雜模態的能力。t-SNE分析顯示MISO聚類在RNA和ATAC模態上分離度高,優于MUSE和SpatialGlue。
總結:MISO能整合RNA、ATAC和圖像三種模態,捕捉復雜的空間和分子關系。
5.小鼠海馬空間轉錄組與代謝組數據(支持三模態整合)(圖5a-c)
- 數據集:轉錄組+代謝組(MALDI-MSI)+組織學成像,增強分辨率后對齊空間坐標。
- 目標:細分海馬錐體層(CA1sp、CA2sp、CA3sp)。
- 結果:MISO準確區分CA1sp、CA2sp和CA3sp(包括CA3的三個子區域),而MUSE和SpatialGlue僅區分CA1sp和CA3sp。MERFISH數據驗證了MISO在CA2sp/CA3sp邊界的精確性。
總結:MISO像個“精密雕刻師”,能把海馬的細微結構雕得清清楚楚,其他方法只能看到“大輪廓”。
MISO能整合三種模態(轉錄組、代謝組、圖像),捕捉更細致的結構。
6.人類扁桃體空間基因與蛋白表達數據(展示了MISO處理三種模態(轉錄組、蛋白質組、組織圖像)的能力)(圖5d-h)
- 數據集:轉錄組+蛋白質組(35個蛋白)+組織學成像。
- 目標:定位生發中心。
- 結果:MISO在所有模態組合下的F1分數最高(0.90,三模態;0.86-0.88,雙模態),優于MUSE(最高0.87)和SpatialGlue(最高0.83)。
總結:MISO能處理三種模態(轉錄組、蛋白質組、組織圖像)的能力,尤其是在免疫組織中定位關鍵結構。MISO像個“免疫系統GPS”,能精準導航到生發中心這個“免疫大本營”,而其他方法可能會“迷路”。
其他應用(擴展數據圖2-10,補充圖2-5)
1. 小鼠前腦空間轉錄組數據(擴展數據圖2)
- 數據集:10x Visium小鼠前腦(前部冠狀切片),包含轉錄組和H&E圖像。
- 目標:根據Allen Brain Atlas的標注,區分前腦的皮質層和其他結構。
- MISO的表現:
- MISO的聚類結果與Allen Brain Atlas的皮質層標注高度一致,準確區分了不同腦區(圖2b)。
- 它的ICC(類內相關系數)在RNA和圖像模態上都高于MUSE和SpatialGlue(圖2c),說明聚類更一致。
- 即使圖像有偽影(圖2e),MISO依然通過RNA和交互特征保持了高精度。
- 對比:SpatialGlue對模態權重敏感,調整權重后結果不穩定(圖2b);MUSE受圖像偽影影響,聚類質量較低。
- 意義:MISO像個“腦區規劃師”,能在圖像質量不完美時,依然精準劃分大腦區域,對研究腦功能和疾病(如癲癇)很有用。
2. 人類乳腺癌空間轉錄組數據(擴展數據圖3)
- 數據集:10x Visium人類乳腺癌數據,包含轉錄組和H&E圖像,病理學家標注了原位癌(DCIS)、侵襲性癌和脂肪區域。
- 目標:區分這些組織區域,找出侵襲性癌的子類。
- MISO的表現:
- MISO的聚類與病理學標注高度吻合(圖3b),甚至在侵襲性癌區域找到一個子類,UMI計數明顯低于其他子類(圖3e),可能反映癌細胞的異質性。
- t-SNE圖(圖3d)顯示MISO的聚類與基因表達模式一致,ICC也高于MUSE和SpatialGlue(圖3c)。
- SpatialGlue對圖像權重敏感,調整權重后仍無法識別脂肪區域(圖3f)。
- 意義:MISO像個“癌癥地圖繪制者”,能找到癌細胞的細微差異,幫醫生判斷腫瘤的侵襲性,指導治療。
3. 斑馬魚黑色素瘤空間轉錄組數據(擴展數據圖4)
- 數據集:10x Visium斑馬魚黑色素瘤數據,包含轉錄組和H&E圖像,但圖像有模糊偽影(圖4a)。
- 目標:在圖像質量低的情況下,區分腫瘤和周圍組織。
- MISO的表現:
- MISO識別出圖像質量低,剔除了圖像特異性特征,只用RNA和RNA×圖像交互特征,依然得到高質量聚類(圖4b)。
- MUSE受模糊偽影影響,聚類被圖像質量“帶偏”;SpatialGlue表現稍好但仍不如MISO(圖4c)。
- 意義:MISO像個“抗干擾偵探”,即使圖像模糊,也能從基因數據里挖出真相,適合處理實驗中常見的數據質量問題。
4. 小鼠嗅球空間轉錄組數據(擴展數據圖5)
- 數據集:10x Visium小鼠嗅球數據,包含轉錄組和H&E圖像,標注了嗅球的層次結構。
- 目標:區分嗅球的各層(如顆粒細胞層、外叢狀層)。
- MISO的表現:
- MISO的聚類與嗅球層次標注完美匹配(圖5a-b),ICC高于MUSE和SpatialGlue(圖5c)。
- MUSE把嗅球分成上下兩部分,沒能細化層次;SpatialGlue稍好但仍不如MISO。
- 意義:MISO像個“嗅覺解剖師”,能精準劃分嗅球的結構,對研究嗅覺處理和神經退行性疾病(如帕金森)有幫助。
5. 人類乳腺癌空間基因與蛋白表達數據(擴展數據圖8)
- 數據集:10x Visium人類乳腺癌數據,包含轉錄組、蛋白質組(3通道免疫熒光:DAPI、Vimentin、PCNA)和H&E圖像。
- 目標:整合三種模態,區分組織結構。
- MISO的表現:
- MISO在所有模態組合(RNA+圖像、RNA+蛋白、蛋白+圖像、或三者一起)下都得到高質量聚類(圖8a-d)。
- ICC顯示MISO在RNA、蛋白和圖像模態上的聚類一致性最高(圖8f),比MUSE和SpatialGlue強。
- 意義:MISO像個“多才多藝的藝術家”,能用多種“顏料”(模態)畫出組織的細致圖像,適合研究復雜癌癥微環境。
6. 小鼠腦空間轉錄組與代謝組數據(擴展數據圖9-10,補充圖5)
- 數據集:10x Visium和MALDI-MSI生成的小鼠腦冠狀切片數據,包含轉錄組、代謝組和H&E圖像。RNA質量低(圖9e),數據量大(擴展數據圖10用299,350個16×16像素偽點)。
- 目標:區分腦區(如海馬、皮質),處理低質量RNA和大數據。
- MISO的表現:
- MISO剔除了低質量RNA特異性特征,只用RNA×代謝組和RNA×圖像交互特征,依然準確區分腦區(圖9a-d,圖10a-d)。
- 在超大數據集(299,350個偽點)上,只有MISO能運行,MUSE和SpatialGlue因內存限制失敗(圖10f)。
- 意義:MISO像個“大數據處理機”,能在數據質量差、量大的情況下,依然挖出腦區的生物學結構,適合高通量組學研究。
7. 小鼠冠狀腦空間CUT&Tag-RNA-seq數據(補充圖3-4)
- 數據集:包含轉錄組和表觀遺傳標記(H3K27AC、H3K27ME3)的CUT&Tag數據,以及H&E圖像。
- 目標:區分腦區,驗證MISO在表觀遺傳數據上的表現。
- MISO的表現:
- MISO準確區分了腦區的功能區域,ICC高于MUSE和SpatialGlue,展現了整合表觀遺傳和轉錄組的能力。
- 意義:MISO像個“表觀遺傳解碼器”,能揭示基因調控和表達的空間關系,對研究腦發育和疾病(如癲癇)有幫助。
MISO的優點與局限性
優點:
- 多模態整合:支持任意數量的模態(轉錄組、蛋白質組、表觀遺傳組、代謝組、組織學成像等)。
- 計算效率:快速處理中小規模數據集,稀疏矩陣優化支持大規模數據。MISO能在1分鐘內處理1,000-10,000個點的數據集,用稀疏矩陣還能搞定幾十萬個點的大數據集。例如胃癌的那個Xenium數據集(69.6萬個細胞)
- 魯棒性:通過過濾低質量模態和最小化超參數調整,適應真實世界中的數據挑戰。MISO能處理低質量數據,比如模糊的圖像或低UMI計數的基因數據。它讓用戶決定是否剔除低質量模態,還通過交互特征保留部分信息。
- 高精度:在多種生物學結構識別中優于MUSE和SpatialGlue,尤其在細粒度結構(如HEV、CA2sp)上表現突出。
局限性:
- 圖像模態限制(僅H&E):目前僅支持H&E染色圖像的特征提取,不適用于其他成像類型(如三色法、免疫熒光、DAPI)。
- 無監督性:需用戶解釋聚類結果與生物學結構的關聯。比如“這個聚類對應的是腫瘤還是正常組織?”這可能需要病理學家的專業知識。
- 模態貢獻評估:無法直接量化各模態對聚類的貢獻或自動檢測低質量模態,需用戶手動評估。
跟其他方法的對比
- MUSE:擅長轉錄組+圖像,但不支持代謝組或蛋白質組,內存需求高,處理大數據時容易“翻車”。
- SpatialGlue:能處理兩個組學模態,但不支持圖像,超參數調整麻煩,對低質量模態敏感。
- MISO:綜合能力最強,支持多模態,超參數少,內存優化好,適合大數據和復雜場景。
MISO如何處理H&E圖像特征
H&E圖像(組織學切片染色圖像)是空間組學的重要模態,提供了組織的空間結構信息。MISO的圖像特征提取過程很聰明,像是把一張復雜的“組織畫”拆解成數字化的“特征拼圖”。具體步驟如下(論文第10頁,補充圖6有詳細示意圖):
- 用層次視覺變換器(ViT)模型:
- MISO用了一個預訓練的‘’Hierarchical Image Pyramid Transformer (HIPT)‘’模型,訓練過10,000多張全景切片圖像,能捕捉組織的局部和全局特征。
- 比喻:這就像一個“超級畫師”,既能看清畫上的小細節(細胞形態),也能把握整幅畫的布局(組織結構)。
- 分層提取特征:
- 局部特征:HIPT把大圖像切成256×256像素的小塊,每個小塊用一個局部ViT模型處理,生成一個384維的“補丁嵌入”(patch embedding),描述小塊的細節。
- 全局特征:把這些256×256小塊的特征(CLS token,192維)輸入一個全局ViT模型,分析它們在更大區域(4096×4096像素)里的關系,生成捕捉遠程依賴的特征。
- 每個16×16像素的小補丁最終得到一個576維嵌入(384維局部+192維全局)。
- 生成點級特征:
? ? ? ? ? ? ?空間組學數據點(比如Visium的55微米點或Xenium的單細胞)通常覆蓋多個16×16像素補丁。MISO把一個數據點覆蓋的所有補丁的576維嵌入取平均,生成這個點的圖像特征。
這就像把一張畫上的每個區域(數據點)用“顏色、紋理、結構”的綜合描述概括出來。
為什么這很重要?
- 在結直腸癌案例中,MISO用圖像特征捕捉癌細胞和SPP1+巨噬細胞的空間關系,區分了更侵襲性的癌區。
- 在扁桃體案例中,圖像特征幫MISO識別生發中心的組織學特征(比如PCNA高表達區的結構),提高了F1分數。
- 這種局部+全局的特征提取讓MISO能看到組織里的“微觀細節”和“宏觀格局”,比只看基因數據的MUSE或SpatialGlue更全面。
協議的全面解析:理論、機制與實踐局限性)


![[SC]SystemC 常見的編譯/語法錯誤與解法(三)](http://pic.xiahunao.cn/[SC]SystemC 常見的編譯/語法錯誤與解法(三))



)



)







