歡迎關注我的CSDN:https://spike.blog.csdn.net/
本文地址:https://blog.csdn.net/caroline_wendy/article/details/136384544
基于冷凍電鏡單顆粒圖像重構蛋白質三維結構,利用冷凍電鏡技術測定生物大分子結構的方法。原理是從冷凍電鏡獲得大量同一種蛋白質分子的二維投影圖像,然后,利用三維重構算法計算出蛋白質的三維結構。這種方法的關鍵步驟是,估計每個投影圖像的投影方向,即蛋白質分子在三維空間中的取向。這是一個非凸優化問題,需要用到一些數學和計算機科學的知識。基于冷凍電鏡單顆粒圖像重構蛋白質三維結構是一種非常先進和有效的技術,可以揭示蛋白質的功能、結構和相互作用,對于生物醫學研究和應用有著重要的意義。可以使用 cryoSPARC 軟件,基于單顆粒圖像從頭重構蛋白質三維結構。
下載數據 10028 Ribosome (核糖體),參考 冷凍電鏡 EMPIAR 數據集的下載過程
核糖體 (Ribosome) 是細胞中的一種細胞器,主要由核糖體RNA和核糖體蛋白質組成,由一大一小兩個亞基結合形成。核糖體的主要功能是根據信使RNA的指示,利用轉運RNA運送的氨基酸,合成蛋白質。核糖體在原核生物和真核生物的細胞中都存在,但是結構和大小有所不同。核糖體也是一種核酶,具有催化能力。
數據樣式,shiny_2sets.star 是 數據描述文件:
├── 10028
│ └── data
│ └── Particles
│ ├── MRC_0601
│ ├── MRC_1901
│ └── shiny_2sets.star
├── biosoft
│ └── aspera
│ └── ibm-aspera-connect_4.1.0.46-linux_x86_64.sh
安裝 CryoSparc 軟件,參考 冷凍電鏡 CryoSPARC 軟件的安裝與環境配置 (CryoEM)
整體的流程如下:
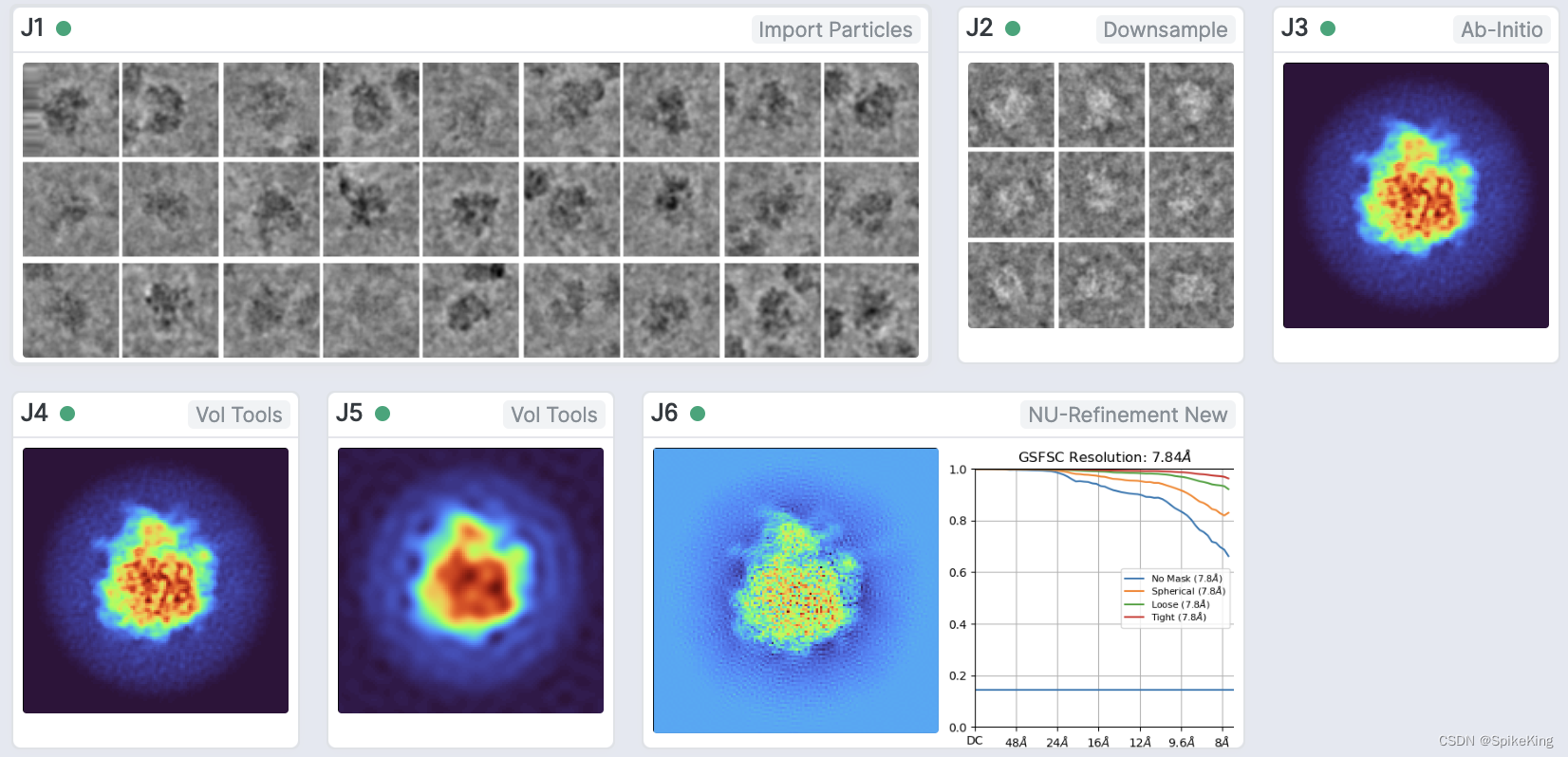
其中:
- Job1:加載數據集
- Job2:降采樣數據集
- Job3:Ab-Initio蛋白質結構重構
- Job4:蛋白質手性顛倒
- Job5:降低蛋白質分辨率
- Job6:蛋白質結構Refinement
Job1 和 Job2 部分參考:冷凍電鏡 CryoSPARC 單顆粒圖像數據集構建
Ab-Initio 蛋白質結構重構
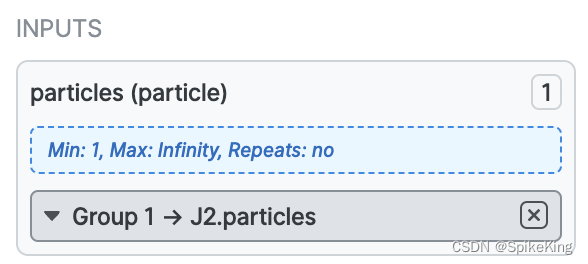
模塊輸入是 Job2 (降采樣數據集) 的輸出,即點擊空格,將 Job2 的輸出文件,直接拖入 Ab-Initio Reconstruction Job 的輸入,即可,參數可以選擇默認。
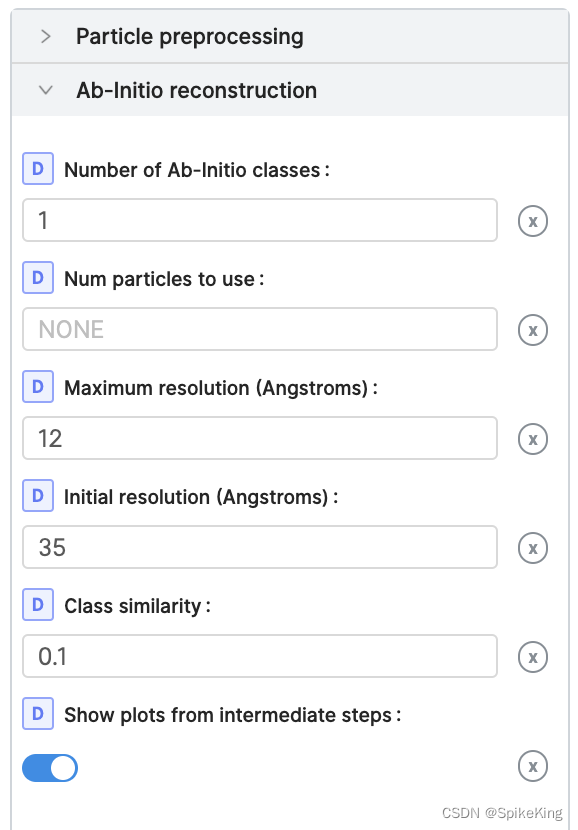
- Maximum resolution: 最大分辨率,默認是12,支持調整,越小精度越高,也與數據集相關。
- Initial resolution:初始分辨率,默認是35。
模塊輸入,配置完成如下:
其他參數如下,默認即可:
運行日志,注意,重構耗時 1989.719s ,如下:
[CPU: 898.9 MB] ----------- Iteration 1159 (epoch 2.708). radwn 

)






)





 靜態合批步驟與所有注意事項\游戲運行時使用代碼啟動靜態合批)

)
與激活函數)
