

GO和KEGG富集分析是我們在篩選出差異表達基因之后,都會去做的套路性分析。然鵝……我相信,總有那么一些“倒霉孩子”會遇到跟我一樣的窘境吧,好不容易篩選出來的差異基因,嘗試了DAVID(https://david.ncifcrf.gov/)、Metascape(https://metascape.org/)、g:Profiler(https://biit.cs.ut.ee/gprofiler/)等之后,仍然富集不出來通路,這真是個令人悲傷的故事嘞~

就在我萬念俱灰的時候,我神奇的發現了一個網站,居然富集到了好幾條通路哦,心情頓時開朗許多~~

現在,就把這個最強大的基因富集分析在線工具分享給大家啦~揭開它神秘面紗的時候到了,它就是——KOBAS!!!
KOBAS(KEGG Orthology Based Annotation System),網址:http://kobas.cbi.pku.edu.cn,是由北京大學魏文麗課題組開發的數據庫,主要功能是用于基因/蛋白質功能注釋和功能富集。隨著數據量不斷增加,KOBAS至今為止共經歷了3次升級,除了1.0版本的KEGG代謝通路之外,2.0版本增加了PID Curated,PID BioCarta,PID Reactome,BioCyc,Reactome和Panther等數據庫,除此之外還增加了疾病的查詢,使得KOBAS的功能更加強大。而3.0版本更是增加了lncRNA的鑒定、注釋以及富集功能,還建立GSEA,GSA和 PADOG等功能。
KOBAS的功能最主要分為兩個部分,Annotate(注釋)和Enrichment(富集)。
接下來我們就以“Annotate”為例來體驗看看如何利用KOBAS做GO和KEGG的富集分析吧!
點擊“Annotate”選項,進入注釋界面。
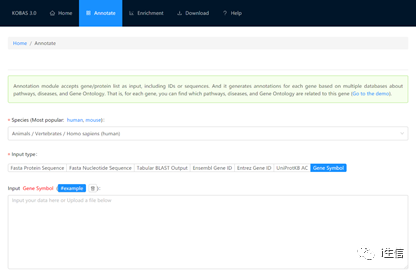
“Species”為物種,可根據自己測序組織來源進行選擇,如人,大鼠,小鼠等。
“Input Type”為輸入格式/類型,支持如圖Fasta Protein Sequence,Gene Symbol等七種格式,可根據自己的輸入數據進行選擇。
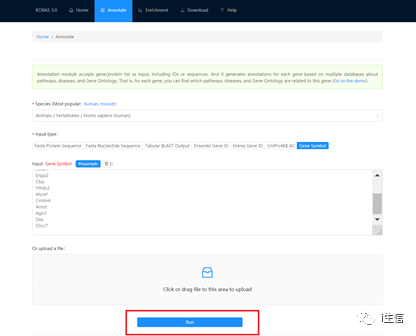
輸入數據后,點擊“Run”進行提交,等待數秒即可出結果。
得到注釋結果,如下圖所示,點擊“Detail”可查看基因注釋的Pathway通路、疾病及GO功能分類。注釋結果下方還提供鏈接進行結果下載哦。
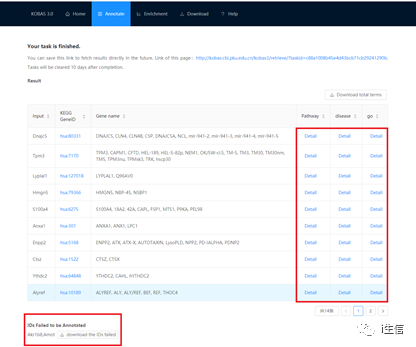
點擊第二列“KEGG Gene ID”可以直接鏈接到KEGG數據庫查看具體信息。
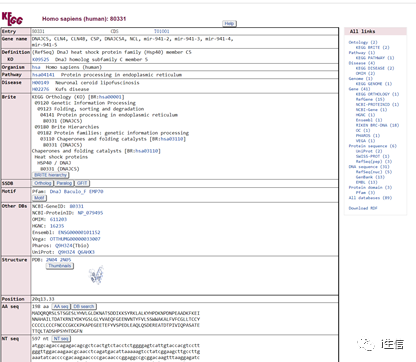
點擊第三列“Disease”中“Detail”注釋結果如下所示,可具體了解與差異表達基因相關的疾病信息哦~
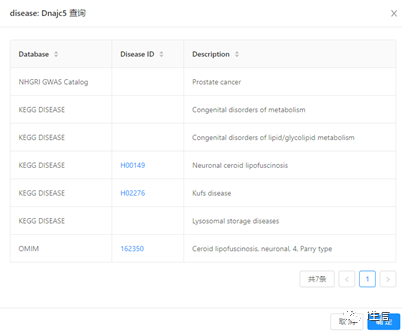
“Gene-list Enrichment”部分,“Input Type”, “Species”及“Input”與注釋時基本一致。
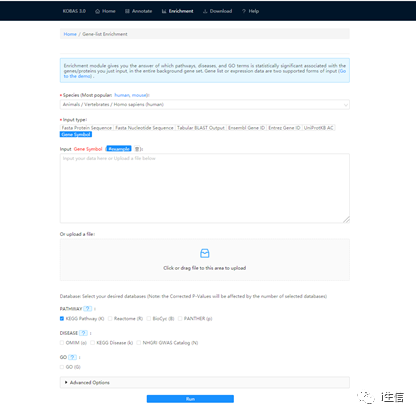
“Datebase”中可選擇自己感興趣的數據庫進行分析。
“Advance Options”中可自行選擇參數,包括背景基因、統計學方法及校正方法等。
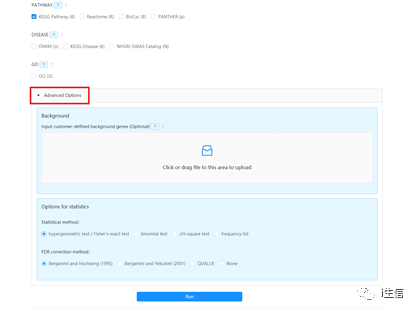
與“Annotate”一樣,設置好所有參數后,點擊“Run”,等待數秒即可得到分析結果哦,且結果都可進行下載,方便又快捷。
此外,KOBAS除在線使用外,還可將下載到本地,具體可點擊官網菜單欄“Download”進行詳細了解哦,在此不做詳細介紹啦。
總之,KOBAS不僅僅可以得到GO和KEGG富集分析結果,還可以了解與之相關的疾病信息。學習了這么多,趕緊把你手中篩選到的差異基因操作分析起來吧,說不定馬上就會得到意想不到的結果哦~~
注:此推文未經許可禁止轉載!
閱讀推薦:
工具篇|MNDR:遞“瓜”小能手,扒一扒ncRNA與疾病之間的那些事
工具篇 | TransCirc:一個預測分析circRNA翻譯的數據庫
工具篇|一站搞定多種形式的韋恩分析可視化,需要哪個選哪個
工具篇|最新出爐的國產m6A修飾精準預測數據庫m6A-Atlas
工具篇|神器一出,作圖無憂:一個完爆R語言的作圖網站




















