歡迎關注我的CSDN:https://spike.blog.csdn.net/
本文地址:https://spike.blog.csdn.net/article/details/132357976

Paper: EigenFold: Generative Protein Structure Prediction with Diffusion Models
EigenFold 是用于蛋白質結構預測的擴散生成模型(即,已知序列 至 結構分布)。基于諧波擴散,將鍵約束納入擴散建模框架,并且產生一個級聯分辨率的生成過程。
- 擴散生成模型 (Diffusion Generative Model):利用隨機擴散過程,生成數據樣本的機器學習模型。
- 諧波擴散 (Harmonic Diffusion):考慮諧波勢能對于擴散過程的影響的數學模型。
- 鍵約束 (Bond Constraints):限制蛋白質中原子間距離和角度變化范圍的物理條件。
- 級聯分辨率 (Cascading-Resolution) :從粗糙到精細,逐步提高生成結果質量的方法。
- OmegaFold 嵌入向量(OmegaFold Embeddings):由 OmegaFold 模型產生的,表示蛋白質序列特征的向量。
關于 EigenFold,即:
We define a diffusion process that models the structure as a system of harmonic oscillators and which naturally induces a cascading-resolution generative process along the eigenmodes of the system.
擴散過程,即將結構模型化為諧振子 (Harmonic Oscillators) 系統,該過程自然地沿著系統的本征模式 (Eigenmodes),產生級聯分辨率的生成過程。
EigenFold 算法重點:
- 蛋白質結構生成的新方法: 基于擴散模型的生成式模型,可以從給定的蛋白質序列生成一組可能的結構。該模型利用 OmegaFold 的預訓練嵌入和得分網絡來學習蛋白質結構的概率分布。
- 諧波擴散過程:定義新的擴散過程,將蛋白質結構建模為一系列諧振子,其勢能為相鄰殘基之間的距離的二次函數。該過程可以保證采樣的結構滿足化學約束,并且可以沿著系統的本征模式進行投影,實現逐步精細化的生成過程。
- 得分網絡架構:使用基于 E3NN 的圖神經網絡作為得分網絡,輸入為殘基坐標和 OmegaFold 嵌入向量,輸出為梯度向量。該網絡具有 SE(3) 等變性,保證最終模型密度也具有 SE(3) 不變性。
EigenFold GitHub: https://github.com/bjing2016/EigenFold
1. 結構預測
準備 new.csv 文件,預測 7skh.B 的結構,即:
# with columns name, seqres (see provided splits for examples) and run
name,valid_alphas,seq,head,resolution,deposition_date,release_date,structure_method,seqres,seqlen
7skh.B.pdb,220,NAPVFQQPHYEVVLDEGPDTINTSLITVQALDGTVTYAIVAGNIINTFRINKHTGVITAAKELDYEISHGRYTLIVTATDQCPILSHRLTSTTTVLVNVNDINDNVPTFPRDYEGPFDVTEGQPGPRVWTFLAHDRDSGPNGQVEYSVVDGDPLGEFVISPVEGVLRVRKDVELDRETIAFYNLTICARDRGVPPLSSTMLVGIRVLDINDNLEHHHHHH,cell adhesion,2.27,2021-10-20,2022-10-26,x-ray diffraction,MNAPVFQQPHYEVVLDEGPDTINTSLITVQALDLDEGPNGTVTYAIVAGNIINTFRINKHTGVITAAKELDYEISHGRYTLIVTATDQCPILSHRLTSTTTVLVNVNDINDNVPTFPRDYEGPFDVTEGQPGPRVWTFLAHDRDSGPNGQVEYSVVDGDPLGEFVISPVEGVLRVRKDVELDRETIAFYNLTICARDRGVPPLSSTMLVGIRVLDINDNLEHHHHHH,227
運行命令:
python make_embeddings.py --out_dir ./embeddings --splits mydata/new.csv
python inference.py --model_dir ./pretrained_model --ckpt epoch_7.pt --pdb_dir ./structures --embeddings_dir ./embeddings --embeddings_key name --elbo --num_samples 5 --alpha 1 --beta 3 --elbo_step 0.2 --splits mydata/new.csv
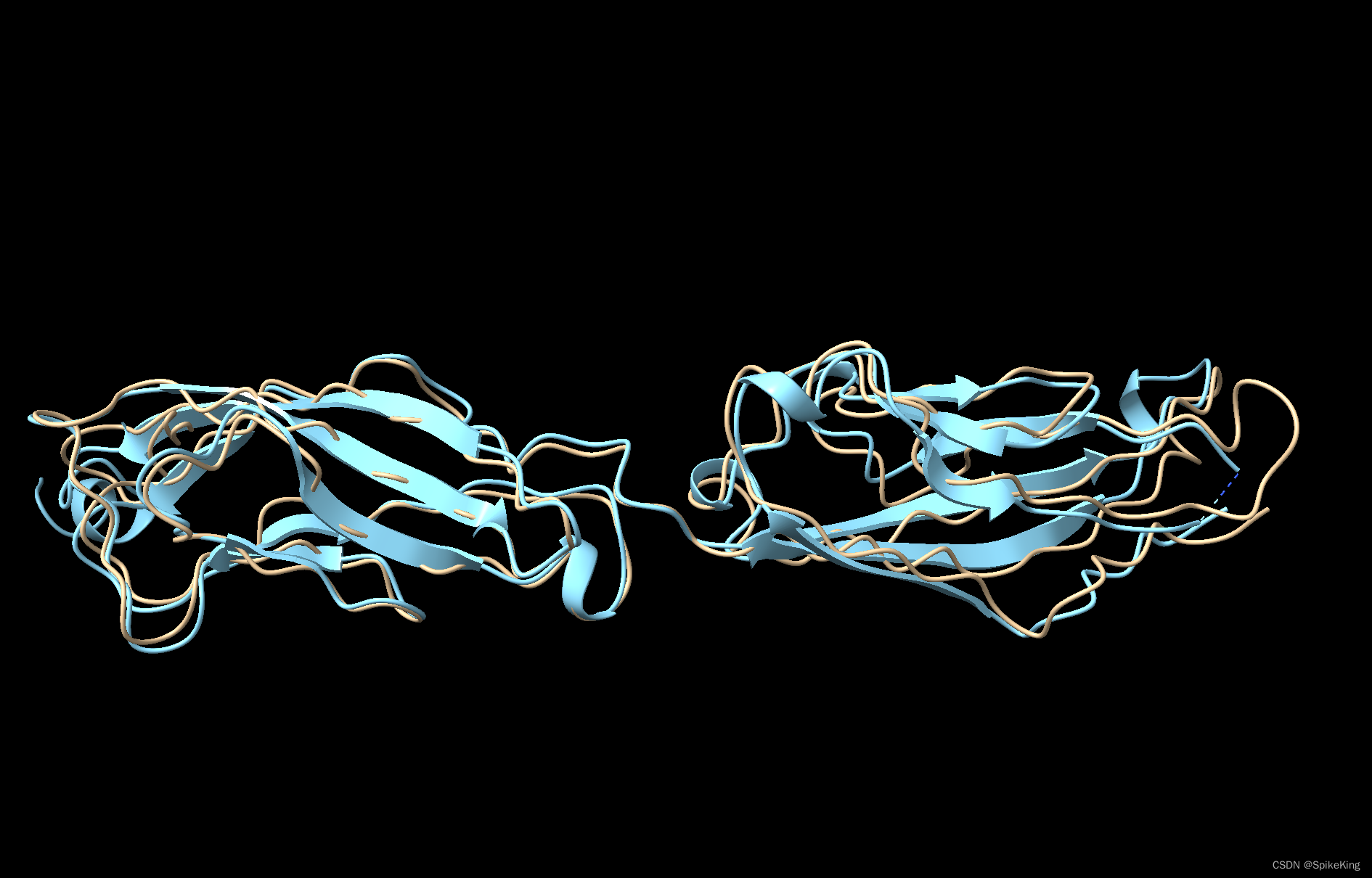
預測的蛋白質結構,如下:
- EigenFold 算法只能預測 CA 骨架,其余需要填充。
- 黃色是 EigenFold 的預測結構,藍色是真實的 PDB 結構 (7skh.B)。
即:
2. 環境配置
下載 GitHub 工程:
git clone git@github.com:bjing2016/EigenFold.git
2.1 配置 Docker 環境
構建 Docker 環境:
nvidia-docker run -it --name eigenfold-[your name] -v [nfs path]:[nfs path] af2:v1.02
預先配置 Docker 環境中的 conda 源 與 pip 源,加速下載過程,參考 開源可訓練的蛋白質結構預測框架 OpenFold 的環境配置
如果安裝錯誤,清空 conda 環境,建議使用 rsync 快速刪除,即:
mkdir tmp
rsync -a --delete tmp/ /opt/conda/envs/eigenfold
rm -rf /opt/conda/envs/eigenfold
配置 conda 環境,即:
# 安裝 conda 環境
conda create -n eigenfold python=3.8
conda activate eigenfold
2.2 配置 PyTorch 系列包
安裝 PyTorch,建議使用 conda 安裝,而不是 pip 安裝,參考 Installing Previous Versions of PyTorch 即:
# pip 安裝異常,建議使用 conda 安裝。
# pip install torch==1.11.0+cu113 torchvision==0.12.0+cu113 torchaudio==0.11.0 --extra-index-url https://download.pytorch.org/whl/cu113
conda install pytorch==1.11.0 torchvision==0.12.0 torchaudio==0.11.0 cudatoolkit=11.3 -c pytorch
預先測試 PyTorch 是否安裝成功,即:
pythonimport torch
print(torch.__version__) # 1.11.0
print(torch.cuda.is_available()) # True
再安裝 PyTorch 相關包,一共 5 個包,即 torch-scatter、torch-sparse、torch-cluster、torch-spline-conv、torch-geometric,建議逐個安裝,排查問題,即:
pip install torch-scatter -f https://data.pyg.org/whl/torch-1.11.0+cu113.html
pip install torch-sparse -f https://data.pyg.org/whl/torch-1.11.0+cu113.html
pip install torch-cluster -f https://data.pyg.org/whl/torch-1.11.0+cu113.html
pip install torch-spline-conv -f https://data.pyg.org/whl/torch-1.11.0+cu113.html
pip install torch-geometric -f https://data.pyg.org/whl/torch-1.11.0+cu113.html
安裝其他依賴包:
pip install e3nn pyyaml wandb biopython matplotlib pandas
2.3 配置 OmegaFold 依賴
安裝 OmegaFold 依賴,即:
# 調用時,需要在 EigenFold 的根目錄下。
wget https://helixon.s3.amazonaws.com/release1.pt
git clone https://github.com/bjing2016/OmegaFold
pip install --no-deps -e OmegaFold
注意需要預先下載 OmegaFold 的模型
release1.pt,大約 3 個 G左右。
OmegaFold GitHub: OmegaFold
This command will download the weight from https://helixon.s3.amazonaws.com/release1.pt to
~/.cache/omegafold_ckpt/model.ptand load the model
cd EigenFold
bypy info
bypy downfile /huggingface/eigenfold/omegafold-release1.pt model.pt
2.4 配置 TMScore 與 LDDT
安裝 TMScore 與 LDDT,即:
mkdir /opt/bin
cd ~/binwget https://openstructure.org/static/lddt-linux.zip
unzip lddt-linux.zip
cp lddt-linux/lddt .
./lddt # 測試wget https://zhanggroup.org/TM-score/TMscore.cpp
g++ -static -O3 -ffast-math -lm -o TMscore TMscore.cpp
./TMscore # 測試export PATH="/opt/bin/:$PATH"
2.6 上傳 Docker
提交 docker image,設置標簽 (tag),以及上傳 docker 至服務器,即:
# 提交 Tag
docker ps -l
docker commit [container id] eigenfold:v1.0# 準備遠程 Tag
docker tag eigenfold:v1.0 harbor.[ip].com/[your name]/eigenfold:v1.0
docker images | grep "eigenfold"# 推送至遠程
docker push harbor.[ip].com/[your name]/eigenfold:v1.0
# 從遠程拉取
docker pull harbor.[ip].com/[your name]/eigenfold:v1.0# 或者保存至本地
docker save eigenfold:v1.0 | gzip > eigenfold_v1_0.tar.gz
# 加載已保存的 docker image
docker image load -i eigenfold_v1_0.tar.gz
docker images | grep "eigenfold"
BugFix
Bug1: torch_sparse 版本不兼容問題。
RuntimeError:
object has no attribute sparse_csc_tensor:File "/opt/conda/envs/eigenfold/lib/python3.8/site-packages/torch_sparse/tensor.py", line 520value = torch.ones(self.nnz(), dtype=dtype, device=self.device())return torch.sparse_csc_tensor(colptr, row, value, self.sizes())~~~~~~~~~~~~~~~~~~~~~~~ <--- HERE
參考: torch has no attribute sparse_csr_tensor
將 torch-sparse 降級至 0.6.14 版本,即可:
conda list torch-sparse
# packages in environment at /opt/conda/envs/eigenfold:
#
# Name Version Build Channel
torch-sparse 0.6.17 pypi_0 pypipip install torch-sparse==0.6.14 -f https://data.pyg.org/whl/torch-1.11.0+cu113.html
Bug2: Python 3.9 新特性不兼容問題
TypeError: unsupported operand type(s) for |: 'dict' and 'dict'
原因:What’s New In Python 3.9
方案1是升級至 Python3.9 版本,方案2是修改源碼,位于EigenFold/utils/pdb.py,即:
# d[key] = {'CA': 'C'} | {key: val['symbol'] for key, val in atoms.items() if val['symbol'] != 'H' and key != 'CA'}
dict1 = {'CA': 'C'}
dict2 = {key: val['symbol'] for key, val in atoms.items() if val['symbol'] != 'H' and key != 'CA'}
d[key] = {**dict1, **dict2}
其余參考:
- Linux 下刪除大量文件效率對比,看誰刪的快!



)








)


)



