實驗篇——亞細胞定位
文章目錄
- 前言
- 一、亞細胞定位的在線網站
- 1. UniProt
- 2. WoLFPSORT
- 3. BUSCA
- 4. TargetP-2.0
- 二、代碼實現
- 1. 基于UniProt(不會)
- 2. 基于WoLFPSORT
- 后續(已完善,有關代碼放置于[python爬蟲學習(一)](https://blog.csdn.net/2301_78630677/article/details/132241087)):
- 總結
前言
有關亞細胞定位的詳細信息,請參考另一篇文章:
理化性質與亞細胞定位
一、亞細胞定位的在線網站
1. UniProt
網址:https://www.uniprot.org/
在這個網站中有一個關鍵的概念——ID映射(若AA序列文件不是在該官網中下載的,而是從外界導入的,那么要先將AA序列的ID轉變為UniProt ID)
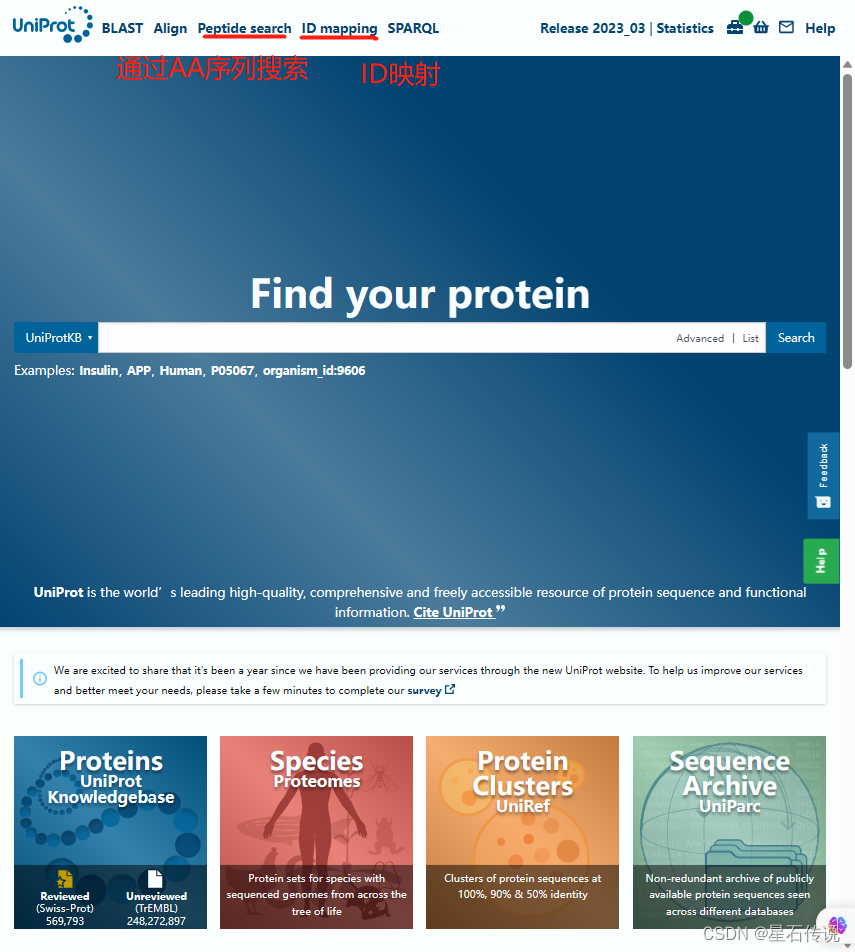
注意:首先要清楚待轉換的AA序列的ID標識符來源于哪個數據庫
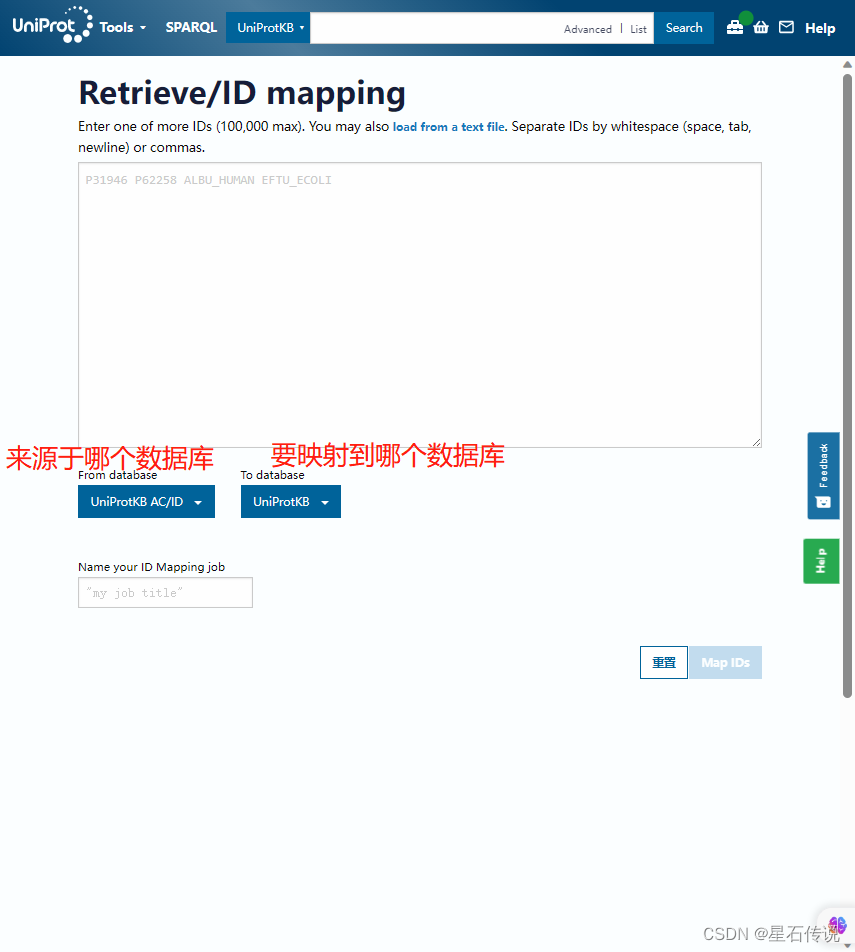
我輸入了“sp|Q9FIK7.1|AACT1_ARATH”,這是某個AA序列的ID,得到如下結果,可知道它對應的UniProt ID 為 “Q8S4Y1”
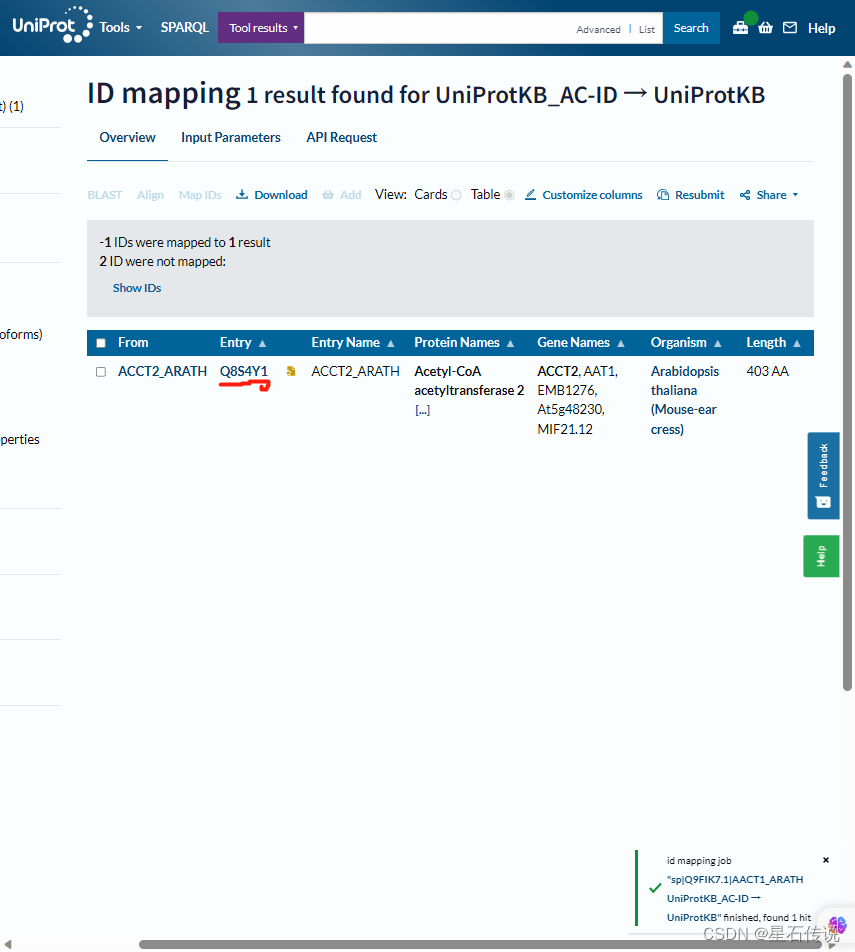
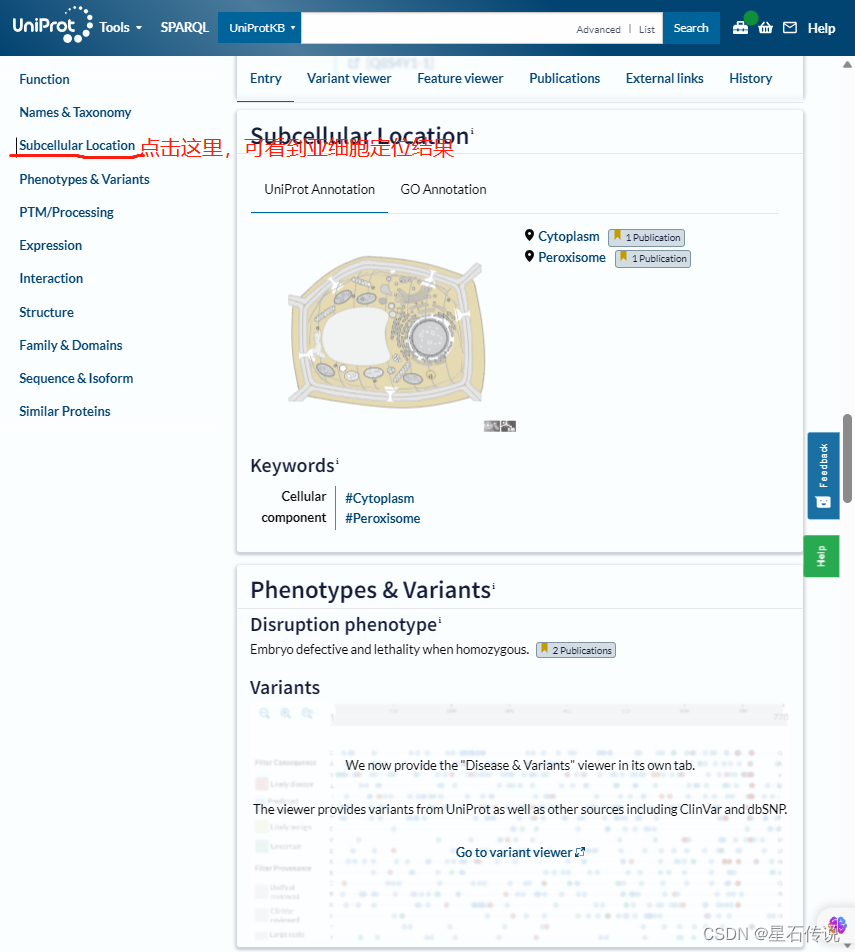
然后在搜索欄中輸入該名稱,得到
2. WoLFPSORT
網址:https://wolfpsort.hgc.jp/
這個網站可以批量處理小量的AA序列,允許的大小(200K),根據實際情況,一般可以容納幾百到幾千個氨基酸序列。
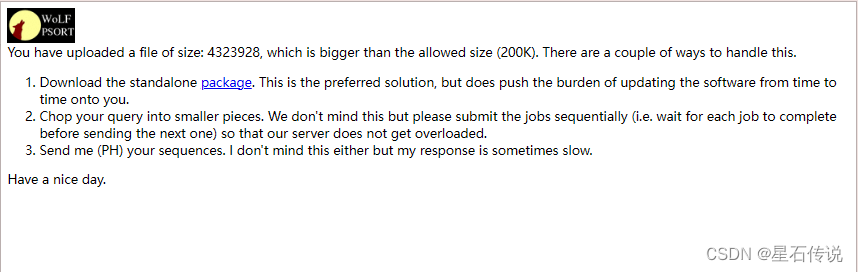
結果查看:

示例:
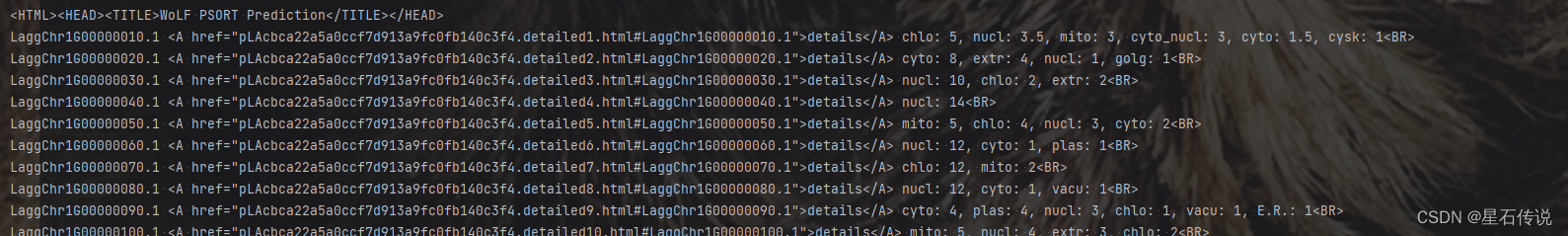
LaggChr1G00000010.1 details chlo: 5, nucl: 3.5, mito: 3, cyto_nucl: 3, cyto: 1.5, cysk: 1
LaggChr1G00000010.1這個蛋白質的亞細胞定位信息如下:
葉綠體(chlo)得分:5
細胞核(nucl)得分:3.5
線粒體(mito)得分:3
細胞質-細胞核(cyto-nucl)得分:3
細胞質(cyto)得分:1.5
細胞骨架(cysk)得分:1
這些得分表示蛋白質在各個亞細胞定位的可能性,較高的得分表示較高的概率。
他們是按得分排列的,故取第一個就行。
3. BUSCA
url: http://busca.biocomp.unibo.it/
最多可以輸入500個序列
可以下載結果表格(還挺方便)
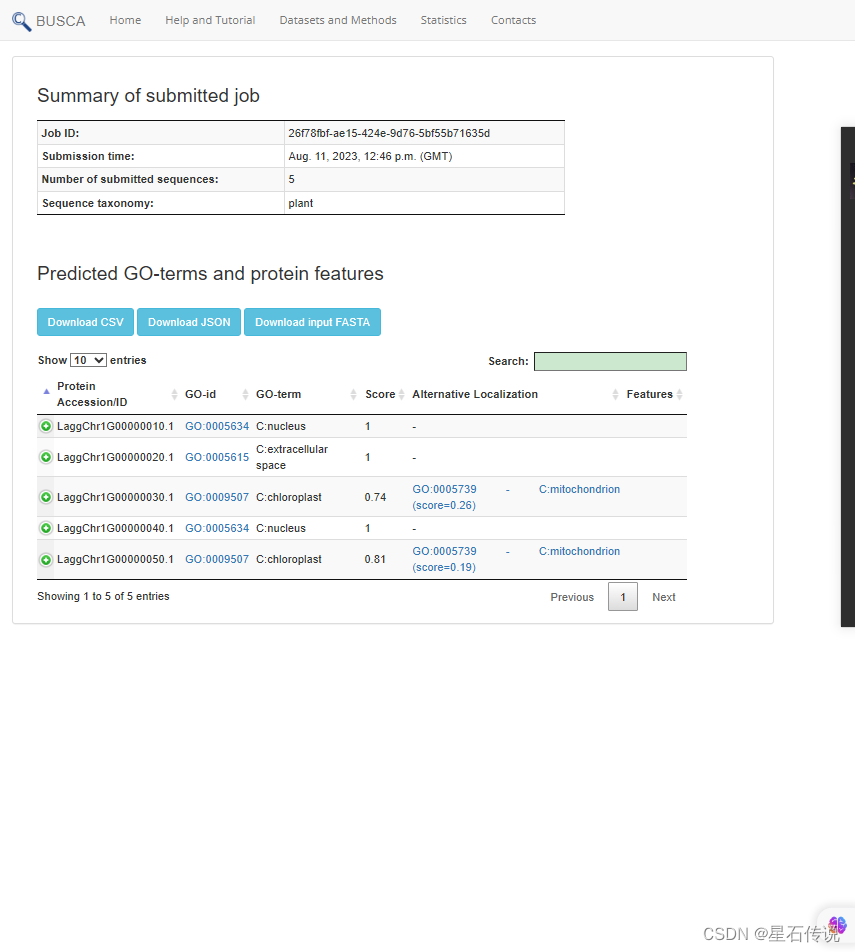
結果查看:
4. TargetP-2.0
TargetP-2.0
url = http://www.cbs.dtu.dk/services/TargetP/
我看了一下,它介紹中說能處理5000個AA序列,但是要得到結果文件是要下載這個軟件,在網頁上看不到結果(我沒下載,因為下載它好像要填一些信息什么的)
二、代碼實現
1. 基于UniProt(不會)
推薦:這是一篇有關于用R語言實現“根據uniprot ID 批量檢測基因的亞細胞定位“
這篇文章是在已知道Uniprot ID的情況下實現的爬取
我最開始也是看的這篇文章,所以想要基于uniProt官網來通過爬蟲爬取。但是我弄了好久才有點搞懂這個網站(它的功能太多了,太繁雜)。最主要是那個ID映射(我完全卡在這第一步了),因為我并不知道我的AA序列的ID來自哪個數據庫 ,而且全是英文,就相當于我用之前還要了解好多數據庫。我也看了許多關于這個官網介紹的教程,也是有點…
2. 基于WoLFPSORT
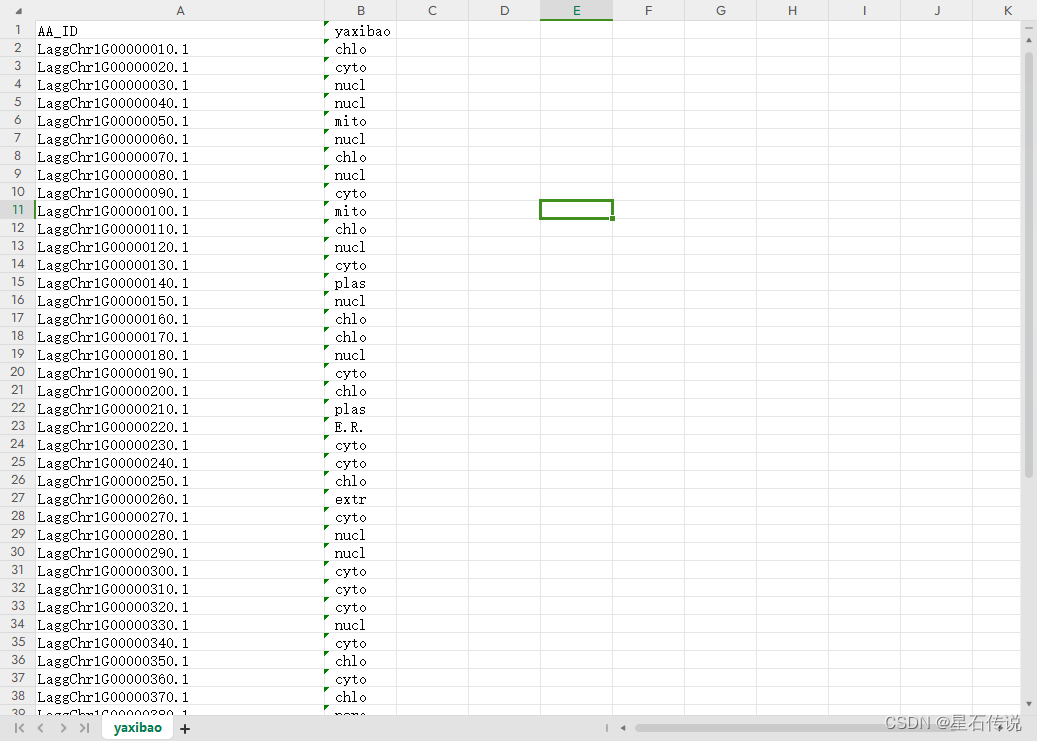
主要是對結果的整理
從前文可知,它返回的結果是一堆的,要想從中提取出來蛋白質的亞細胞定位,可以用代碼實現
import requests
url = "https://wolfpsort.hgc.jp/results/pLAcbca22a5a0ccf7d913a9fc0fb140c3f4.html"
r = requests.get(url)
print(r.status_code)
# print(r.encoding)
text = r.text
# print(text)
lines = text.split("<BR>")
AA_ID_list = []
yaxibao_list =[]
for i in lines:if "details" in i:AA_ID = i.split("<A")[0].strip().split()[-1]yaxibao = i.split("details")[1].strip().split()[1][:-1]AA_ID_list.append(AA_ID)yaxibao_list.append(yaxibao)
with open("yaxibao.csv","w",encoding="utf-8") as f:f.write("AA_ID, yaxibao\n") # 寫入列名for j in range(len(AA_ID_list)):f.write(f"{AA_ID_list[j]}, {yaxibao_list[j]}\n")
text:
yaxibao.csv:
后續(已完善,有關代碼放置于python爬蟲學習(一)):
因為在WoLF PSORT官網中一次提交的數據大小最多200kb,那 我可以試著將原來幾萬kb大小的AA序列的大文件分為小文件,(之前說錯了,之前的那個劃分文件的函數是根據文件的行數劃分的,而不是AA序列的ID數。我要將大的AA序列文件劃分為小的AA序列文件,是要根據AA序列的ID劃分,不然就會導致AA的ID 與AA的序列不連貫這種情況。故我又重新修改了一下)。
所以根據估算,這個WoLF PSORT官網中一次提交的AA序列最多也是差不多500左右。與那個BUSCA網站的差不多。
若是一定要批量處理大量的AA序列,可以嘗試運用爬蟲:
(一個思路,其中結果頁面的url無法獲得)
import requests
import os
import pandas as pd
from bs4 import BeautifulSoupdef split_gene_file(source_file, output_folder, ids_per_file):os.makedirs(output_folder, exist_ok=True)current_file = Nonecount = 0with open(source_file, "r") as f:for line in f:if line.startswith(">"):count += 1if count % ids_per_file == 1:if current_file:current_file.close()output_file = f"{output_folder}/gene_file_{count // ids_per_file + 1}.csv"current_file = open(output_file, "w", encoding='utf-8')current_file.write(line)else:current_file.write(line)if current_file:current_file.close()split_gene_file("D:\yuceji\Lindera_aggregata.gene.pep", "gene1", 500)files = os.listdir("D:\python\PycharmProjects\pythonProject1\爬蟲\gene1")base_url = "https://wolfpsort.hgc.jp/"
new_url = []
for i in range(len(files)):with open(f"D:\python\PycharmProjects\pythonProject1\爬蟲\gene1\gene_file_{i + 1}.csv", "r") as f:aa_sequence = f.read()# 構建WoLFPSORT請求的數據data = {"seq": aa_sequence}# 發送POST請求到WoLFPSORT官網response = requests.post(base_url, data=data)print(response.status_code)print(response.text)# 檢查請求是否成功if response.status_code == 200:# 解析結果頁面的URLsoup = BeautifulSoup(response.content, "html.parser")result_links = soup.find_all("a", href=True)print(result_links)result_url = None# 遍歷所有的鏈接for link in result_links:href = link.get("href", "")# 判斷鏈接是否包含 "results"if "results" in href:result_url = base_url + hrefbreakif result_url:print(result_url)new_url.append(result_url)else:print("無法找到亞細胞定位結果頁面的URL")for i in range(len(new_url)):# url = "https://wolfpsort.hgc.jp/results/pLAcbca22a5a0ccf7d913a9fc0fb140c3f4.html"r = requests.get(new_url[i])print(r.status_code)# print(r.encoding)text = r.text# print(text)lines = text.split("<BR>")AA_ID_list = []yaxibao_list = []for i in lines:if "details" in i:AA_ID = i.split("<A")[0].strip().split()[-1]yaxibao = i.split("details")[1].strip().split()[1][:-1]AA_ID_list.append(AA_ID)yaxibao_list.append(yaxibao)with open(f"yaxiba{i}o.csv", "w", encoding="utf-8") as f:f.write("AA_ID, yaxibao\n") # 寫入列名for j in range(len(AA_ID_list)):f.write(f"{AA_ID_list[j]}, {yaxibao_list[j]}\n")這個爬蟲代碼中返回的url并不是我要的那種,例如:https://wolfpsort.hgc.jp/results/pLA2dbb41dafad4afb342b5000abcb263b1.html
而是:(如圖所示)
我點進這個鏈接是這樣的:

我也不知道為什么,只能等我再學學爬蟲,希望之后能解決這個問題吧!(當然也希望有大佬能幫忙指教一下)
我還看了看結果頁面的源代碼(HTML語言):
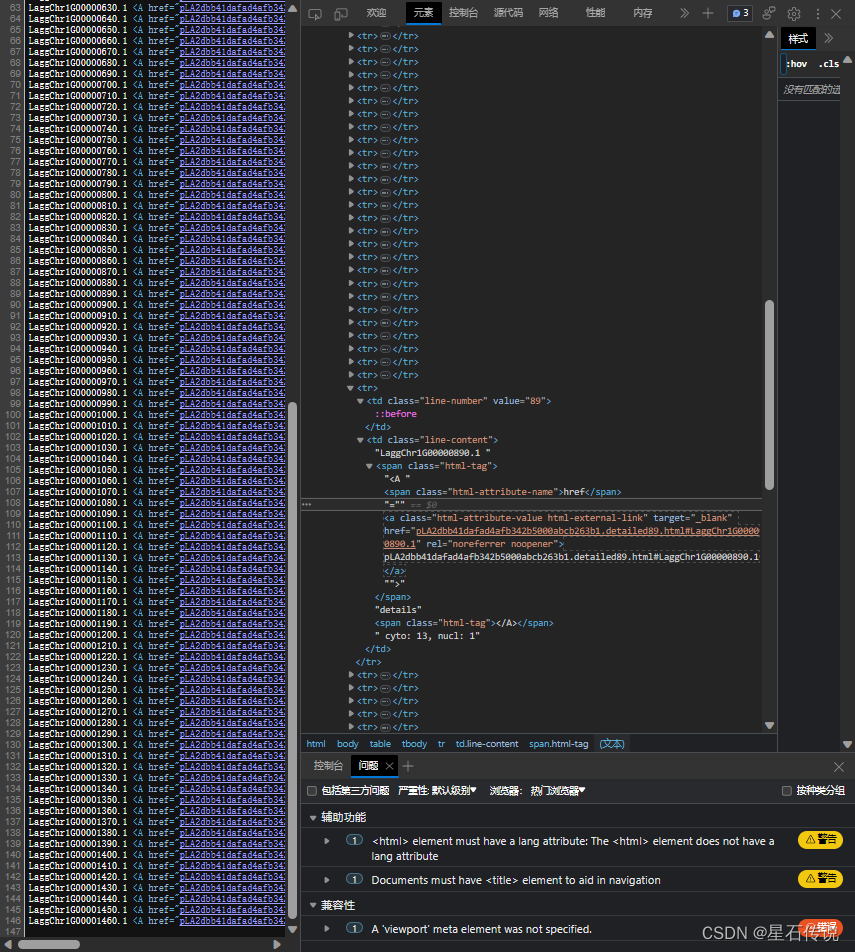
怎么說呢,既然我不能爬取到結果頁面的url,那我只能將結果頁面url的獲得的步驟放在官網中實現,而后面的結果整理則用代碼實現。
總結
本章詳細介紹了許多用于亞細胞定位的網站,其中,我還是比較推薦 WoLFPSORT這個網站的(簡單易懂,十分好上手)。至于后續的代碼實現我也是基于這個網站,但是因為爬蟲學習還不到位(無法爬取到結果頁面的url)。只能等以后在學習爬蟲時,再修改。
羌笛何須怨楊柳,春風不度玉門關。
–2023-8-12 實驗篇



Linux高級命令擴展)
)



)










