轉載自:MetaAI
在生物學研究中,隨著實驗和計算技術的進步,生物系統研究產生了大量高通量數據。技術努力主要集中在提高吞吐量、降低成本和提升實驗與計算效率。因此,整合不同類型組學數據,并通過關聯分析識別關鍵因素和機制的計算方法變得尤為重要。

發表在《Nature Protocols》中的這篇文章,提出了一個可以從多組學中推斷因果關系的系統框架 - Transkingdom Network Analysis (TkNA),并詳細介紹了該框架的使用流程。TkNA是一種獨特的因果推理分析框架,能夠整合多個數據集和不同類型的組學數據,執行薈萃分析并識別關鍵的調控關系。它之前被用于研究抗生素耐藥微生物、2型糖尿病和免疫缺陷相關腸病,以及宮頸癌、淋巴瘤和黑色素瘤中微生物組的作用。TkNA可以識別微生物和微生物基因、宿主途徑、宿主基因以及控制宿主-微生物群相互作用的主要調節因素。
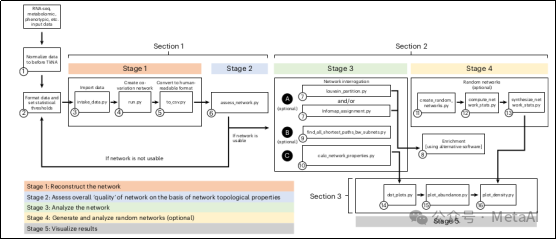
圖1. TkNA流程圖
TkNA流程包括3個主要部分,5個階段,并在這些之前有兩個預處理步驟(圖1)。TkNA可以用于分析實驗驗證后的結果,構成一個循環的分析框架(圖2)。
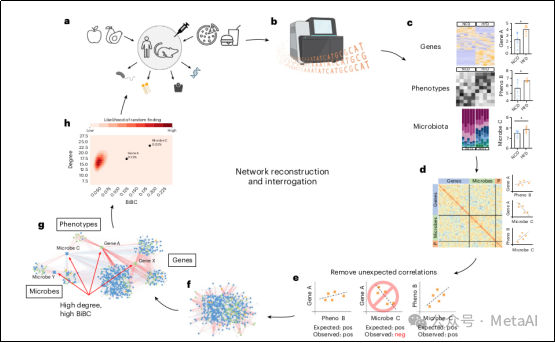
圖2. TkNA管道以循環方式進行
a、進行實驗并獲得樣品。b,在開始TkNA之前對樣品進行測序和標準化。c、對每種數據類型進行比較。bar圖分別代表測量的基因表達水平、微生物豐度和表型。星號代表治療組之間的顯著差異,因為僅保留顯著變化的特征下游分析。d、在每種數據類型之間和內部執行相關性。e,刪除意外的相關性。f、重建網絡。g,詢問網絡以尋找調控節點。h,確定從網絡中找到前部節點的概率,然后進行后續驗證研究并重復該循環。
TkNA不僅可以識別關鍵調節節點,還能計算網絡的拓撲屬性,并通過如Cytoscape這樣的外部程序可視化網絡。TkNA的方法已經用于驗證多項研究中的推論。它提供了一種分析不同組學數據之間交互作用的方法,可以用于分析遺傳和轉錄數據、代謝物、蛋白質和表型之間的相互作用。與其他方法相比,TkNA側重于通過薈萃分析識別跨多個隊列的穩健模式,并且專注于建立因果關系而非僅僅發現關聯。盡管TkNA提供了強大的工具,但用戶仍需要具備一定的統計知識來理解軟件的適用性和局限性。
TkNA的目標用戶是宿主和/或微生物組領域的研究人員,這些人員可能缺乏計算和統計專業知識。該方法適用于生物和生物醫學研究的多個學科,從建立新的細胞和分子治療靶點到研究基礎生物學問題。用戶無需編程專業知識,但需要熟悉Unix環境中的命令行操作,以及能夠理解JSON文件格式以自定義和修改程序選項。
該手冊詳細描述了一個復雜的分析流程和具體步驟,并給出了相應的命令行,這個流程分為3個主要部分,涵蓋了5個階段,以及在這些階段之前的兩個預處理步驟(圖1)。下面只做簡單描述,具體詳細步驟見原文:
第一部分:重建網絡
這部分涉及數據的標準化、文件格式化以及設定統計閾值(預處理步驟)。
階段1:數據導入、計算/薈萃分析和按用戶指定的條件過濾數據。這一步驟首先找出基于用戶定義的統計標準的不同類別樣本(如病態與健康對照組)間表達/豐富的變量(基因、微生物、代謝物等)。然后,進行每組內和組間的相關性分析。
階段2:基于網絡的拓撲特性,用戶決定是否進入下一個分析階段。這些特性包括網絡密度、觀察到的正/負相關偏差及意外相關比例等。
第二部分:詢問/分析重建的網絡
階段3:分析重建的網絡以找出在調查的生物過程中因果作用的節點或節點組。用戶可以使用TkNA識別網絡中的節點集群,并通過外部推薦軟件進行富集分析,以識別集群中的節點所貢獻的生物途徑或功能。
第三部分:從用戶重建的網絡分析中創建發布就緒的圖表
階段4:評估特定節點顯示非隨機值的概率。在這里,TkNA重建了許多隨機網絡,與重建網絡進行比較。
階段5:創建多種高質量的圖表,包括度分布的點圖、節點及其計算屬性的點圖,以及前調節節點的豐富度或表達水平。
圖3展示了TkNA的一些輸出結果。
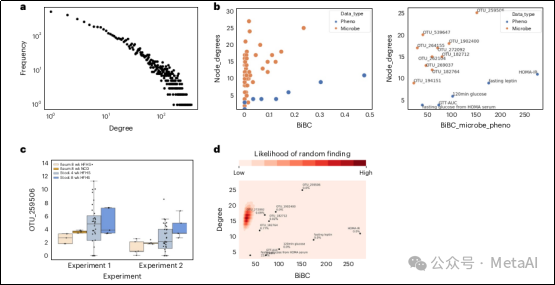
圖3. TkNA生成的示例圖
a,網絡屬性度(degree)分布圖。b,左:節點屬性可視化示例,其中每個點代表重建網絡中的一個節點。右圖:同一張圖,放大了前10個微生物BiBC節點。c,b中前部BiBC節點的豐度/表達圖示例。圖例顯示了數據集中的兩個類。在本例中,將名為“高”的樣本類別與名為“低”的樣本類別進行比較。盒子顯示每次實驗每組的四分位數;須線包括除邊遠點之外的其余分布。d,10,000個隨機網絡的二維密度圖。
TkNA方法依賴于在多個實驗中進行薈萃分析,以識別多個隊列中倍數變化(fold change)和相關性的穩健模式。默認情況下,它使用Fisher方法來組合來自多個獨立測試的P值。其他通用R包(例如,meta、netmeta和mixmeta)提供了多種元分析方法,但這些方法并未考慮因果關系原理,例如相關不等式。其他R軟件包(例如MixOmics、MOFA+和iClusterPlus)也使用復雜的統計方法來組合從同一患者測量的多個組學數據。然而,它們同時應用于多個隊列或獨特的組學數據(其中數據組成或不滿足分布假設)可能具有挑戰性。TkNA提供了一個框架來實現多種組學類型和群組的同時整合。請注意,“整合”一詞指的是兩種截然不同的分析。具體來說,在整篇文章中以以下方式使用它:薈萃分析是來自多個獨立數據集的數據的集成,而網絡重建涉及在多種類型的組學數據之間建立統計依賴關系的集成。
參考文獻:
Newman, N.K., Macovsky, M.S., Rodrigues, R.R. et al. Transkingdom Network Analysis (TkNA): a systems framework for inferring causal factors underlying host–microbiota and other multi-omic interactions. Nat Protoc (2024). https://doi.org/10.1038/s41596-024-00960-w

)


應用APP)


放大器+單片機的中斷系統(中斷的產生背景+使用中斷重寫秒表程序+中斷優先級))











)