一、引言
?????? 周集中老師團隊于2021年在Nature climate change發表的文章,闡述了網絡穩定性評估的原理算法,并提供了完整的代碼。自此對微生物生態網絡的評估具有更全面的指標,自此網絡穩定性的評估廣受大家歡迎。本系列將介紹網絡穩定性之魯棒性和易損性指數的原理,計算方法以及代碼,如有疑問歡迎討論交流。

?????? 為了理解增溫是否以及如何影響構建的微生物生態網絡(MEN)的穩定性,我們使用了多種指標來表征網絡及其嵌入成員的穩定性,包括穩健性、易損性、節點和連接的穩定性、節點持久性以及組成穩定性。我們通過消除模擬和經驗觀察評估了網絡及網絡內微生物群落的穩定性。這一節集中于基于模擬的方式評估網絡穩定性的穩健性(Robustness)和易損性指標。下一節我們將討論基于經驗觀測值評估網絡穩定性。
二、基礎知識之基于模擬的網絡穩定性
穩健性(Robustness)
?????? 微生物生態網絡(MEN)的穩健性定義為在隨機或定向移除節點后網絡中剩余物種的比例。在隨機移除物種的模擬中,隨機移除一定比例的節點;在定向移除模擬中,移除了一定數量的模塊中心節點。為了測試物種移除對剩余物種的影響,計算了節點 i 的加權平均相互作用強度(wMIS),其公式如下:

?????? 式中,bj是物種 j 的相對豐度;
???????????????? sij 是物種 i 和 j 之間的關聯強度,以皮爾森相關系數表示。因此在本研究中,sij = sji。
???????? 從MEN中移除選定節點后,如果 wMISi = 0(即所有與物種 i 的鏈接均被移除)或 wMISi< 0(即物種 i 與其他物種之間的共生協同不足以維持其生存),則節點 i 被視為滅絕/孤立并從網絡中移除。該過程持續進行,直到所有物種的 wMIS 均為正數。剩余節點的比例即為網絡的穩健性。在隨機移除50%的節點或移除5個模塊樞紐節點的情況下,測量了網絡的穩健性。

圖中a,穩健性測量方法是從每個經驗MEN 中隨機移除 50% 的分類單元后剩余的分類單元比例。b,穩健性測量方法是從每個經驗MEN中移除五個模塊中心后剩余的分類單元比例。在a和b中,每個誤差線對應于 100 次模擬的標準差。變暖(W)和控制(Ctl)之間的顯著性差異(雙側t檢驗)用 *** P < 0.001表示。
易損性(Vulnerability)
?????? 每個節點的易損性衡量該節點對全局效率的相對貢獻。網絡的易損性由網絡中節點的最大易損性表示,公式如下:

?????? 式中,E 是網絡的全局效率;
???????????????? Ei 是移除節點 i 及其所有鏈接后的全局效率。
?????? 網絡圖的全局效率計算為所有節點對間效率的平均值:

?????? 式中,d(i, j) 表示節點 i 和節點 j 之間最短路徑上的邊數量。
?????? 效率描述了信息在網絡中傳播的速度。在生態網絡中,效率可以提供關于生物或生態事件后果在網絡局部或整體傳播速度的信息。
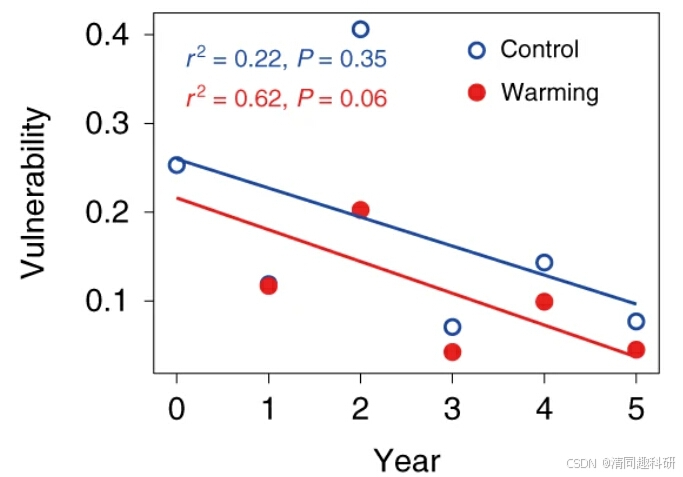
圖中,網絡易損性通過每個網絡中的最大節點易損性來衡量。顯示了線性回歸的調整后R2和P值。
三、示例數據及R代碼
????? 本文的示例數據和代碼均來自于2021年周集中老師團隊的Nature climate change文章,感興趣的同學可以自行去學習。
3.1 魯棒性(Robustness)指標
🌟準備數據
準備otu.csv表格,該表格為要計算魯棒性的網絡otu數據表。代碼每次計算一個網絡的穩健性,因此需要計算幾個網絡就運行幾次代碼,每次將輸入文件名修改。

🌟完整代碼
# 清理工作環境中的所有對象
rm(list = ls())
# 讀取 OTU 表格數據
otutab <- read.csv("control_otu.csv", row.names = 1)
otutab[is.na(otutab)] <- 0 # 將 NA 值替換為 0
# 篩選出在至少 12 個樣本中存在的 OTU
counts <- rowSums(otutab > 0)
otutab <- otutab[counts >= 12,]
# 轉置 OTU 表,計算每種物種的相對豐度
comm <- t(otutab)
sp.ra <- colMeans(comm) / 30000 # 每種物種的相對豐度
#從 OTU 表中計算相關矩陣
cormatrix <- matrix(0, ncol(comm), ncol(comm)) # 初始化相關矩陣
for (i in 1:ncol(comm)) {for (j in i:ncol(comm)) {speciesi <- sapply(1:nrow(comm), function(k) {ifelse(comm[k, i] > 0, comm[k, i], ifelse(comm[k, j] > 0, 0.01, NA))})speciesj <- sapply(1:nrow(comm), function(k) {ifelse(comm[k, j] > 0, comm[k, j], ifelse(comm[k, i] > 0, 0.01, NA))})corij <- cor(log(speciesi)[!is.na(speciesi)], log(speciesj)[!is.na(speciesj)]) # 計算對數變換后的相關性cormatrix[i, j] <- cormatrix[j, i] <- corij # 填充對稱矩陣}
}# 設置行名和列名為物種名稱
row.names(cormatrix) <- colnames(cormatrix) <- colnames(comm)# 僅保留相關系數大于等于 0.80 的連接
cormatrix2 <- cormatrix * (abs(cormatrix) >= 0.80)
cormatrix2[is.na(cormatrix2)] <- 0 # 將 NA 替換為 0
diag(cormatrix2) <- 0 # 去除自連接# 計算網絡連接數量和節點數量
sum(abs(cormatrix2) > 0) / 2 # 連接數
sum(colSums(abs(cormatrix2)) > 0) # 至少有一個連接的節點數# 提取連接網絡的矩陣
network.raw <- cormatrix2[colSums(abs(cormatrix2)) > 0, colSums(abs(cormatrix2)) > 0]
sp.ra2 <- sp.ra[colSums(abs(cormatrix2)) > 0]
sum(row.names(network.raw) == names(sp.ra2)) # 檢查是否匹配# 魯棒性模擬函數
# 隨機移除部分物種后計算剩余物種比例
rand.remov.once <- function(netRaw, rm.percent, sp.ra, abundance.weighted = TRUE) {id.rm <- sample(1:nrow(netRaw), round(nrow(netRaw) * rm.percent))net.Raw <- netRawnet.Raw[id.rm, ] <- 0; net.Raw[, id.rm] <- 0 # 移除物種if (abundance.weighted) {net.stength <- net.Raw * sp.ra} else {net.stength <- net.Raw}sp.meanInteration <- colMeans(net.stength)id.rm2 <- which(sp.meanInteration <= 0) # 移除無連接的物種remain.percent <- (nrow(netRaw) - length(id.rm2)) / nrow(netRaw)remain.percent
}# 魯棒性模擬
rmsimu <- function(netRaw, rm.p.list, sp.ra, abundance.weighted = TRUE, nperm = 100) {t(sapply(rm.p.list, function(x) {remains <- sapply(1:nperm, function(i) {rand.remov.once(netRaw = netRaw, rm.percent = x, sp.ra = sp.ra, abundance.weighted = abundance.weighted)})remain.mean <- mean(remains)remain.sd <- sd(remains)remain.se <- sd(remains) / sqrt(nperm)result <- c(remain.mean, remain.sd, remain.se)names(result) <- c("remain.mean", "remain.sd", "remain.se")result}))
}# 加權和非加權模擬
Weighted.simu <- rmsimu(netRaw = network.raw, rm.p.list = seq(0.05, 1, by = 0.05), sp.ra = sp.ra2, abundance.weighted = TRUE, nperm = 100)
Unweighted.simu <- rmsimu(netRaw = network.raw, rm.p.list = seq(0.05, 1, by = 0.05), sp.ra = sp.ra2, abundance.weighted = FALSE, nperm = 100)# 整合結果
dat1 <- data.frame(Proportion.removed = rep(seq(0.05, 1, by = 0.05), 2),rbind(Weighted.simu, Unweighted.simu),weighted = rep(c("weighted", "unweighted"), each = 20),year = rep(2014, 40),treat = rep("Warmed", 40)#根據自己的處理修改treat名稱
)# 保存結果到文件
write.csv(dat1, "random_removal_result_Y14_W.csv")# 加載 ggplot2 包
library(ggplot2)# 生成加權網絡的結果圖
ggplot(dat1[dat1$weighted == "weighted",], aes(x = Proportion.removed, y = remain.mean, group = treat, color = treat)) +geom_line() +geom_pointrange(aes(ymin = remain.mean - remain.sd, ymax = remain.mean + remain.sd), size = 0.2) +scale_color_manual(values = c("blue", "red")) +xlab("Proportion of species removed") +ylab("Proportion of species remained") +theme_light() +facet_wrap(~year, ncol = 3)# 生成非加權網絡的結果圖
ggplot(dat1[dat1$weighted == "unweighted",], aes(x = Proportion.removed, y = remain.mean, group = treat, color = treat)) +geom_line() +geom_pointrange(aes(ymin = remain.mean - remain.sd, ymax = remain.mean + remain.sd), size = 0.2) +scale_color_manual(values = c("blue", "red")) +xlab("Proportion of species removed") +ylab("Proportion of species remained") +theme_light() +facet_wrap(~year, ncol = 3)🌟輸出結果
如下所示,結果輸出名為random_removal_result_Y14_W.csv的表格。
在隨機移除50%的節點情況下即Proportion.removed值為0.5時,獲得該網絡的魯棒性值0.21278;標準差是0.01966,該值對應于 100 次模擬的標準差。
?? weighted考慮到物種豐度了,ubweighted沒有考慮物種豐度。

隨后可基于這個結果繪制柱狀圖或其他圖紙。
??移除5個模塊中心點的魯棒性值計算代碼我們也放在了示例代碼和數據文件中,需要的可以留言獲取。
3.2 易損性(vulnerability)指標
🌟準備數據
準備otu.csv表格,該表格為要計算易損性的網絡otu數據表。
代碼每次計算一個網絡的易損性,因此需要計算幾個網絡就運行幾次代碼,每次將輸入文件名修改。

🌟完整代碼
# 清理工作環境中的所有對象
rm(list = ls())# 導入igraph包和自定義的centrality函數
if(!require("igraph")){install.packages("igraph")}
library(igraph)
source("info.centrality.R")#自定義函數#### 獲取圖結構(Graph)####
# 可以通過以下三種方式獲取圖結構:
# 1. 讀取已有的圖
# 2. 從OTU表中構建圖
# 3. 從MENAP下載的相關矩陣構建圖## 2) 從OTU表中構建圖
# 讀取OTU表,缺失值替換為0
otutab <- read.csv("treat_otu.csv", row.names = 1)
otutab[is.na(otutab)] <- 0# 篩選至少出現在12個樣本中的OTU
counts <- rowSums(otutab > 0)
otutab <- otutab[counts >= 12,]# 轉置OTU表,以便進行后續計算
comm <- t(otutab)# 初始化相關矩陣,存儲物種間的相關性
cormatrix <- matrix(0, ncol(comm), ncol(comm))# 使用嵌套循環計算OTU之間的相關性
for (i in 1:ncol(comm)) {for (j in i:ncol(comm)) {speciesi <- sapply(1:nrow(comm), function(k) {ifelse(comm[k, i] > 0, comm[k, i], ifelse(comm[k, j] > 0, 0.01, NA))})speciesj <- sapply(1:nrow(comm), function(k) {ifelse(comm[k, j] > 0, comm[k, j], ifelse(comm[k, i] > 0, 0.01, NA))})# 計算物種對數值的相關性corij <- cor(log(speciesi)[!is.na(speciesi)], log(speciesj)[!is.na(speciesj)])cormatrix[i, j] <- cormatrix[j, i] <- corij}
}# 設置相關矩陣的行名和列名
row.names(cormatrix) <- colnames(cormatrix) <- colnames(comm)# 僅保留相關系數大于等于0.80的連接
cormatrix2 <- cormatrix * (abs(cormatrix) >= 0.80)
cormatrix2[is.na(cormatrix2)] <- 0
diag(cormatrix2) <- 0 # 移除自連接
cormatrix2[abs(cormatrix2) > 0] <- 1 # 轉換為鄰接矩陣# 從鄰接矩陣構建無向圖g
g <- graph_from_adjacency_matrix(as.matrix(cormatrix2), mode = "undirected", weighted = NULL, diag = FALSE)### 結束圖構建部分# 移除孤立節點
iso_node_id <- which(degree(g) == 0)
g2 <- delete.vertices(g, iso_node_id) # 生成不含孤立節點的圖g2# 檢查節點和連接數
length(V(g2)) # 節點數
length(E(g2)) # 邊數# 計算每個節點的易損性
node.vul <- info.centrality.vertex(g2)
max(node.vul) # 輸出最大易損性
🌟輸出結果

四、參考文獻
[1] Yuan, M.M., Guo, X., Wu, L. et al. Climate warming enhances microbial network complexity and stability. Nat. Clim. Chang. 11, 343–348 (2021).
[2] Mengting-Maggie-Yuan/warming-network-complexity-stability
五、相關信息
!!!本文內容由小編總結互聯網和文獻內容總結整理,如若侵權,聯系立即刪除!
?!!!有需要的小伙伴評論區獲取今天的測試代碼和實例數據。
?📌示例代碼中提供了數據和代碼,小編已經測試,可直接運行。
以上就是本節所有內容。
?
?







)


)


:Linux權限管理)





