
Nifty graph
A contribution by Matt Asher.

3D animation
A 3D animated scatterplot made with R and rgl.
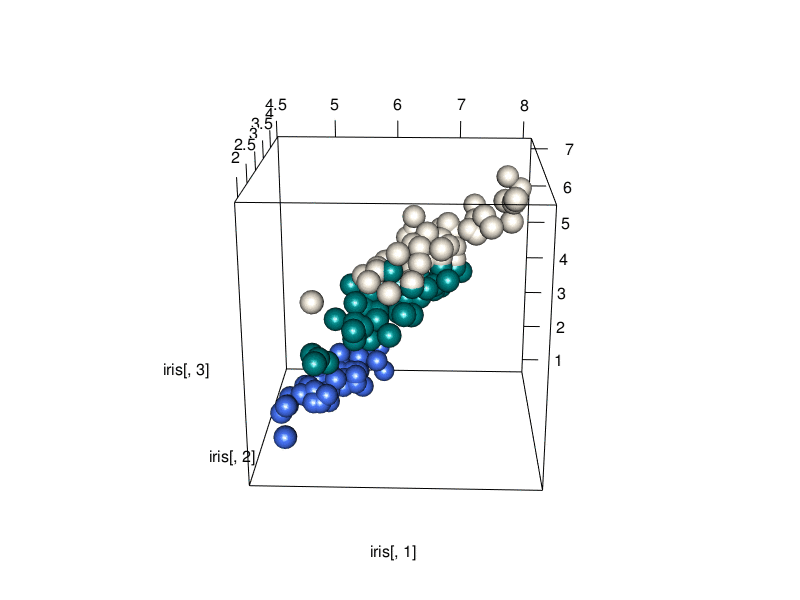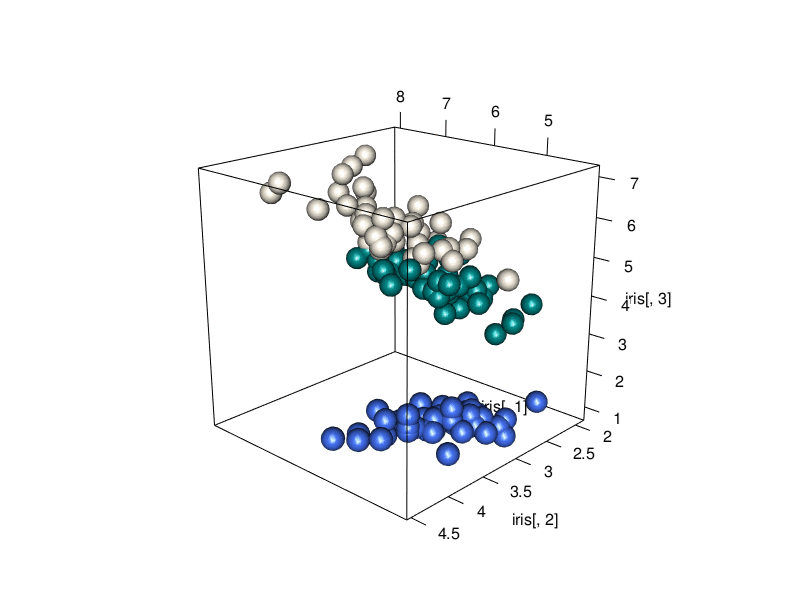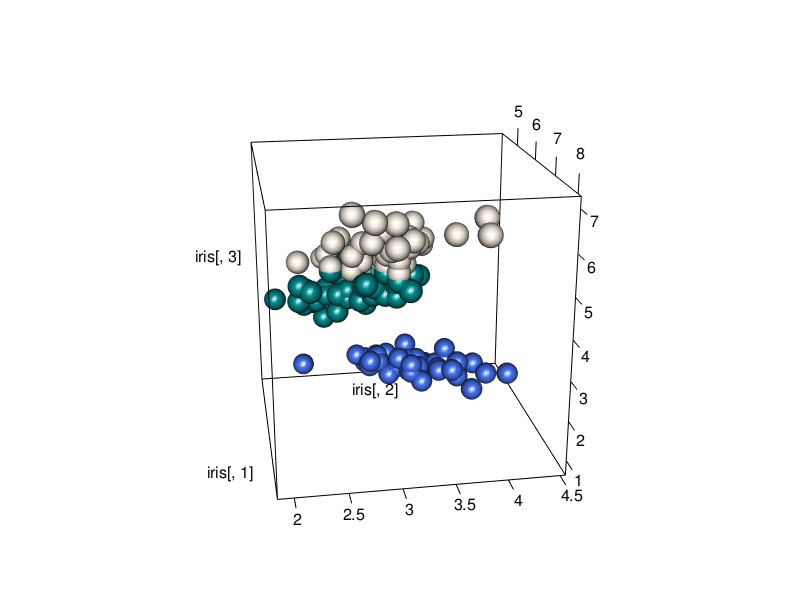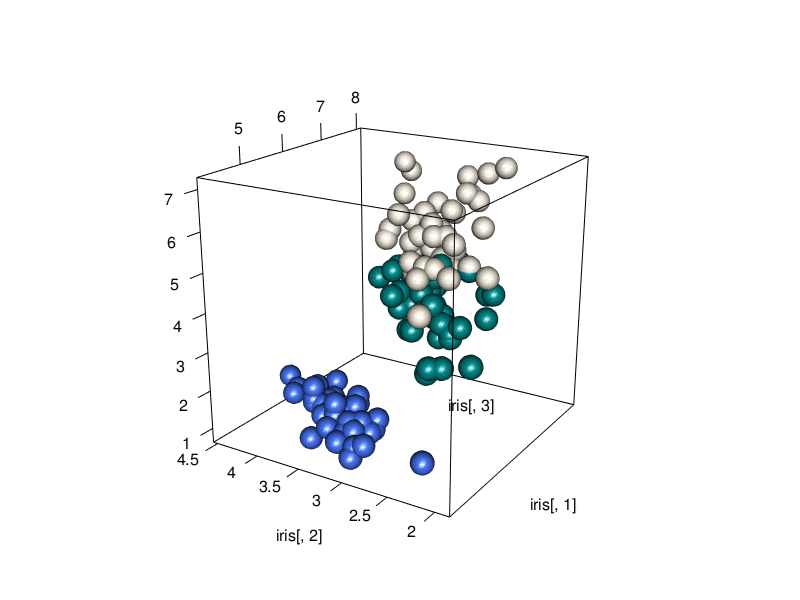
文章目錄
- Nifty graph
- 3D animation
- 2025.04.24【3D】| 3D繪圖入門指南
- 什么是3D繪圖?
- 為什么使用3D繪圖?
- 如何在R中進行3D繪圖?
- 安裝和加載`rgl`包
- 創建基本的3D散點圖
- 添加軸和標簽
- 自定義視圖
- 3D繪圖在生物信息學中的應用
- 基因表達數據的可視化
- 蛋白質結構的可視化
- 復雜生物網絡的可視化
- 結論
2025.04.24【3D】| 3D繪圖入門指南
在生物信息學領域,數據可視化是理解復雜數據集的關鍵。3D圖表,盡管在某些情況下被認為是不良實踐,但在特定情境下仍能提供獨特的視角和洞察。R語言通過rgl包允許我們構建三維圖表,這對于展示多層次數據結構尤其有用。本摘要旨在概述R中3D圖表的構建方法,以及如何在生物信息學中應用這些技術來更好地解釋和呈現數據。通過一系列實例,我們將探討如何利用R的rgl包來創建和定制3D圖表,以及如何將這些圖表應用于生物信息學數據分析,從而幫助我們更深入地理解基因表達、蛋白質結構和復雜生物網絡。
什么是3D繪圖?
3D繪圖是一種數據可視化技術,它通過在三維空間中展示數據點來提供更豐富的視覺信息。這種技術可以幫助我們理解數據的深度和層次結構,尤其是在處理復雜的生物信息學數據時。
為什么使用3D繪圖?
盡管3D繪圖有時被認為是不良實踐,因為它可能會增加認知負荷并導致誤解,但在特定情況下,它能夠提供獨特的視角和洞察。例如,在展示蛋白質結構或基因表達數據的空間分布時,3D圖表可以提供更直觀的理解。
如何在R中進行3D繪圖?
R語言提供了強大的工具來創建3D圖表,其中最著名的是rgl包。以下是如何在R中使用rgl包進行3D繪圖的基本步驟。
安裝和加載rgl包
首先,我們需要安裝并加載rgl包。如果你還沒有安裝這個包,可以使用以下命令進行安裝:
install.packages("rgl")然后,加載這個包:
library(rgl)創建基本的3D散點圖
讓我們從一個簡單的3D散點圖開始。這個例子將展示如何在三維空間中繪制一組隨機生成的數據點。
# 生成隨機數據
set.seed(123)
x <- rnorm(100)
y <- rnorm(100)
z <- rnorm(100)# 創建3D散點圖
plot3d(x, y, z, col = "blue", pch = 19)在這個例子中,plot3d函數用于創建3D散點圖。col參數用于設置點的顏色,pch參數用于設置點的形狀。
添加軸和標簽
為了使圖表更易于理解,我們可以添加軸和標簽。
# 添加軸和標簽
axes3d(col = "red")axes3d函數用于添加軸和標簽。col參數用于設置軸的顏色。
自定義視圖
我們可以通過調整視圖參數來自定義圖表的視角。
# 自定義視圖
view3d(theta = -40, phi = 30, fov = 60, zoom = 0.7)在這個例子中,view3d函數用于調整視圖參數。theta和phi參數用于設置視角的角度,fov參數用于設置視場角,zoom參數用于設置縮放級別。
3D繪圖在生物信息學中的應用
現在我們已經了解了如何在R中創建基本的3D圖表,讓我們看看這些技術如何在生物信息學中應用。
基因表達數據的可視化
基因表達數據通常包含大量的變量和樣本。3D圖表可以幫助我們理解基因表達模式的空間分布。
# 假設我們有一個基因表達矩陣
gene_expression <- matrix(rnorm(1000), nrow = 10, ncol = 100)# 創建3D散點圖
plot3d(1:100, rep(1, 100), gene_expression[,1], col = "blue", pch = 19)# 添加軸和標簽
axes3d(col = "red")# 自定義視圖
view3d(theta = -40, phi = 30, fov = 60, zoom = 0.7)在這個例子中,我們使用了一個假設的基因表達矩陣來創建一個3D散點圖。每個點代表一個基因在特定條件下的表達水平。
蛋白質結構的可視化
蛋白質結構數據可以通過3D圖表直觀地展示。這種類型的圖表可以幫助我們理解蛋白質的三維結構和功能。
# 假設我們有一個蛋白質結構數據集
protein_structure <- data.frame(x = rnorm(100),y = rnorm(100),z = rnorm(100),type = sample(c("alpha", "beta"), 100, replace = TRUE)
)# 創建3D散點圖
plot3d(protein_structure$x, protein_structure$y, protein_structure$z, col = protein_structure$type, pch = 19)# 添加軸和標簽
axes3d(col = "red")# 自定義視圖
view3d(theta = -40, phi = 30, fov = 60, zoom = 0.7)在這個例子中,我們使用了一個假設的蛋白質結構數據集來創建一個3D散點圖。每個點代表一個氨基酸殘基的位置,顏色表示殘基的類型。
復雜生物網絡的可視化
生物網絡通常包含大量的節點和邊。3D圖表可以幫助我們理解網絡的結構和動態。
# 假設我們有一個生物網絡數據集
bio_network <- data.frame(x = rnorm(50),y = rnorm(50),z = rnorm(50),node_id = 1:50
)# 創建3D網絡圖
plot3d(bio_network$x, bio_network$y, bio_network$z, col = "green", pch = 16)# 添加邊
for (i in 1:(nrow(bio_network) - 1)) {lines3d(bio_network[i,], bio_network[i + 1,], col = "grey")
}# 添加軸和標簽
axes3d(col = "red")# 自定義視圖
view3d(theta = -40, phi = 30, fov = 60, zoom = 0.7)在這個例子中,我們使用了一個假設的生物網絡數據集來創建一個3D網絡圖。每個點代表一個節點,線表示節點之間的連接。
結論
通過這篇文章,我們學習了如何在R中使用rgl包創建基本的3D圖表,并探討了這些技術在生物信息學中的應用。雖然3D圖表在某些情況下可能不是最佳選擇,但在特定情境下,它們可以提供獨特的視角和洞察。希望這篇文章能幫助你更好地理解和應用3D繪圖技術。
🌟 非常感謝您抽出寶貴的時間閱讀我的文章。如果您覺得這篇文章對您有所幫助,或者激發了您對生物信息學的興趣,我誠摯地邀請您:
👍 點贊這篇文章,讓更多人看到我們共同的熱愛和追求。
🔔 關注我的賬號,不錯過每一次知識的分享和探索的旅程。
📢 您的每一個點贊和關注都是對我最大的支持和鼓勵,也是推動我繼續創作優質內容的動力。
📚 我承諾,將持續為您帶來深度與廣度兼具的生物信息學內容,讓我們一起在知識的海洋中遨游,發現更多未知的奇跡。
💌 如果您有任何問題或想要進一步交流,歡迎在評論區留言,我會盡快回復您。
![[特殊字符] 分布式事務中,@GlobalTransactional 與 @Transactional 到底怎么配合用?](http://pic.xiahunao.cn/[特殊字符] 分布式事務中,@GlobalTransactional 與 @Transactional 到底怎么配合用?)
)











)


等級考試試卷-理論綜合)
)

