多模態融合算法應用 · CT +臨床文本數據 + pyradiomics提取圖像特征
- 單模態建模
- 臨床數據建模
- pyradiomics提取圖像特征建模
- CT建模
- 多模態建模
- 前融合
- 為什么能直接合并在一起?
- 后融合
- Med-CLIP:深度學習 + 可解釋性
?
單模態建模
臨床數據建模
臨床文本數據:
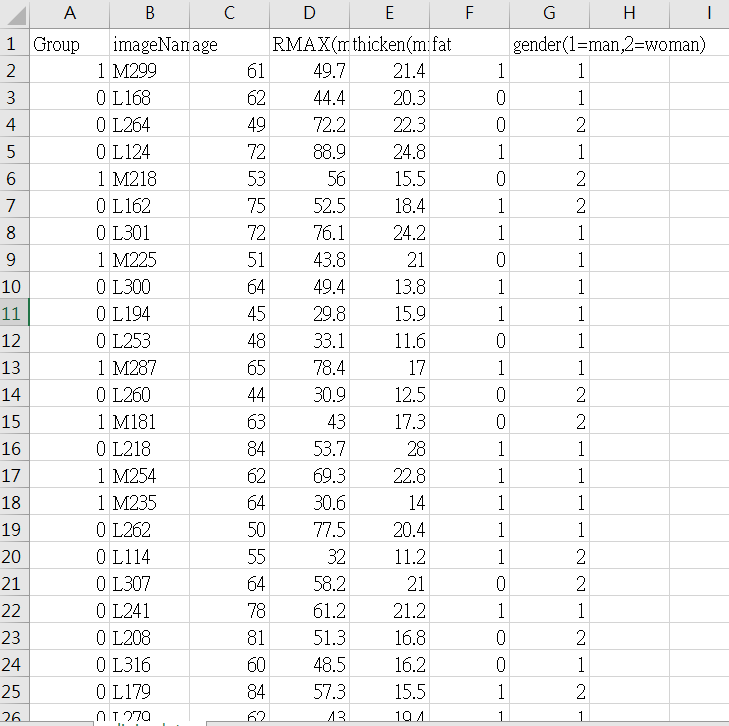
- Group: 目標分類標簽,表示樣本屬于哪一組(0或1)。
- imageName: 圖像名稱,表示每個樣本對應的圖像的名稱。
- age: 年齡,表示樣本的年齡。
- RMAX(mm): 某一特征的數值,單位為毫米。
- thicken(mm): 另一特征的數值,單位為毫米。
- fat: 體脂率,表示樣本的體脂含量。
- gender(1=man,2=woman): 性別編碼,1表示男性,2表示女性。
-
第1行
- Group: 1(正類)
- imageName: M299(對應哪個病)
- age: 61
- RMAX(mm): 49.7
- thicken(mm): 21.4
- fat: 1(有體脂)
- gender: 1(男性)
-
第2行
- Group: 0(負類)
- imageName: L168(對應哪個病)
- age: 62
- RMAX(mm): 44.4
- thicken(mm): 20.3
- fat: 0(無體脂)
- gender: 1(男性)
-
第3行
- Group: 0(負類)
- imageName: L264(對應哪個病)
- age: 49
- RMAX(mm): 72.2
- thicken(mm): 22.3
- fat: 0(無體脂)
- gender: 2(女性)
這張圖展示了數據集的前幾行樣本及其特征,包括年齡、RMAX值、thicken值、體脂率和性別。
樣本比(數據平衡):
-
0 類 122
-
1 類 106
# 導入常用庫
import sys # 系統特定參數和函數
import pandas as pd # 數據處理和分析庫
import os # 操作系統接口模塊
import random # 生成隨機數的模塊
import shutil # 文件操作模塊
import sklearn # 機器學習庫
import scipy # 科學計算庫
import numpy as np # 數組和矩陣處理庫
import matplotlib.pyplot as plt # 數據可視化庫
from sklearn.linear_model import LassoCV # 導入LassoCV回歸模型
from sklearn.preprocessing import StandardScaler # 導入標準化工具
import seaborn as sns # 數據可視化庫clinic_df = pd.read_csv("clinic_data.csv") # 讀取臨床數據CSV文件
new_clinic_df = clinic_df.drop('imageName', axis=1) # 刪除無用的'imageName'列
selected_columns1 = new_clinic_df.columns # 獲取數據框的列名from pycaret.classification import * # 導入PyCaret分類模塊
s1 = ClassificationExperiment() # 創建分類實驗對象
s1.setup(data = new_clinic_df, target = 'Group', session_id=123, fix_imbalance_method=False, normalize=True)
# 設置分類實驗的初始參數:使用new_clinic_df數據框,目標列為'Group',設置隨機種子為123,不進行數據平衡處理,對數據進行標準化
best1 = s1.compare_models() # 比較不同的分類模型,選擇表現最好的模型
s1.evaluate_model(best1) # 評估最優模型的性能
s1.plot_model(best1, 'auc') # 繪制最優模型的ROC曲線并顯示AUC值
s1.predict_model(best1) # 使用最優模型對數據進行預測
預測結果:模型的準確率 (Accuracy) 為 68.12%。
嘗試的模型有:
- Logistic Regression (LR):邏輯回歸
- K Nearest Neighbors (KNN):K近鄰
- Naive Bayes (NB):樸素貝葉斯
- Decision Tree (DT):決策樹
- Random Forest (RF):隨機森林
- Gradient Boosting Classifier (GB):梯度提升分類器
- Support Vector Machine (SVM):支持向量機
- Light Gradient Boosting Machine (LightGBM):輕量梯度提升機
- Extreme Gradient Boosting (XGBoost):極限梯度提升
- CatBoost Classifier (CatBoost):CatBoost分類器
- Extra Trees Classifier (ET):極端隨機樹
- AdaBoost Classifier (ADA):AdaBoost分類器
- Linear Discriminant Analysis (LDA):線性判別分析
- Quadratic Discriminant Analysis (QDA):二次判別分析
?
pyradiomics提取圖像特征建模
pyradiomics提取圖像特征,有近50種。

import sys
import pandas as pd
import os
import random
import shutil
import sklearn
import scipy
import numpy as np
import radiomics # 這個庫專門用來提取特征
from radiomics import featureextractor
from sklearn.linear_model import LassoCV # 導入Lasso工具包LassoCV
from sklearn.preprocessing import StandardScaler # 標準化工具包StandardScalerfeature_df = pd.read_csv("radiomics_feature_data.csv") # 讀取放射學特征數據的CSV文件
new_feature_df = feature_df.drop("imageName", axis=1) # 刪除無用的'imageName'列
from pycaret.classification import * # 導入PyCaret分類模塊s2 = ClassificationExperiment() # 創建分類實驗對象
s2.setup(data = new_feature_df, target = 'Group', session_id=123, fix_imbalance_method=False, normalize=True, feature_selection = True, feature_selection_method='classic', n_features_to_select=0.2)
# 設置分類實驗的初始參數:使用new_feature_df數據框,目標列為'Group',設置隨機種子為123,不進行數據平衡處理,對數據進行標準化,
# 使用特征選擇,特征選擇方法為'classic',選擇20%的特征(機器學習不合適太多特征,需要選擇一些主要信息的特征)selected_columns = s2.dataset_transformed.columns # 獲取轉換后數據集的列名(代表選擇的特征有哪些)
best2 = s2.compare_models() # 比較不同的分類模型,選擇表現最好的模型
s2.predict_model(best2) # 評估最優模型的性能
預測結果:模型的準確率 (Accuracy) 為 67.33%。
比臨床數據預測的 68.12% 低一些。
從所有特征選了20%特征,具體是哪些:
['A_wavelet-LHL_glszm_SmallAreaEmphasis','A_wavelet-HHH_glszm_SizeZoneNonUniformityNormalized','A_wavelet-LLL_glszm_GrayLevelNonUniformityNormalized','A_wavelet-LHH_glcm_InverseVariance','A_original_glszm_SizeZoneNonUniformityNormalize','A_original_ngtdm_Strength','A_wavelet-LHH_glszm_GrayLevelNonUniformity','A_log-sigma-5-0-mm-3D_firstorder_90Percentile','A_wavelet-HLL_glcm_MCC', 'A_log-sigma-4-0-mm-3D_ngtdm_Contrast']
CT建模
?
多模態建模
前融合
把臨床文本數據(刪除imageName了,5個)和 pyradiomics提取到的圖像特征(20%,9個)
columns = ['Group', 'age', 'RMAX(mm)', 'thicken(mm)', 'fat','gender(1=man,2=woman)','A_wavelet-LHL_glszm_SmallAreaEmphasis','A_wavelet-HHH_glszm_SizeZoneNonUniformityNormalized','A_wavelet-LLL_glszm_GrayLevelNonUniformityNormalized','A_wavelet-LHH_glcm_InverseVariance','A_original_glszm_SizeZoneNonUniformityNormalized','A_original_ngtdm_Strength','A_wavelet-LHH_glszm_GrayLevelNonUniformity','A_log-sigma-5-0-mm-3D_firstorder_90Percentile','A_wavelet-HLL_glcm_MCC', 'A_log-sigma-4-0-mm-3D_ngtdm_Contrast']
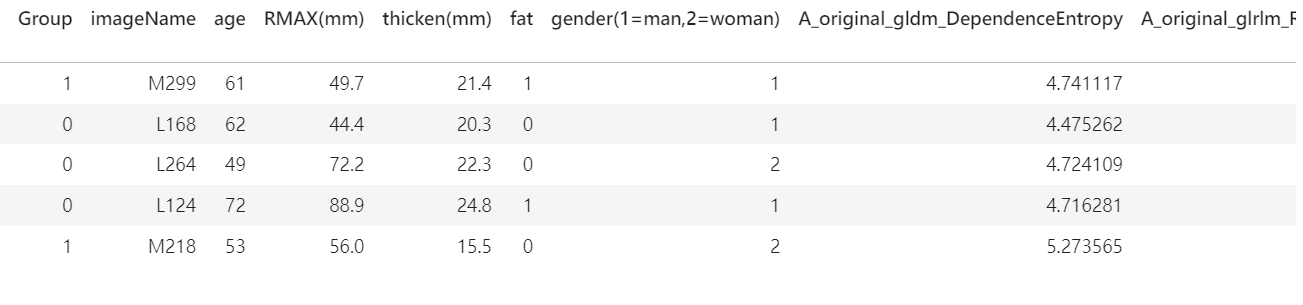
merge_df = pd.read_csv("merge.csv") # 讀取合并數據的CSV文件
columns = ['Group', 'age', 'RMAX(mm)', 'thicken(mm)', 'fat','gender(1=man,2=woman)', 'A_wavelet-LHL_glszm_SmallAreaEmphasis','A_wavelet-HHH_glszm_SizeZoneNonUniformityNormalized','A_wavelet-LLL_glszm_GrayLevelNonUniformityNormalized','A_wavelet-LHH_glcm_InverseVariance','A_original_glszm_SizeZoneNonUniformityNormalized','A_original_ngtdm_Strength','A_wavelet-LHH_glszm_GrayLevelNonUniformity','A_log-sigma-5-0-mm-3D_firstorder_90Percentile','A_wavelet-HLL_glcm_MCC', 'A_log-sigma-4-0-mm-3D_ngtdm_Contrast']
# 指定需要的列,包含目標列和若干特征列new_merge_df = merge_df[columns] # 從數據框中選擇指定的列
from pycaret.classification import * # 導入PyCaret分類模塊s3 = ClassificationExperiment() # 創建分類實驗對象
s3.setup(data = new_merge_df, target = 'Group', session_id=123, fix_imbalance_method=False, normalize=True)
# 設置分類實驗的初始參數:使用new_merge_df數據框,目標列為'Group',設置隨機種子為123,不進行數據平衡處理,對數據進行標準化best3 = s3.compare_models() # 比較不同的分類模型,選擇表現最好的模型
s3.predict_model(best3) # 使用最優模型對數據進行預測
前融合預測結果:71.01%。
-
pyradiomics提取圖像(20%)特征建模:67.33%
-
臨床數據建模:68.12%
如果想進一步優化,可以多選一些特征,這個只選了20%。
為什么能直接合并在一起?
臨床數據
(Group’, ‘age’, ‘RMAX(mm)’, ‘thicken(mm)’, ‘fat’,
‘gender(1=man,2=woman)’)
和pyradiomics提取圖像特征
(Group、‘A_wavelet-LHL_glszm_SmallAreaEmphasis’,
‘A_wavelet-HHH_glszm_SizeZoneNonUniformityNormalized’,
‘A_wavelet-LLL_glszm_GrayLevelNonUniformityNormalized’,
‘A_wavelet-LHH_glcm_InverseVariance’,
‘A_original_glszm_SizeZoneNonUniformityNormalized’,
‘A_original_ngtdm_Strength’,
‘A_wavelet-LHH_glszm_GrayLevelNonUniformity’,
‘A_log-sigma-5-0-mm-3D_firstorder_90Percentile’,
‘A_wavelet-HLL_glcm_MCC’, ‘A_log-sigma-4-0-mm-3D_ngtdm_Contrast’)
為什么能直接合并在一起?
臨床數據和從圖像中提取的放射學特征可以直接合并在一起是因為它們都描述了同一組樣本的不同方面。
后融合
Med-CLIP:深度學習 + 可解釋性
不止 pyradiomics 能提取圖像特征,深度學習方法更好,但深度學習方法提取的特征沒有可解釋性。
從高維空間提取的特征,最后壓縮成一行給你,完全看不懂到底是什么。
那使用多模態大模型方式更好。
雖然也是臨床數據(年齡、性別、疾病標簽等)+ 患者圖像數據進行預測,但大模型會給你詳細的解釋 — 之所以說 yyy 病,是因為 xxx 特征,是真能解釋清楚。







)




![[leetcode hot 150]第五百三十題,二叉搜索樹的最小絕對差](http://pic.xiahunao.cn/[leetcode hot 150]第五百三十題,二叉搜索樹的最小絕對差)



進程與線程)


)