R包的安裝,每次做分析的時候先運行這段代碼把R包都安裝好了,這段代碼不需要任何改動,每次分析直接運行。
options("repos"="https://mirrors.ustc.edu.cn/CRAN/")
if(!require("BiocManager")) install.packages("BiocManager",update = F,ask = F)
options(BioC_mirror="https://mirrors.ustc.edu.cn/bioc/")cran_packages <- c('tidyr','tibble','dplyr','stringr','ggplot2','ggpubr','factoextra','FactoMineR','devtools','cowplot','patchwork','basetheme','paletteer','AnnoProbe','ggthemes','VennDiagram','tinyarray')
Biocductor_packages <- c('GEOquery','hgu133plus2.db','ggnewscale',"limma","impute","GSEABase","GSVA","clusterProfiler","org.Hs.eg.db","preprocessCore","enrichplot")for (pkg in cran_packages){if (! require(pkg,character.only=T,quietly = T) ) {install.packages(pkg,ask = F,update = F)require(pkg,character.only=T) }
}for (pkg in Biocductor_packages){if (! require(pkg,character.only=T,quietly = T) ) {BiocManager::install(pkg,ask = F,update = F)require(pkg,character.only=T) }
}#前面的所有提示和報錯都先不要管。主要看這里
for (pkg in c(Biocductor_packages,cran_packages)){require(pkg,character.only=T)
}
#沒有任何提示就是成功了,如果有warning xx包不存在,用library檢查一下。#library報錯,就單獨安裝。表達矩陣和臨床信息的獲取、疾病組和健康組的分組工作
rm(list = ls())#首先清空一下環境防止原來數據的影響
#打破下載時間的限制,改前60秒,改后10w秒
options(timeout = 100000)
options(scipen = 20)#不要以科學計數法表示#傳統下載方式
library(GEOquery)
eSet = getGEO("GSE57338", destdir = '.', getGPL = F)#這是一個示例數據集,是關于DCM這種病的
#網速太慢,下不下來怎么辦
#1.從網頁上下載/發鏈接讓別人幫忙下,放在工作目錄里
#2.試試geoChina,只能下載2019年前的表達芯片數據
#library(AnnoProbe)
#eSet = geoChina("GSE7305") #選擇性代替第8行#前面下載下來的數據保存到了eSet這個變量中
class(eSet)#發現是一個
length(eSet)eSet = eSet[[1]]
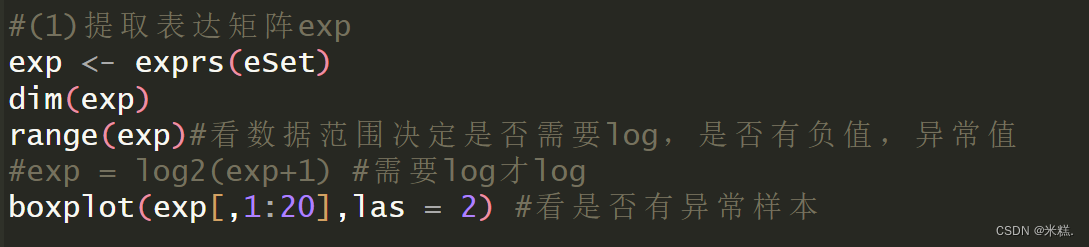
class(eSet)#(1)提取表達矩陣exp
exp <- exprs(eSet)
dim(exp)
range(exp)#看數據范圍決定是否需要log,是否有負值,異常值
#exp = log2(exp+1) #需要log才log
boxplot(exp[,1:20],las = 2) #看是否有異常樣本#(2)提取臨床信息
pd <- pData(eSet)
as.data.frame(pd)
pd$title
k = str_detect(pd$title,"Non|CMP");table(k)
pd<-pd[k,]
#(3)讓exp列名與pd的行名順序完全一致
p = identical(rownames(pd),colnames(exp));p
if(!p) {
#intersect用于找出兩個向量間的公共元素,返回值是兩個向量中相同的元素
#intersect函數比較的時候是不考慮下標的,只要某個元素在傳給該函數的兩個向量中同時存在,那這個元素就是函數返回值的某個元素s = intersect(rownames(pd),colnames(exp))exp = exp[,s]#根據名字提取exp中的列pd = pd[s,]#根據名字提取pd的行
}#這樣得到的exp列名和pd行名順序完全一致#(4)提取芯片平臺編號,后面要根據它來找探針注釋
gpl_number <- eSet@annotation;gpl_number
save(pd,exp,gpl_number,file = "step1output.Rdata")# 原始數據處理的代碼,按需學習
# https://mp.weixin.qq.com/s/0g8XkhXM3PndtPd-BUiVgw
eSet = getGEO("GSE57338", destdir = '.', getGPL = F)是我們需要根據情況修改的第一句代碼,用哪個數據集就把第一個參數改成哪個數據集。
再來研究一下eSet

getGEO函數下載下來的數據集我們是用eSet這個變量接收的,通過上面一系列代碼可以知道這是一個列表,且只有一個元素(如果有兩個元素就說明是不同批次的或者同一個數據集是不同公司測的),既然只有一個元素那直接提取出來就是了,提取列表中的元素要用兩個中括號,提取出來第一個元素之后覆蓋掉eSet中的內容,此時再次查看eSet的類型,發現是一個看不懂的東西,反正也是一個類型,這是一個高級類型,總之就理解成接收了我們下載的GEO數據集的一個對象。這個eSet的內容長這樣子

這一大堆亂七八糟的是肯定沒法直接扔進后續的函數里面做差異分析的,因此接下來我們要提取表達矩陣,直接運行這段代碼就行,這段代碼不需要修改
其中把eSet這個對象扔到exprs這個函數里面就能得到對應的表達矩陣了,表達矩陣長這樣

其中每一列是一個樣本,每一行的行名其實是一個探針,而每一個探針都會對應一個基因,后續我們會根據探針與基因名的對應關系把這些行名改成基因名。現在只需要明確一點就是探針的表達量代表基因的表達量就行。
我們把表達矩陣存到exp里面,dim(exp)用來查看這個矩陣的維度(查看維度要做什么后面再說),range(exp)用來查看這個矩陣中元素的范圍,如果數據從個位數到幾萬都有,那這個矩陣中元素就是沒有取過對數的,我們需要手動取一下以2為底的對數,不要問為什么取2的對數不是其他的,這就是一個約定俗成的東西,其中在取對數的這一步我們對exp矩陣的每個元素都做了+1的運算,這個確實會改變基因的表達量,但是我們是做差異分析,基因表達量都+1并不會改變他們之間的差異性,之所以+1是為了防止出現一些負數等等。然后使用基本繪圖系統的boxplot函數繪制了一個箱線圖,boxplot的第一個參數傳了exp這個矩陣的前20列,這是因為我用的這個數據集前面查看維度的時候發現是三萬多行,三百多列,直接用exp作為參數繪制箱線圖非常的密集而導致難以觀察,所以我取了前20行,如果前面dim(exp)的時候發現矩陣的列很少,那就不用取前20列了。我用的這個數據集前20繪制的箱線圖長這樣

發現數據還是很正常的,沒有上下浮動很明顯的樣本。如果有異常就需要做一些另外的處理比如刪除等等,這個以后單獨介紹一下,這里就不介紹了。
接下來再提取臨床信息
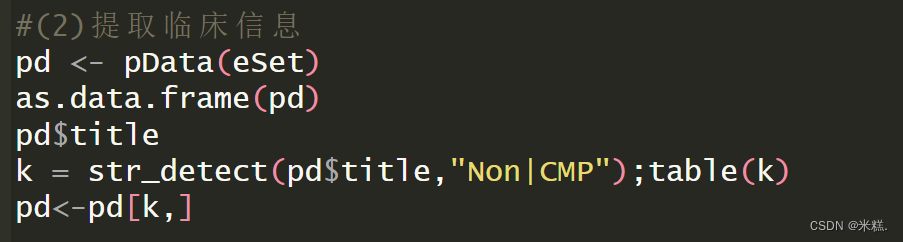
使用pData(eSet)函數就能提取到臨床信息了,這個eSet就是我們最開始時用來接收下載的數據的那個對象,函數的返回結果是一個數據框,也就是說臨床信息我們使用一個名為pd的數據框保存。pd長這樣

可以看到所謂的臨床信息包含了title,這一列有特發性擴張型心肌病左心室,缺血性左心室,非衰竭左心室這三種情況,后面還有什么日期等等一系列的臨床信息,我想要研究擴張型心肌病和健康人基因表達的差異,那我就根據title這一列中的字符串篩選出這兩種情況,也就是把缺血性左心室剔除,我們發現特發性擴張型心肌病左心室這種情況有一個特有的字符串叫做Non,而非衰竭左心室也就是健康樣本有一個字符串叫做CMP,運行代碼
k = str_detect(pd$title,"Non|CMP");table(k)將會得到一個邏輯向量,如果title這一列(也就是一個向量)的元素中有Non或者CMP,就返回TRUE,否則返回False,再運行代碼pd擴張型心肌病和健康樣本這兩種情況的行了。
接下來要讓exp的列名和pd的行名完全一致,至于為什么后面再解釋。

identical(rownames(pd),colnames(exp))用于查看pd(只有擴張型心肌病和健康樣本這兩種情況的數據框)的行名和exp(表達矩陣)的列名 是不是相同,pd的行名和exp的列名都是不同樣本名,他們是有可能順序相同的,如果不同我們執行if語句里面的內容把他們變成順序相同,intersect函數用于找出兩個向量中的公共元素,返回值是一個向量,該向量中的元素均為傳給intersect函數的兩個參數比如x和y的公共元素,無需考慮下標是不是相同,只要一個元素在x和y兩個向量中同時存在,那么這個元素就是intersect返回結果的其中一個元素。我們用s來接收這個intersect函數的結果,這個結果就是一些樣本名,然后根據這些樣本名提取出exp的列和pd的行并覆蓋掉exp和pd,這樣就能保證exp的列和pd的行是完全一樣的。
預處理還需要把探針轉換成對應的基因名,這個代碼一般是不需要改動的
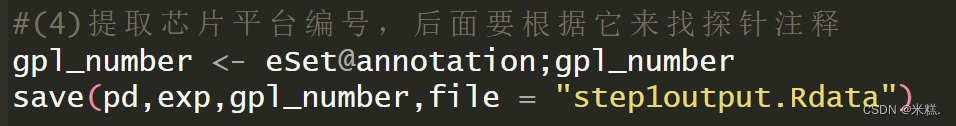
我們看到這里提取eSet中的元素用的是@,這個符號的功能和數據框提取某一列用的那個$是一個道理。一般eSet第一次提取用的是@,后續可能是@也可能是$至于怎么提取可以通過點左上角環境中的這個eSet變量,點開是這樣的,根據這里的結構決定是用@還是$提取元素

為了防止代碼太長不便于維護,我們把目前所得到的,后續有用的變量存起來
save(pd,exp,gpl_number,file = "step1output.Rdata"),其實目前有用的就是存有臨床信息且經過處理之后只剩下擴張型心肌病和健康兩種情況的那個數據框pd,以及表達矩陣exp,還有剛才提取到的芯片平臺編號,這個編號用于后續把探針轉換成基因名。
# Group(實驗分組)和ids(探針注釋)
rm(list = ls())
load(file = "step1output.Rdata")
# 1.Group----
library(stringr)
# 標準流程代碼是二分組,多分組數據的分析后面另講# 使用字符串處理的函數獲取分組k = str_detect(pd$title,"Non");table(k)
# 需要把Group轉換成因子,并設置參考水平,指定levels,對照組在前,處理組在后
Group = ifelse(k,"healthy","DCM")
table(Group)
#這個數據框是為了檢查一下分組是不是成功
check_df<-data.frame(pd$title,Group)
#把Group這個向量轉換成因子類型,前面是對照組
Group<-factor(Group,levels = c("healthy","DCM"))
#2.探針注釋的獲取-----------------
#捷徑
library(tinyarray)
find_anno(gpl_number) #輔助寫出找注釋的代碼
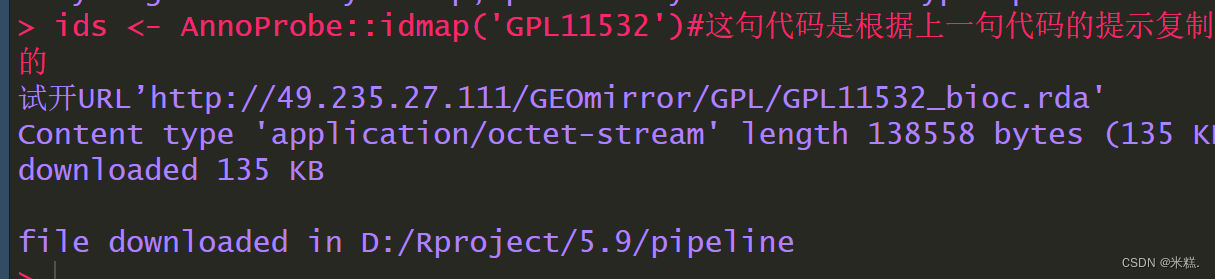
ids <- AnnoProbe::idmap('GPL11532')#這句代碼是根據上一句代碼的提示復制來的
#如果能打出代碼就不需要再管其他方法。
#如果使用復制下來的AnnoProbe::idmap('xxx')代碼發現報錯了,請注意嘗試不同的type參數
#如果不知道type能取什么,可以使用?idmap來查看
#如果顯示no annotation avliable in Bioconductor and AnnoProbe則要去GEO網頁上看GPL表格里找啦。#捷徑里面包含了全部的R包、一部分表格、一部分自主注釋
#方法1 BioconductorR包(最常用,已全部收入find_anno里面,不用看啦)
if(F){gpl_number #看看編號是多少#http://www.bio-info-trainee.com/1399.html #在這里搜索,找到對應的R包library(hgu133plus2.db)ls("package:hgu133plus2.db") #列出R包里都有啥ids <- toTable(hgu133plus2SYMBOL) #把R包里的注釋表格變成數據框
}
# 方法2 讀取GPL網頁的表格文件,按列取子集
##https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GPL570
# 方法3 官網下載注釋文件并讀取
# 方法4 自主注釋,了解一下
#https://mp.weixin.qq.com/s/mrtjpN8yDKUdCSvSUuUwcA
save(exp,Group,ids,file = "step2output.Rdata")
重新開一個R腳本清空原來環境中的變量以防止以前運行的內容對本次要運行的代碼造成影響
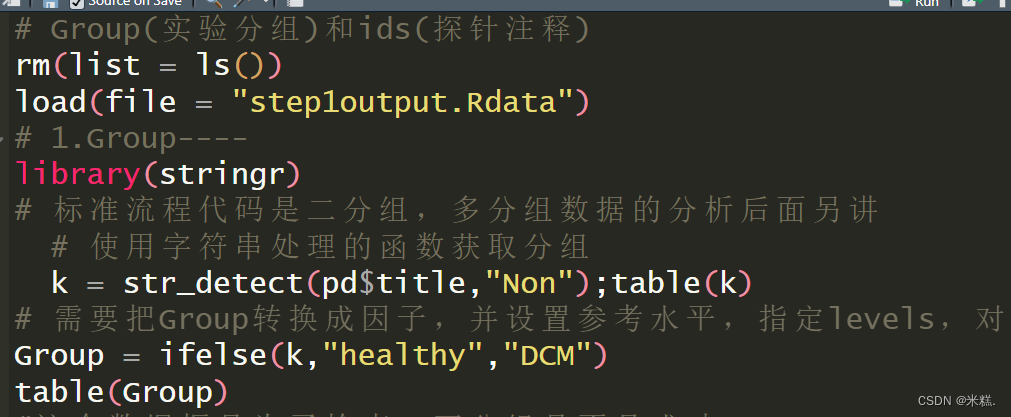
使用r包stringr中的函數str_detect根據title這一列中是否含有Non這個字符串進行篩選,得到一個邏輯向量k,結果如圖

這個結果顯示有136個健康樣本,82個擴張型心肌病樣本。
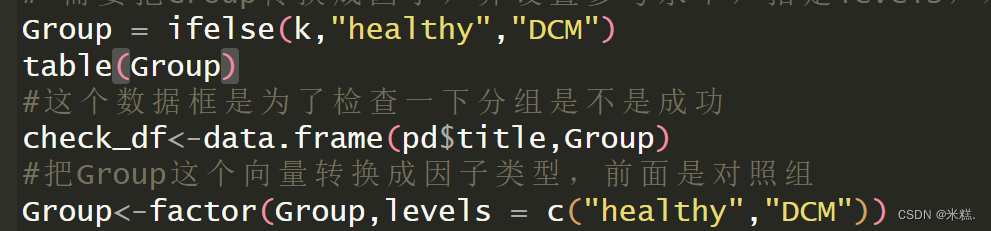
創建一個名為Group的向量,并根據前面的把這個向量的內容改成healthy和DCM(DCM是擴張型心肌病的簡稱),結果應該是有原本136個TRUE的位置元素被改成了healthy,82個FALSE對應位置的元素被改成DCM。
然后緊接著創建了一個名為check_df的數據框把title這一列和我們剛才創建的Group這個向量放在了一起,顧名思義這個數據框就是為了讓我們直觀地檢查一下是不是正確的分組了。我在這里展示部分check_df的結果

從結果來看我們確實正確的分組了。知道Group這個變量是對的之后,我們就把他轉換成因子類型,這是因為后續分析所用到的函數的參數要求。因子這種類型特別適用于處理一些有很多重復元素的向量。

上面這三句代碼是在進行探針轉換為基因名的工作,find_anno是R包tinyarray中的一個函數,他需要的參數是我們前面得到的編號,扔進去之后會得到一個提示性的結果,我這里是這樣

根據這個代碼運行結果的提示寫了第三句代碼











】矩陣)


方案助力建設智慧校園)


PyLammps 教程)
![[JAVASE] 類和對象(二)](http://pic.xiahunao.cn/[JAVASE] 類和對象(二))
pixabay免費可商用的圖片、視頻、插畫、矢量圖、音樂)