拿到表達矩陣之后,如何使用ggplot2繪圖系統繪制箱線圖?
目錄
預備知識
繪制箱線圖示例
預備知識
1.pivot_longer函數
pivot_longer 是tidyr包中的一個函數,用于將數據框(data frame)從寬格式轉換為長格式。在寬格式數據中,每個觀測值可能是多列的行,而長格式數據則將這些多列轉換為兩列:一個包含觀測值的變量,另一個包含相應的值。
以下是pivot_longer的基本語法和主要參數:
pivot_longer(data, cols, names_to = NULL, values_to = "value")- data:要轉換的數據框。
- cols:要轉換為長格式的列,可以是一個單獨的列名,也可以是一個包含多個列名的向量。可以使用c()函數來指定多個列。
- names_to:新變量名稱的列名。可以是一個字符串,或者一個包含字符串的向量,用于指定轉換后的列名。
- values_to:新數值列的列名,默認為"value"。
2.starts_with()函數
starts_with()是dplyr包中的函數,用于在數據操作中篩選出以特定字符串開始的列。
以下是starts_with()函數的基本語法和參數:
starts_with(x, prefix, ignore.case = FALSE)- x:一個字符向量,即你要檢查的列。
- prefix:一個字符串,表示你想要匹配的前綴。
- ignore.case:一個邏輯值,指定是否忽略大小寫進行匹配。默認為FALSE,即默認情況下是區分大小寫的。
通常只需要傳入參數prefix也就是想要匹配的前綴即可。這個函數不能單獨使用,而是作為某些函數的參數來使用,單獨使用會報這樣的一個錯誤
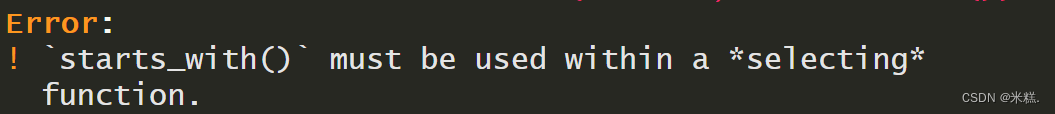
說是這個函數必須配合一種selecting函數來使用,比如select函數,pivot_longer函數等。舉個例子
select(df,starts_with("name"))表示篩選出df數據框中有前綴是name的列,結果仍然是數據框。
繪制箱線圖示例
首先運行代碼
set.seed(10086)
exp<-matrix(round(rnorm(18),2),ncol = 6)
rownames(exp)<-paste0("gene",1:3)
colnames(exp)<-paste0("test",1:6)
exp[,1:3]<-exp[,1:3]+1生成一個三行六列的矩陣,設置種子數是為了讓產生的隨機數可重復。然后對這個矩陣的行名和列名進行了修改。exp[,1:3]
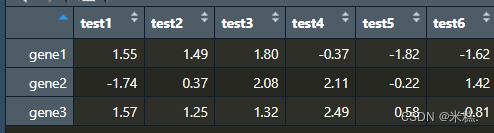
其中行表示不同的基因,列表示不同的樣本,對于這樣一個矩陣,是不能扔到ggplot函數里面畫箱線圖的,因為首先函數要求的數據格式是數據框,我們得到的是矩陣,那是不是直接把這個表達矩陣轉換成數據框就能扔進ggplot函數里面繪制箱線圖了?我們先來看一下使用ggplot函數繪制箱線圖的語法格式:
ggplot(數據框)+ geom_boxplot(aes(列名,列名))
基本的要求是數據框中兩列的列名,第一個列名用作自變量(x),第二個列名用作因變量(y),再來看我們現在的矩陣,如果直接把他轉換成數據框,其實內容沒有變化,但是我們要怎么給geom_boxplot函數傳參?傳哪兩列都不合適,正確處理方式如下:
1.使用t函數轉置表達矩陣,并使用as.data.frame函數把這個矩陣轉換成數據框。
2.使用rownames_to_column把原來的行名變成第一列,因為我們要根據基因名這一列和后面搞出來的表達量那列繪圖。
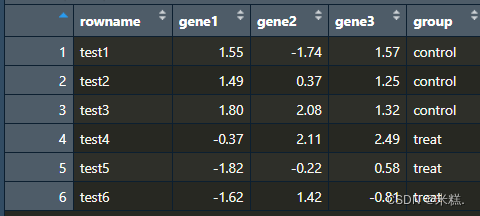
3.使用mutate函數增加一列,通常命名為group,這一列的內容通常為健康和患病。這是為了在箱線圖中顯示圖例。經過這一步之后我們的數據框如圖,假設這個數據框名為dat,也可以不使用mutate函數,直接dat$group
4.使用pivot_longer函數把這個數據框“寬變長”,得到我們需要的數據框。所謂的寬變長是指:原來每個test都有其對應的gene1,2,3,現在我想要把帶gene的這些列都合成一列,假設這一列叫做gene,同時要能夠讓test1,gene1唯一的確定一個基因表達量,就只能把test1,test2等等這些帶test的每一個都變成三個,也就是下圖中這樣子

在這張表中我們仍然可以通過test1和gene1唯一的確定一個表達量,由于行數的變化,表達量也要用新的一列來存起來,比如表格里面的count就是我們新創建的一列。完成這一系列操作只需要使用一個函數,他就是 pivot_longer,這樣傳參
pdat<-dat%>%pivot_longer(cols = starts_with("gene"),names_to = "gene",values_to = "count")管道符用于把前面的參數傳到后面去,默認是傳到后面函數的第一個參數位置上去。其中start_with("gene")是用來篩選出以gene開頭的列,如果要篩選的基因沒有這樣gene1,gene2的,而是三個基因的名字,我們可以直接給cols參數寫2:4,names_to=“gene”表示指定的這三列合成的新的那一列名字叫做gene,同時原來由test1和gene1這樣的兩個維度唯一確定的那些基因的表達量由于格式的變化也要創建新的一列來存起來,這一列我們給他起名為count。
5.使用ggplot函數繪制箱線圖。
示例代碼:
library(ggplot2)
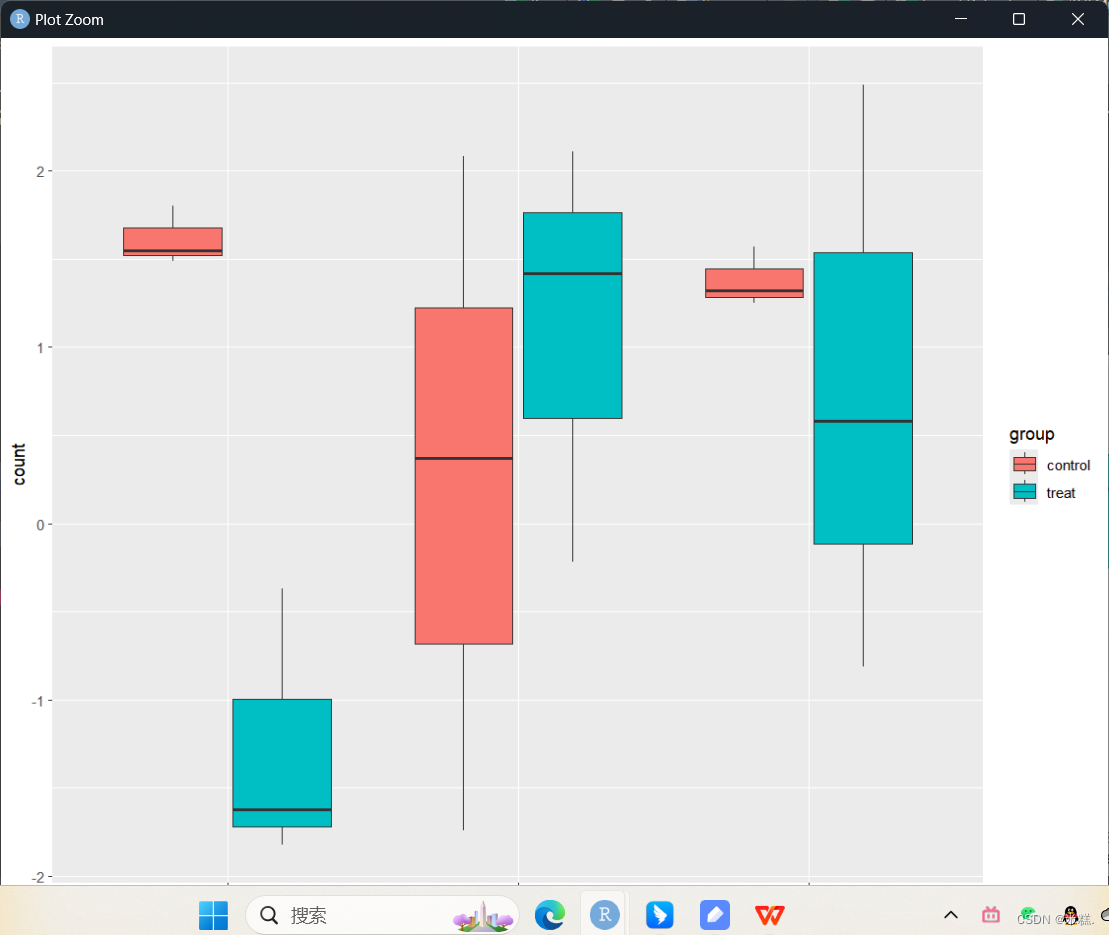
ggplot(pdat,aes(gene,count))+geom_boxplot(aes(fill=group))表示橫坐標是gene這一列,縱坐標是count這一列,填充顏色由group這一列的取值來決定。
運行代碼之后就繪制了這樣一張圖
最后送上完整源代碼,可以直接運行
set.seed(10086)
exp<-matrix(round(rnorm(18),2),ncol = 6)
rownames(exp)<-paste0("gene",1:3)
colnames(exp)<-paste0("test",1:6)
exp[,1:3]<-exp[,1:3]+1
library(tidyr)
library(tibble)
library(dplyr)
dat<-t(exp)%>%as.data.frame()%>%rownames_to_column()%>%mutate(group=rep(c("control","treat"),each=3))
pdat<-dat%>%pivot_longer(cols = starts_with("gene"),names_to = "gene",values_to = "count")
library(ggplot2)
ggplot(pdat,aes(gene,count))+geom_boxplot(aes(fill=group))


)


)




![[初學rust] 05_ rust struct](http://pic.xiahunao.cn/[初學rust] 05_ rust struct)
)






