目錄
- 原理
- 保守組檢測
- 非保守組檢測
- 二次重復測試
- 自適應二次重復測試
- 四種測試方法的核心代碼
- 保守群組測試
- 非保守群組測試
- 二次重復測試與自適應二次重復測試
- 測試代碼
- 參考文獻
原理
假設該病在人群中的患病率(先驗概率)為p,我們想用群體檢驗法檢驗N個獨立樣本組大小n,即樣本數量。
單個試驗的診斷方法的FNR用fN表示,FPR可忽略不計。
用G表示一組樣本,G=0表示G中的所有樣本均未感染。相反,G=1意味著G是一個陽性組;即,它至少有一個感染標本。通過觀察不同方法的分組試驗結果,我們將一個組分為感染組和未感染組,分別用G?=1和G?=0表示。
保守組檢測

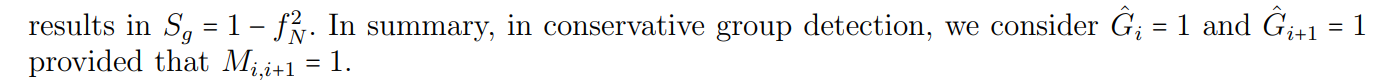
非保守組檢測

步驟如下:
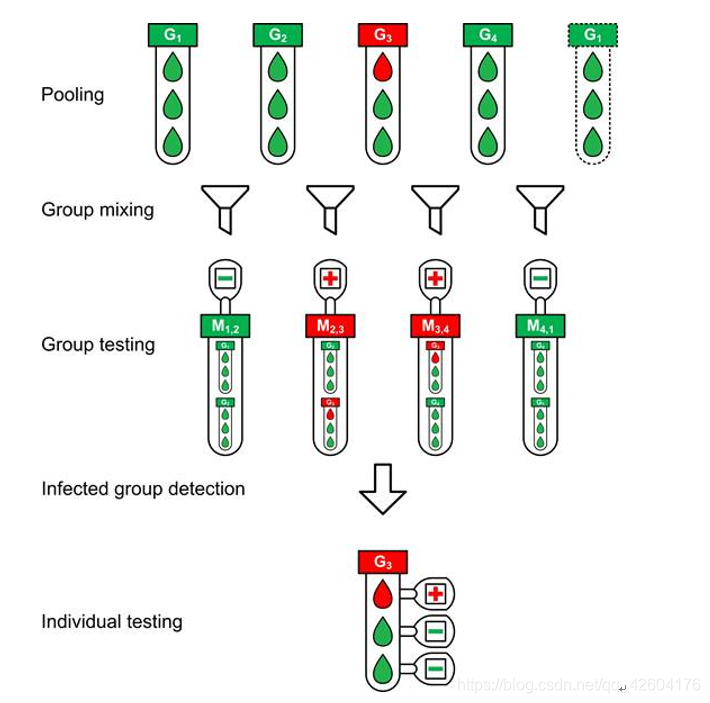
每個Group由三個sample人的血液組成。
這樣能保證每個Group能被測試兩次(也代表著每個sample人的血液能夠被測試兩次),fN表示單個試驗的FNR(false negative rate),這樣就能保證Sg(群體水平敏感度)= 1 - fN^2;
總的來說,Gi、Gi+1為陽性,則Mi,i+1也為陽性。
這樣,圖1中的M2,3和M3,4都是正的,很可能是因為G3=1。盡管在G2=1和G4=1的情況下也會出現這些陽性結果,但缺少M1,2=1和M4,1=1使得G2和G4都不太可能等于1。在這種方法中,例如,如果G2為真陽性,M1,2=0為假陰性結果,我們將忽略G2=1,從而導致靈敏度降低。
在非保守組檢測中有以下規則:
二次重復測試
簡單來說就是對分組之后檢測的所有組都檢測兩遍,只要有一次為陽則說明這個組中有陽性。

自適應二次重復測試
第二次測試將第一次測試中為陰性的進行測試。

四種測試方法的核心代碼
【0】保守群組測試 【1】非保守群組測試 【2】二次重復測試 【3】二次自適應重復測試
保守群組測試
//非保守測試
//輸入:splited_group 、 分組混合血液 、 假陰率 fnr
//輸出:檢測出來的陽性樣本數、總檢測次數
int No_Conservative_Group_Detection(vector<vector<int>>& splited_group,vector<int>& result, float fnr, vector<int>& Positive_Group, vector<int>& PositiveSamples)
{//將Gi、Gi+1混合到一起為Mi//如果相鄰兩個M(Mj,Mj+1)檢測結果是陽性,則認定是Gj+1是陽性。//如果只有單獨的Mj檢測結果是陽性,則將Gj與Gj+1都檢測一遍。int All_Test_Times = 0;//【step1】將G血液結果兩兩混合到M中vector<int> Mixed_Group_Result;for (int i = 0; i < result.size() - 1; i++){if (result[i] == 1 || result[i + 1] == 1)Mixed_Group_Result.emplace_back(1);else Mixed_Group_Result.emplace_back(0);}//第一個樣本與最后一個樣本混合if(result[0] == 1 || result[result.size()-1] == 1) Mixed_Group_Result.emplace_back(1);else Mixed_Group_Result.emplace_back(0);//【step2】對M結果進行檢測,需要考慮到假陰性影響,得到陽性G序號vector<int> Blured_Mixed_Group_Result;Test_Synthetic_Blood(Mixed_Group_Result, Blured_Mixed_Group_Result, fnr);All_Test_Times += Mixed_Group_Result.size();//尋找檢測結果為陽性的Mfor (int j = 0; j < Blured_Mixed_Group_Result.size() - 1; j++){//如果是兩個連在一起的為陽性,取交集的Gif (Blured_Mixed_Group_Result[j] == 1 && Blured_Mixed_Group_Result[j + 1] == 1){Positive_Group.emplace_back(j+1);}else if(Blured_Mixed_Group_Result[j] == 1 && Blured_Mixed_Group_Result[j + 1] == 0){//如果Gj已經被送入結果,那么只需要再送入Gj+1if (!Positive_Group.empty() && Positive_Group.back() == j){Positive_Group.emplace_back(j + 1);}//否則,將兩個都送入else{Positive_Group.emplace_back(j);Positive_Group.emplace_back(j + 1);}}}cout << "非保守測試可能為陽性的Group個數" << Positive_Group.size() << endl;//對每個可能陽性的Group進行核酸檢測for (int i = 0; i < Positive_Group.size(); i++){int pos_index = Positive_Group[i];//對可能陽性Group中每個樣本都進行檢測,要考慮到假陰性的影響,得到陽性的樣本的序號。vector<int> Blured_Splited_GroupResult;Test_Synthetic_Blood(splited_group[pos_index], Blured_Splited_GroupResult, fnr);//對檢測結果進行記錄,記錄下在原序列中的坐標for (int j = 0; j < Blured_Splited_GroupResult.size(); j++){if (Blured_Splited_GroupResult[j] == 1){PositiveSamples.emplace_back(pos_index* splited_group[0].size() + j);} }}All_Test_Times += Positive_Group.size() * splited_group[0].size();return All_Test_Times;}
非保守群組測試
//保守測試
//輸入:splited_group 、 分組混合血液 、 假陰率 fnr
//輸出:檢測出來的陽性樣本數、總檢測次數
int Conservative_Group_Detection(vector<vector<int>>& splited_group, vector<int>& result, float fnr, vector<int>& Positive_Group, vector<int>& PositiveSamples)
{//將Gi、Gi+1混合到一起為Mi//如果M檢測結果是陽性,則認定是Gj,Gj+1是陽性。int All_Test_Times = 0;//【step1】將G血液結果兩兩混合到M中vector<int> Mixed_Group_Result;for (int i = 0; i < result.size() - 1; i++){if (result[i] == 1 || result[i + 1] == 1)Mixed_Group_Result.emplace_back(1);elseMixed_Group_Result.emplace_back(0);}//第一個樣本與最后一個樣本混合if (result[0] == 1 || result[result.size() - 1] == 1) Mixed_Group_Result.emplace_back(1);else Mixed_Group_Result.emplace_back(0);//【step2】對M結果進行檢測,需要考慮到假陰性影響,得到陽性G序號vector<int> Blured_Mixed_Group_Result;Test_Synthetic_Blood(Mixed_Group_Result, Blured_Mixed_Group_Result, fnr);All_Test_Times += Mixed_Group_Result.size();//尋找檢測結果為陽性的Mfor (int j = 0; j < Blured_Mixed_Group_Result.size() - 1; j++){if (Blured_Mixed_Group_Result[j] == 1){if (!Positive_Group.empty() && Positive_Group.back() == j){Positive_Group.emplace_back(j + 1);}else{Positive_Group.emplace_back(j);Positive_Group.emplace_back(j + 1);}}}//對每個可能陽性的Group進行核酸檢測cout << "保守測試可能為陽性的Group個數"<< Positive_Group.size() <<endl;for (int i = 0; i < Positive_Group.size(); i++){int pos_index = Positive_Group[i];//對可能陽性Group中每個樣本都進行檢測,要考慮到假陰性的影響,得到陽性的樣本的序號。vector<int> Blured_Splited_GroupResult;Test_Synthetic_Blood(splited_group[pos_index], Blured_Splited_GroupResult, fnr);//對檢測結果進行記錄,記錄下在原序列中的坐標for (int j = 0; j < Blured_Splited_GroupResult.size(); j++){if (Blured_Splited_GroupResult[j] == 1){PositiveSamples.emplace_back(pos_index * splited_group[0].size() + j);}}}All_Test_Times += Positive_Group.size() * splited_group[0].size();return All_Test_Times;}
二次重復測試與自適應二次重復測試
/*
* 重復二次測量
* 輸入:result實際感染情況,blured_result第一次檢測情況,rep_blured_result第二次檢測情況
* 輸出:
*/
void Replicate_Test(vector<int>& result, vector<int>& blured_result, vector<int>& rep_blured_result, float fnr)
{rep_blured_result.clear();srand((unsigned)time(NULL));int fnr_100 = fnr * 100;for (int i = 0; i < result.size(); i++) //第二次全覆蓋測試{//對每個陽性樣本進行混淆處理if (result[i] == 1){int num = rand() % 100;// r % 概率檢測為陰性if (num <= fnr_100){rep_blured_result.emplace_back(0);}//1-fnr%概率檢測為陽性else{rep_blured_result.emplace_back(1);}}//由于covid-19不存在假陽性,這里對陰性樣本不做處理else{rep_blured_result.emplace_back(0);}}for (int i = 0; i < result.size(); i++) { //將第一第二次的檢測結果合并if (blured_result[i] == 1) {rep_blured_result[i] = 1;}}return;
}
/*
* 自適應重復二次測量,對象為第一次測量中為陰性的組
* 輸入:result實際感染情況,blured_result第一次檢測情況,rep_blured_result第二次檢測情況
* 輸出:二次測量的測量數
*/
int Adt_Replicate_Test(vector<int>& result, vector<int>& blured_result, vector<int>& rep_blured_result, float fnr) {rep_blured_result.clear();srand((unsigned)time(NULL));int fnr_100 = fnr * 100;int Test_Times = 0;for (int i = 0; i < blured_result.size(); i++){//對第一次檢測結果再進行挑選檢測if (blured_result[i] == 1) //第一次檢測為陽性,跳過檢測{rep_blured_result.emplace_back(1);}else //第一次檢測為陰性,再次檢測{Test_Times++;if (result[i] == 1) {int num = rand() % 100;// r % 概率檢測為陰性if (num <= fnr_100){rep_blured_result.emplace_back(0);}//1-fnr%概率檢測為陽性else{rep_blured_result.emplace_back(1);}}else rep_blured_result.emplace_back(0);}}return Test_Times;
}/*
* 重復二次測量
* 輸入:splited_group分組,result實際感染情況,fnr假陰率,Positive_Group二次測量后的陽性組,PositiveSamples二次測量后的陽性樣本,Mode方法模式,0為重復二次測量,1為自適應重復二次測量
* 輸出:
*/
int Replicate_TestDetection(vector<vector<int>>& splited_group, vector<int>& result, float fnr, vector<int>& Positive_Group, vector<int>& PositiveSamples,int Mode)
{//將Gi、Gi+1混合到一起為Mi//如果M檢測結果是陽性,則認定是Gj,Gj+1是陽性。int All_Test_Times = 0;//【step1】將G血液結果兩兩混合到M中vector<int> Mixed_Group_Result;for (int i = 0; i < result.size() - 1; i++){if (result[i] == 1 || result[i + 1] == 1)Mixed_Group_Result.emplace_back(1);elseMixed_Group_Result.emplace_back(0);}//第一個樣本與最后一個樣本混合if (result[0] == 1 || result[result.size() - 1] == 1) Mixed_Group_Result.emplace_back(1);else Mixed_Group_Result.emplace_back(0);vector<int> Blured_Mixed_Group_Result;//第一次檢測Test_Synthetic_Blood(Mixed_Group_Result, Blured_Mixed_Group_Result, fnr);All_Test_Times += Mixed_Group_Result.size();vector<int> Sec_Blured_Mixed_Group_Result;if (Mode == 0){//第二次檢測,采用重復二次測量Replicate_Test(result, Blured_Mixed_Group_Result, Sec_Blured_Mixed_Group_Result, fnr);All_Test_Times += Blured_Mixed_Group_Result.size();}else{//第二次檢測,采用自適應重復二次測量,對象為第一次測量中為陰性的組int Sec_Test_Times = Adt_Replicate_Test(result, Blured_Mixed_Group_Result, Sec_Blured_Mixed_Group_Result, fnr);All_Test_Times += Sec_Test_Times;}//記錄二次結果后的陽性組for (int i = 0; i < Sec_Blured_Mixed_Group_Result.size(); i++){if (Sec_Blured_Mixed_Group_Result[i] == 1) Positive_Group.emplace_back(i);}//對每個可能陽性的Group進行核酸檢測cout << "二次重復測試可能為陽性的Group個數" << Positive_Group.size() << endl;for (int i = 0; i < Positive_Group.size(); i++){int pos_index = Positive_Group[i];//對可能陽性Group中每個樣本都進行檢測,要考慮到假陰性的影響,得到陽性的樣本的序號。vector<int> Blured_Splited_GroupResult;Test_Synthetic_Blood(splited_group[pos_index], Blured_Splited_GroupResult, fnr);//對檢測結果進行記錄,記錄下在原序列中的坐標for (int j = 0; j < Blured_Splited_GroupResult.size(); j++){if (Blured_Splited_GroupResult[j] == 1){PositiveSamples.emplace_back(pos_index * splited_group[0].size() + j);}}}All_Test_Times += Positive_Group.size() * splited_group[0].size();return All_Test_Times;
}
測試代碼
//檢測總人數int sample_nums = 175000;//流行率設定double p = 0.04;//分組設定int sec_group_sample_nums =2;//假陰率設定float FNR = 0.2;int positive_sample_nums = p * sample_nums;if (sample_nums <= 0){cout <<"error1" <<endl;return 0;}sec_group_sample_nums = pow(2, i + 1);vector<int> Group(sample_nums, 0);//混合血液后的真實結果vector<int> Result;//受到假陰性影響后的混合血液測試結果vector<int> Blured_Result;vector<vector<int>> Splited_Group;//【1】初始化樣本序列Initial_Group(Group, positive_sample_nums);//Pirnt_Group(Group);cout << endl;//【2】Pooling操作Split_Group(Group, Splited_Group, sec_group_sample_nums);//Pirnt_Split_Group(Splited_Group);cout << endl;//【3】混合血液Synthetic_Blood(Splited_Group, Result);vector<int> Positive_Group;vector<int> PositiveSamples;int All_Test_Times = 0;int Methods = 3;//【4】使用不同的測試方法switch (Methods){//【0】保守群組測試case 0:{All_Test_Times = Conservative_Group_Detection(Splited_Group, Result, FNR, Positive_Group, PositiveSamples);cout << "重復性測試總檢測次數" << All_Test_Times << endl;if (positive_sample_nums != 0)cout << "檢測出的陽性樣本個數為" << PositiveSamples.size() << "準確率" << PositiveSamples.size() * 1.0f / positive_sample_nums << endl;elsecout << "error2" << endl;}break;//【1】非保守群組測試case 1:{All_Test_Times = No_Conservative_Group_Detection(Splited_Group, Result, FNR, Positive_Group, PositiveSamples);cout << "非重復性測試總檢測次數" << All_Test_Times << endl;if (positive_sample_nums != 0)cout << "檢測出的陽性樣本個數為" << PositiveSamples.size() << "準確率" << PositiveSamples.size() * 1.0f / positive_sample_nums << endl;elsecout << "error2" << endl;}break;//【2】二次重復測試case 2:{All_Test_Times = Replicate_TestDetection(Splited_Group, Result, FNR, Positive_Group, PositiveSamples, 0);cout << "重復性測試總檢測次數" << All_Test_Times << endl;cout << "陽性樣本組數" << Positive_Group.size() << endl;if (positive_sample_nums != 0)cout << "檢測出的陽性樣本個數為" << PositiveSamples.size() << "準確率" << PositiveSamples.size() * 1.0f / positive_sample_nums << endl;elsecout << "error2" << endl;}break;//【3】二次自適應重復測試case 3:{All_Test_Times = Replicate_TestDetection(Splited_Group, Result, FNR, Positive_Group, PositiveSamples, 1);cout << "重復性測試總檢測次數" << All_Test_Times << endl;if (positive_sample_nums != 0)cout << "檢測出的陽性樣本個數為" << PositiveSamples.size() << "準確率" << PositiveSamples.size() * 1.0f / positive_sample_nums << endl;elsecout << "error2" << endl;}break;default:break;}//根據仿真得出一個測試實際社會成本(即找出最后為陰性的組數/總組數*每組多少人*每個人的閑置成本)int Neg_Groups = Splited_Group.size() - Positive_Group.size();double Neg_rate = Neg_Groups * 1.0f / Splited_Group.size();cout << "Neg_rate " << Neg_rate << endl;cout << "總樣本個數 " << Splited_Group.size() << endl;
參考文獻
https://www.medrxiv.org/content/10.1101/2020.07.31.20154070v1

)




![[轉載]Struts2 獲得Session和Request](http://pic.xiahunao.cn/[轉載]Struts2 獲得Session和Request)












![int main(int argc,char* argv[])講解](http://pic.xiahunao.cn/int main(int argc,char* argv[])講解)