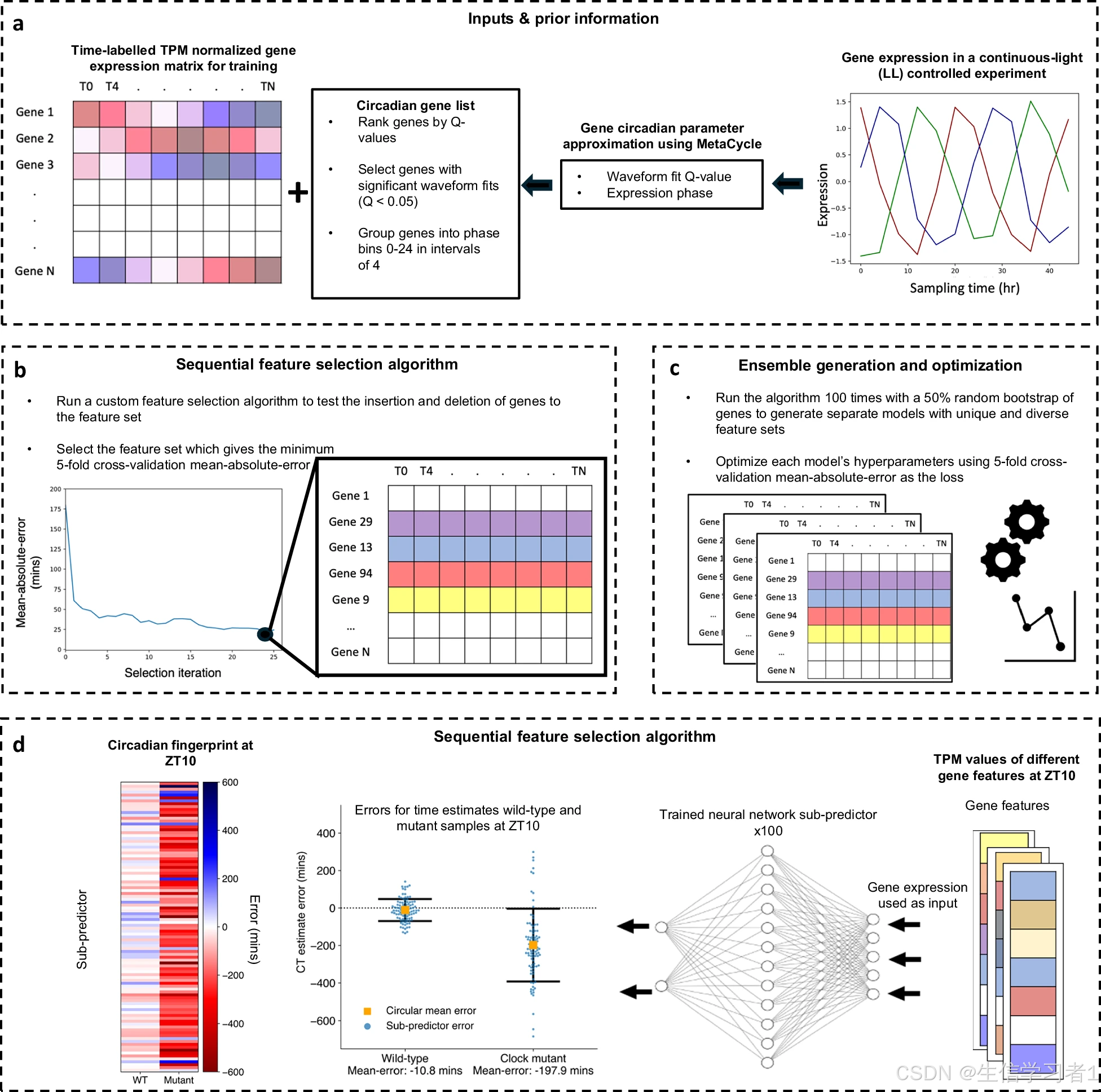
數據輸入及前期信息:ChronoGauge 需要一個基因表達矩陣,其中包括來自多個時間進程 RNA-測序實驗的觀測數據,用于訓練,并且需要有關每個基因在連續光照(LL)條件下經過光暗(LD)周期調整后的時程采集過程中表達的晝夜節律性和相位的先驗信息(這些信息由 MetaCycle 確定)。具有非顯著晝夜節律性(meta2d Q 值小于 0.05)的基因會被過濾掉。基因被分為 6 個相位區間,范圍在 0 至 24 小時之間,間隔為 4 小時。b 特征選擇:使用一種包含正向和反向步驟的自定義順序特征選擇(SFS)算法,通過迭代構建具有不同波形相位的基因特征集,以五折交叉驗證(CV)的平均絕對誤差(MAE)作為成本。從每次運行中選擇具有最小 MAE 的特征集。c 集成生成與優化:使用隨機 50% 的基因抽樣對 SFS 算法進行 100 次運行,從而得到獨特的特征集,這些特征集用于調整和訓練 100 個不同的子預測神經網絡(NN)。單時間點 CT 估計:將訓練好的子預測器應用于單時間點測試樣本,從而得出 100 種不同的晝夜時間(CT)估計值,這些估計值通過圓形平均聚合方法合并為一個總體估計值。可以對不同樣本中的 CT 估計值的誤差進行比較,例如在野生型植物和時鐘突變體之間進行比較28。在每個樣本內,晝夜特征指紋代表了針對不同基因特征的子預測器所產生誤差的差異。蜂群圖的屬性包括:中央橙色框 = 平均值,羽狀線 = 標準差,藍色點 = 每個子預測器的誤差。

文章目錄
- 介紹
- 代碼
- 參考
介紹
植物的生物鐘對其生存和適應能力起著重要作用。然而,要從轉錄組層面理解生物鐘的功能及其對環境的響應,需要通過高分辨率的時間進程實驗來進行測定。生成這些數據集是一項勞動密集型且成本高昂的任務,在大多數情況下是在嚴格控制的實驗室條件下進行的。為了克服這些障礙,我們開發了 ChronoGauge:一個集成模型,能夠利用少量指示時間的基因在單個時間點的轉錄組樣本中的表達情況,可靠地估計擬南芥植物的內源生物鐘時間。ChronoGauge 可以在未見過的擬南芥整體 RNA-測序和微陣列樣本中高精度地預測植物的生物鐘時間,并且還可以進一步應用于在非模式物種(包括田間樣本)的樣本中進行非隨機預測。最后,我們展示了如何將 ChronoGauge 應用于生成關于生物鐘轉錄組對特定基因型或環境條件反應的假設。
The circadian clock of plants contributes to their survival and fitness. However, understanding clock function at the transcriptome level and its response to the environment requires assaying across high resolution time-course experiments. Generating these datasets is labour-intensive, costly and, in most cases, performed under tightly controlled laboratory conditions. To overcome these barriers, we have developed ChronoGauge: an ensemble model that can reliably estimate the endogenous circadian time of Arabidopsis plants using the expression of a handful of time-indicating genes within a single time-pointed transcriptomic sample. ChronoGauge can predict a plant’s circadian time with high accuracy across unseen Arabidopsis bulk RNA-seq and microarray samples, and can be further applied to make non-random predictions across samples in non-model species, including field samples. Finally, we demonstrate how ChronoGauge can be applied to generate hypotheses regarding the response of the circadian transcriptome to specific genotypes or environmental conditions.
代碼
https://github.com/ConnorReynoldsUK/ChronoGauge
“ChronoGauge”是一種類似裝袋的組合模型,用于從轉錄組樣本(例如 RNA 測序、微陣列)中估算晝夜時間(CT)。該模型是專門為植物轉錄組數據的分析而開發的。
ChronoGauge 是基于擬南芥 RNA-測序數據進行訓練的,并已在多種應用場景中得到應用,包括:
在 RNA-seq 數據中檢驗與生物鐘相關的假設(例如,對照樣本與接受實驗干擾的樣本之間的差異)
微陣列樣本
使用基因同源物的非模式物種
通過多次使用不同的種子值運行自定義順序特征選擇(SFS)包裝器來生成子預測器的集合。每次 SFS 運行都會輸出一個獨特的特征集,可用于訓練子預測器。這些子預測器可以應用于包含這些特征集的測試數據集,并將它們的預測結果匯總以提供一個穩健的 CT 估計,盡管存在技術變異/批次效應。
- https://github.com/ConnorReynoldsUK/ChronoGauge_Xspecies
此存儲庫是“ChronoGauge”的擴展,其中包含了專門針對我們使用擬南芥表達數據訓練而成的組合模型在非模式物種中的應用(用于晝夜時間(CT)預測)的相關工作。
在此我們先說明一下,將 ChronoGauge 應用于不同物種(在僅針對擬南芥進行訓練后)所得到的預測結果,預計不會像在相同物種上進行訓練和測試那樣具有高度的可靠性。
我們還注意到,對于非模型物種,有如下要求:需采集至少兩個時間點的樣本,且這兩個時間點之間的間隔應約為 12 小時。
參考
- https://github.com/ConnorReynoldsUK/ChronoGauge_Xspecies
- https://github.com/ConnorReynoldsUK/ChronoGauge
- Machine learning models highlight environmental and genetic factors associated with the Arabidopsis circadian clock





:Vue3響應式編程中this綁定機制與解決方案)

的使用——以NDVI去水體為例)




flutter命令行工具)



)


